Содержание
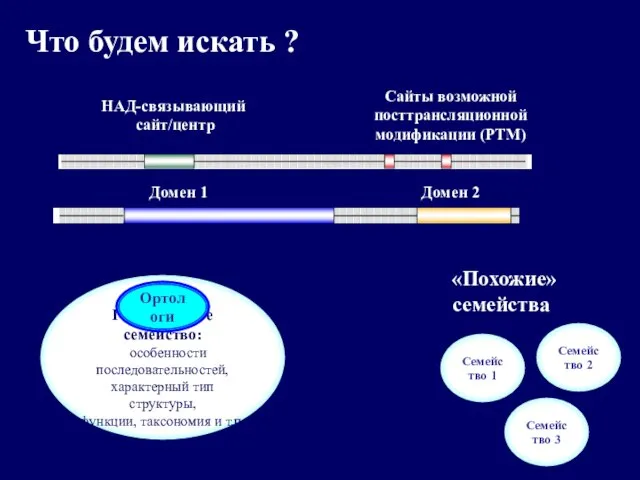
- 2. Что будем искать ? НАД-связывающий сайт/центр Сайты возможной посттрансляционной модификации (РТМ) Домен 1 Домен 2 Гомологичное
- 3. Паттерн (pattern) – Позиционно специфическая матрица весов (PSSM) – Профиль–PSSM – Профиль–HМM - Подпись (signature) –
- 4. Домен – единица эволюции, структуры и функции белков. Домен – компактная, относительно независимо сворачивающаяся структура, относительно
- 5. Мотив ? Мотив в аминокислотной последовательности - набор консервативных остатков, важных для функции белка и расположенных
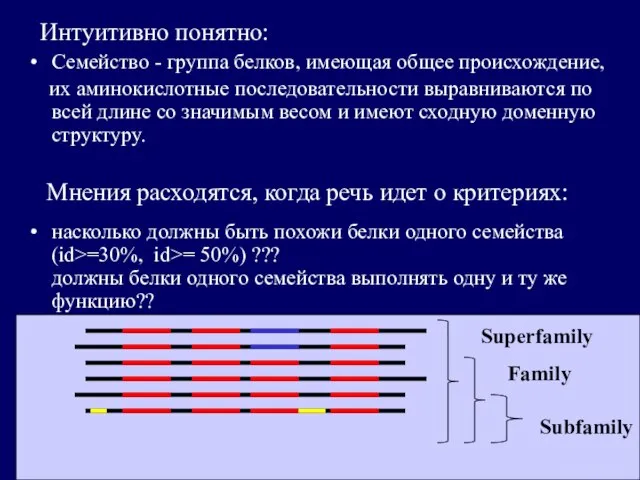
- 6. Интуитивно понятно: Семейство - группа белков, имеющая общее происхождение, их аминокислотные последовательности выравниваются по всей длине
- 7. No comments
- 8. Паттерн (pattern) – Позиционно специфическая матрица весов (PSSM) – Профиль–PSSM – Профиль–HМM - Подпись (signature) –
- 10. Банки белковых семейств и доменов, производные от банков аминокислотных последовательностей Коллекции мотивов Коллекции доменов PROSITE ,
- 11. PROSITE - биологически значимые сайты, паттерны и профили Выравнивание хорошо изучен-ного семейства Функционально важные остатки 4-5
- 12. PROSITE - биологически значимые сайты, паттерны и профили
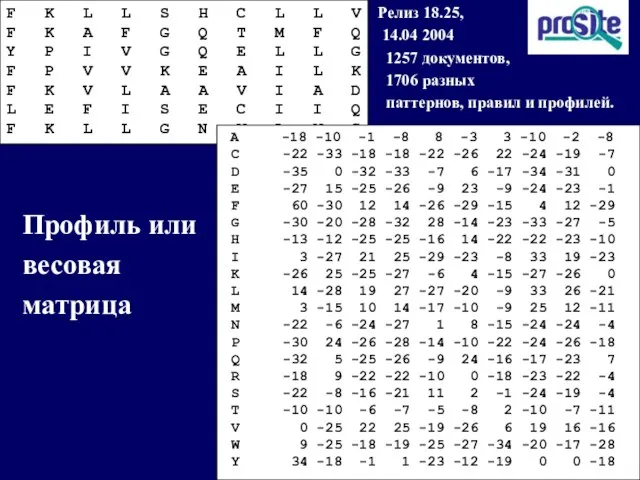
- 13. PROSITE Релиз 18.25, 14.04 2004 1257 документов, 1706 разных паттернов, правил и профилей. Профиль или весовая
- 14. Pfam http://www.sanger.ac.uk/Software/Pfam/index.shtml Большая коллекция множественных выравниваний, доменов, семейств и профилей-HMM для них. Состоит из 2-х частей:
- 15. Pfam Множественное выравнивание (ClustalX) некоторого семейства или кластера. Экспертиза и корректировка выравнивания-затравки. Построение профиля-НММ для затравки.
- 16. ProDom http://www.toulouse.inra.fr/prodom.html Рассматриваются все последовательности в SWISS-Prot+TrEMBL. Автоматическое выделение доменов (программа DOMAINER: сначала локальное попарное выравнивание
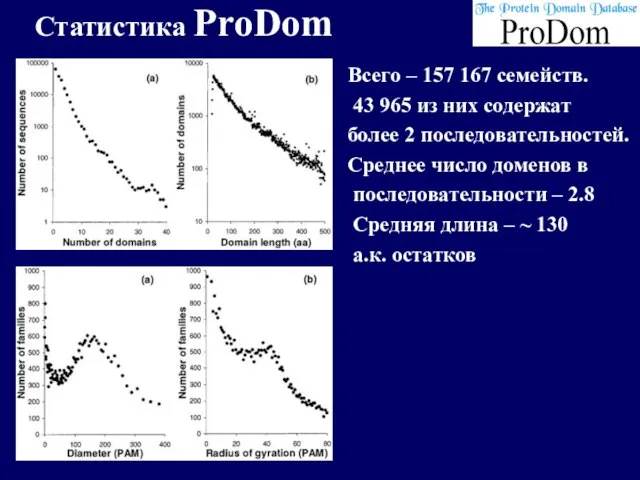
- 17. Статистика ProDom Всего – 157 167 семейств. 43 965 из них содержат более 2 последовательностей. Среднее
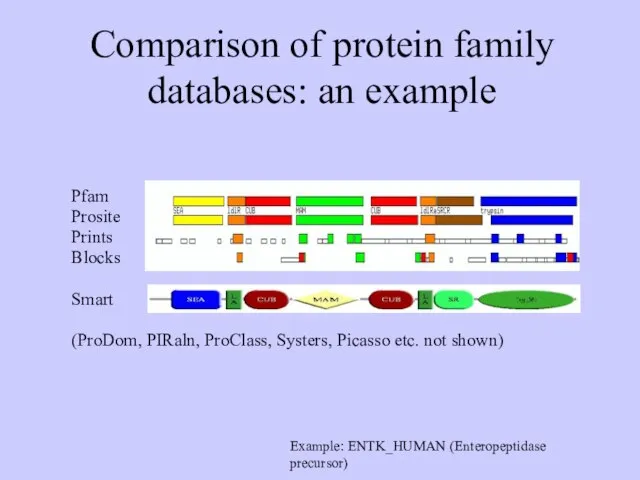
- 18. Pfam Prosite Prints Blocks Smart (ProDom, PIRaln, ProClass, Systers, Picasso etc. not shown) Example: ENTK_HUMAN (Enteropeptidase
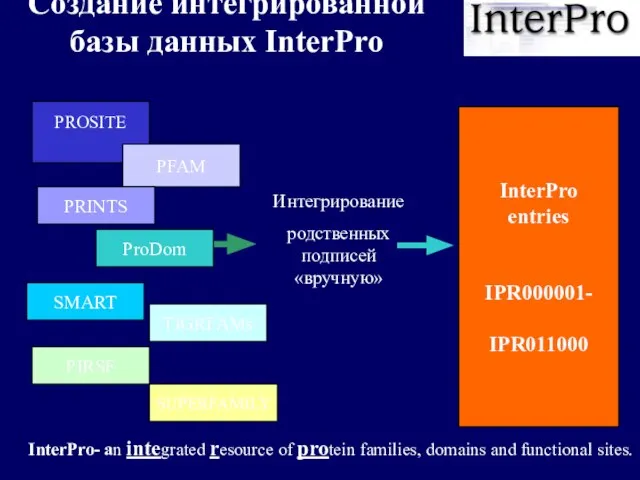
- 19. Создание интегрированной базы данных InterPro PROSITE PFAM PRINTS InterPro entries IPR000001- IPR011000 Интегрирование родственных подписей «вручную»
- 20. Entry types in InterPro Family - group of evolutionarily related proteins, that share one or more
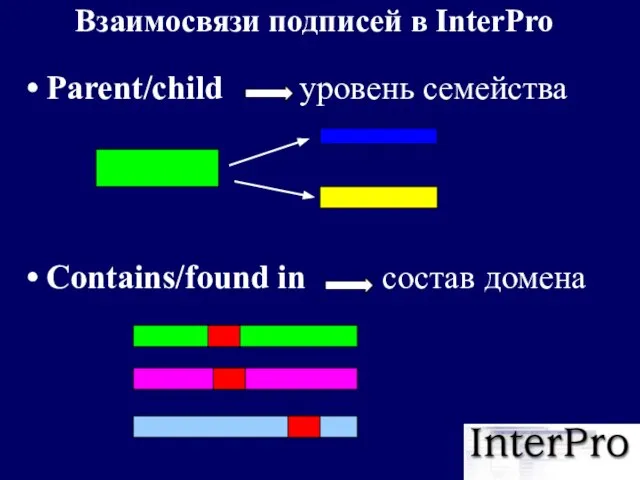
- 21. Взаимосвязи подписей в InterPro Parent/child уровень семейства Contains/found in состав домена
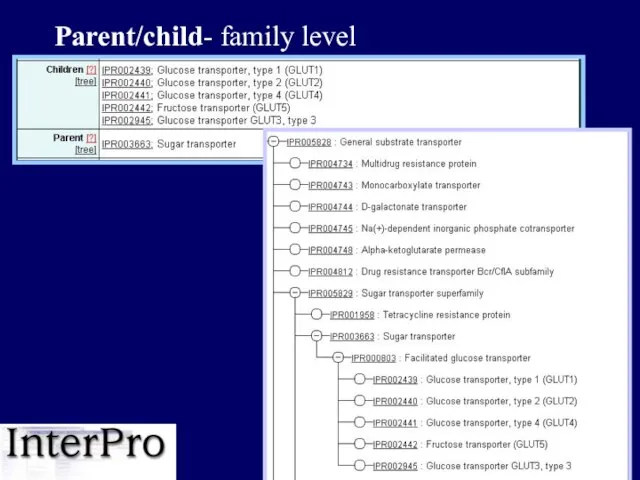
- 22. Parent/child- family level
- 23. Contains/found in
- 24. PROTOMAP http://www.protomap.cs.huji.ac.il Automatic classification of all SWISS-PROT proteins into groups of related proteins (also including TrEMBL
- 26. Скачать презентацию















 Тема: «Спортивные игры зимой»
Тема: «Спортивные игры зимой» Список детей на получение кроватей и полотенец
Список детей на получение кроватей и полотенец Тепловые двигатели. Паровая турбина.
Тепловые двигатели. Паровая турбина. Система образования в Англии
Система образования в Англии Геометрические паркеты
Геометрические паркеты Подходы к системному целеполаганию. Лекция 2
Подходы к системному целеполаганию. Лекция 2 Конституция РФ
Конституция РФ Проект "Задачи на смеси и сплавы"
Проект "Задачи на смеси и сплавы" Раздел #. Дизайн интерьера
Раздел #. Дизайн интерьера Основы научных исследований
Основы научных исследований 000834aa-f5be4922
000834aa-f5be4922 Погружение
Погружение Невербальное общение
Невербальное общение Однородность керамзита
Однородность керамзита Презентация на тему Длина и меры ее измерения
Презентация на тему Длина и меры ее измерения Презентация на тему Экономика как наука
Презентация на тему Экономика как наука Кофемашина Yamaguchi CoffeeMann
Кофемашина Yamaguchi CoffeeMann Танковая викторина
Танковая викторина Петр Петрович Семёнов-Тян-Шанский
Петр Петрович Семёнов-Тян-Шанский Александр
Александр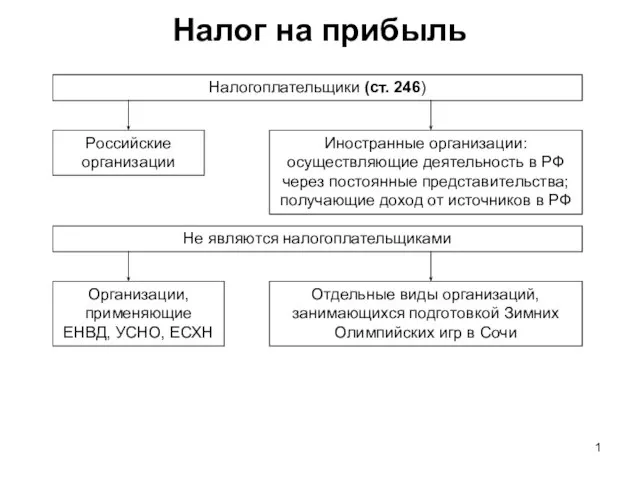 Налог на прибыль
Налог на прибыль SPORTS in Our Life
SPORTS in Our Life  Аудирование —цель и средство обучения
Аудирование —цель и средство обучения Отношения мужчины и женщины. Женская психология
Отношения мужчины и женщины. Женская психология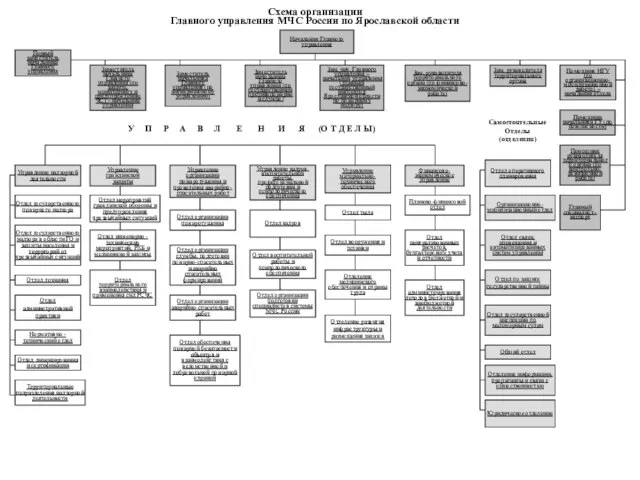 Схема организации Главного управления МЧС России по Ярославской области
Схема организации Главного управления МЧС России по Ярославской области Презентация на тему Афанасий Афанасьевич Фет (4 класс)
Презентация на тему Афанасий Афанасьевич Фет (4 класс) муниципальное образовательное учреждениеОбходская основная общеобразовательная школаУренского муниципального районаНижегор
муниципальное образовательное учреждениеОбходская основная общеобразовательная школаУренского муниципального районаНижегор куликовская битва
куликовская битва