МИНИСТЕРСТВО ОБРАЗОВАНИЯ И НАУКИ УКРАИНЫ Национальный технический университет Украины Факультет биотехнологии и биотехники;
Содержание
- 2. Фенилкетонурия Одним из наиболее распространенных наследственных заболеваний является фенилкетонурия В Украине на 8300 новорожденных один ребенок
- 3. Фенилаланингидроксилаза катализирует реакцию преобразования фенилаланин + О2 + тетрагидробиоптерин ↓ тирозин + вода + окисленный биоптерин
- 4. Цель работы Провести ДНК-анализ мутаций и полиморфизма гена ФАГ и оптимизировать технологию проведения полимеразной цепной реакции
- 5. Для достижения цели были поставлены основные задачи исследования: ДНК-анализ мутаций R408W, R158Q, Y414C, IVS10nt546, IVS12nt1 гена
- 6. Объект и предмет исследования Объект исследования - ДНК человека полученная из лейкоцитов периферической крови Предмет исследования
- 7. Методы: Выделение и очистка ДНК; амплификация in vitro последовательностей гена ФАГ методом полимеразной цепной реакции; рестрикционный
- 8. Этапы ДНК-анализа: Выделение геномной ДНК из образцов венозной крови методом фенольной экстракции; Амплификация in vitro фрагмента
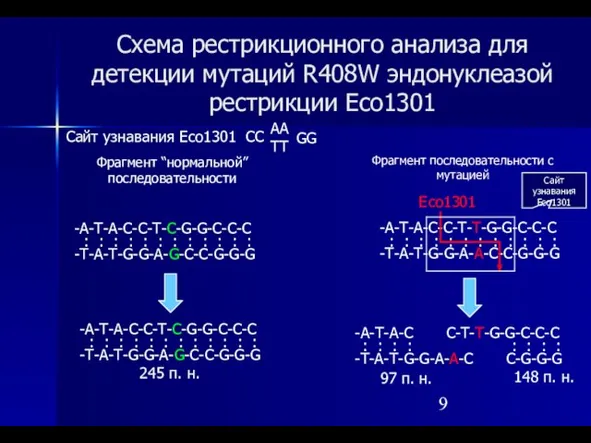
- 9. Схема рестрикционного анализа для детекции мутаций R408W эндонуклеазой рестрикции Eco1301 Сайт узнавания Eco1301 CC -A-T-A-C-C-T-C-G-G-C-C-C -T-A-T-G-G-A-G-C-C-G-G-G
- 10. Анализ мутации R408W. 1 2 3 4 1, 2 – R408W/норма, 3 – норма/норма, 4 –
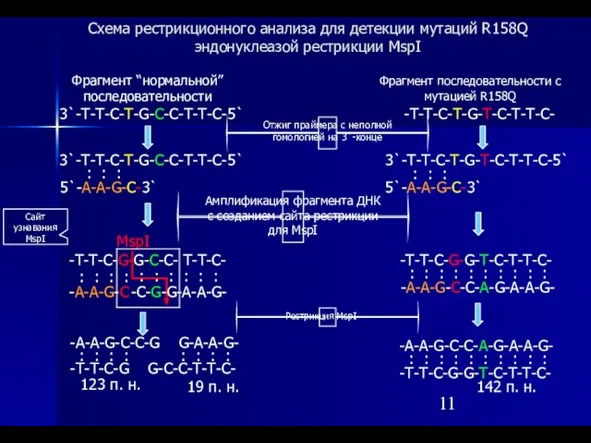
- 11. : : : : 19 п. н. Фрагмент “нормальной” последовательности Фрагмент последовательности с мутацией R158Q -A-A-G-C
- 12. Анализ мутации R158Q. 123 п.н. 119 п.н. 1 2 3 4 1, 2 - R158Q/норма; 3,
- 13. На первом этапе мы рассматривали четыре паралельно протикающие реакции: ДНК+Pr →ДНК.Pr Образование комплекса ДНК.Pr ДНК.Pr →ДНК+Pr
- 14. Концентрации реагентов описываются системой диф. уравнений: Решив данную систему мы получили:
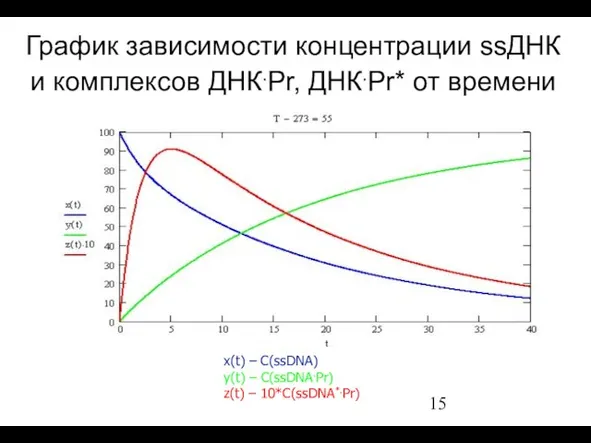
- 15. График зависимости концентрации ssДНК и комплексов ДНК.Pr, ДНК.Pr* от времени x(t) – C(ssDNA) y(t) – C(ssDNA.Pr)
- 16. На втором этапе мы рассматривали шесть паралельно протикающие реакции: ДНК+Pr →ДНК.Pr Образование комплекса ДНК.Pr ДНК.Pr →ДНК+Pr
- 17. Данные процессы описываются системой диф. уравнений:
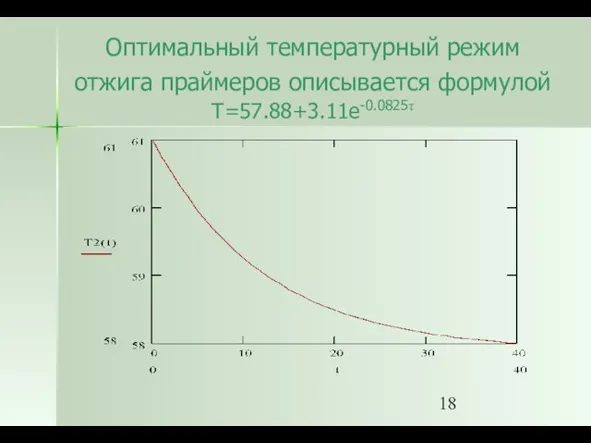
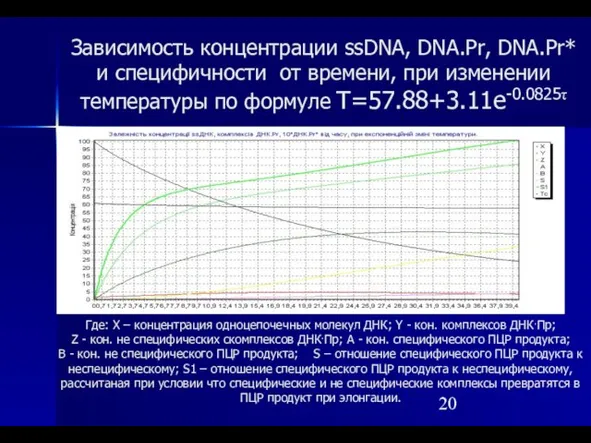
- 18. Оптимальный температурный режим отжига праймеров описывается формулой T=57.88+3.11e-0.0825τ
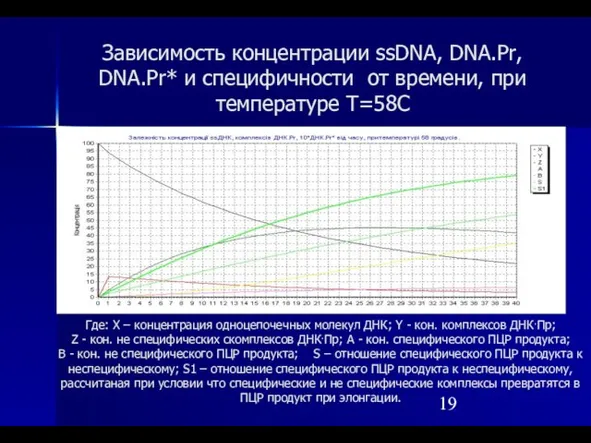
- 19. Зависимость концентрации ssDNA, DNA.Pr, DNA.Pr* и специфичности от времени, при температуре T=58С Где: X – концентрация
- 20. Зависимость концентрации ssDNA, DNA.Pr, DNA.Pr* и специфичности от времени, при изменении температуры по формуле T=57.88+3.11e-0.0825τ Где:
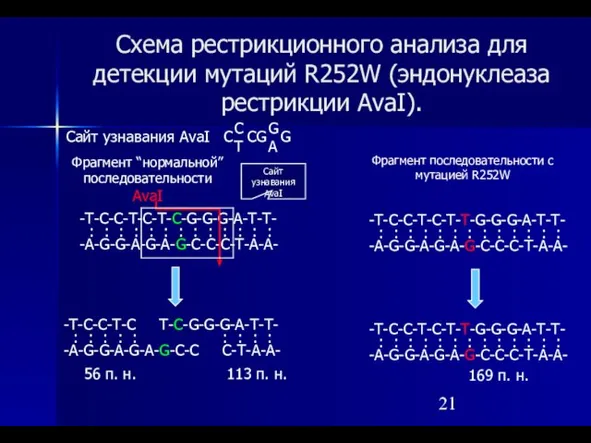
- 21. Схема рестрикционного анализа для детекции мутаций R252W (эндонуклеаза рестрикции AvaI). -T-C-C-T-C-T-C-G-G-G-A-T-T- -A-G-G-A-G-A-G-C-C-C-T-A-A- : : : :
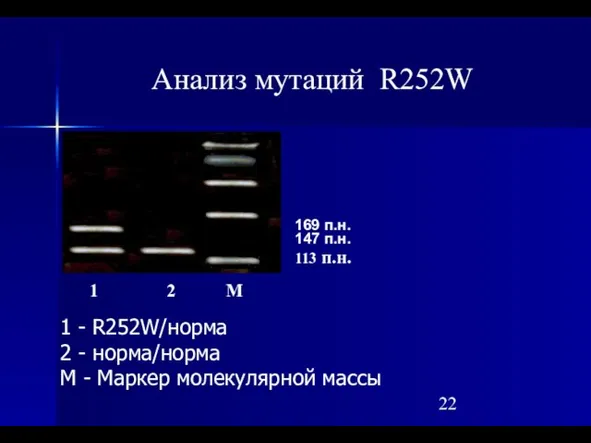
- 22. Анализ мутаций R252W 1 - R252W/норма 2 - норма/норма М - Маркер молекулярной массы
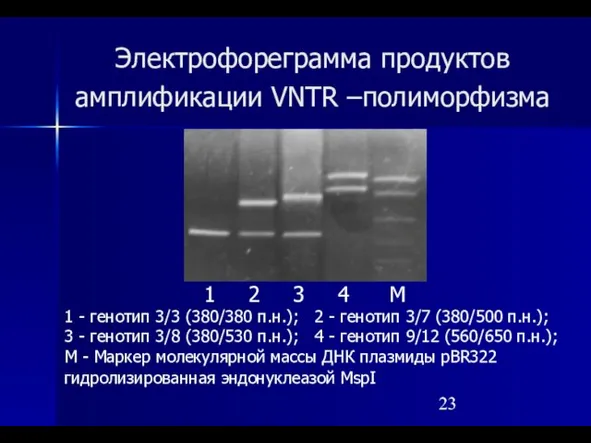
- 23. Электрофореграмма продуктов амплификации VNTR –полиморфизма 1 2 3 4 М 1 - генотип 3/3 (380/380 п.н.);
- 24. Выводы 1. Проведен ДНК-анализ мутаций R408W, IVS12nt1, R158Q, Y414C, IVS10nt546 гена ФАГ в 68 образцах. Двадцать
- 26. Скачать презентацию














 Висит за окошком Кулёк ледяной, Он полон капели И пахнет весной
Висит за окошком Кулёк ледяной, Он полон капели И пахнет весной Средства регулирования движением поездов
Средства регулирования движением поездов Афины при Перикле
Афины при Перикле Электронная таблица EXCEL
Электронная таблица EXCEL Архетипы
Архетипы УНИДРУА: международный институт по унификации частного права
УНИДРУА: международный институт по унификации частного права Органы чувств
Органы чувств  «Этимологический анализ прозвищ жителей деревень Завражье, Мешково и поселка Судоверфь Судоверфского сельского поселения»
«Этимологический анализ прозвищ жителей деревень Завражье, Мешково и поселка Судоверфь Судоверфского сельского поселения» Новогодние открытки
Новогодние открытки Робототехника. Проект
Робототехника. Проект Балансирующий рынок
Балансирующий рынок Теорема Пифагора
Теорема Пифагора Культура XX века
Культура XX века Деревня Юрьевец
Деревня Юрьевец Wine. The benefits of wine with moderate consumption
Wine. The benefits of wine with moderate consumption Биопсихосоциодуховный подход к профилактике профессионального выгорания психологов
Биопсихосоциодуховный подход к профилактике профессионального выгорания психологов Воздействие высокоскоростных магистралей на окружающую среду
Воздействие высокоскоростных магистралей на окружающую среду Презентация экскурсии в животноводческий комплекс «ИП Каюмов»
Презентация экскурсии в животноводческий комплекс «ИП Каюмов» 07_0___163
07_0___163 каждую неделю около 600 000 слушателей включаются на частоту 100,5 FM каждый день Радио BEST FM слушают около 300 000 человек каждые 15 минут на в
каждую неделю около 600 000 слушателей включаются на частоту 100,5 FM каждый день Радио BEST FM слушают около 300 000 человек каждые 15 минут на в «Белые» в Гражданской войне
«Белые» в Гражданской войне Внедрение здоровьесберегающих технологий
Внедрение здоровьесберегающих технологий Русское искусство 2 пол.19 века
Русское искусство 2 пол.19 века Всегда ли правы мы- родители?
Всегда ли правы мы- родители? Итоги работы отрасли в 2010 году и задачи на 2011 год Глава департамента Елена Борисовна Чеснокова
Итоги работы отрасли в 2010 году и задачи на 2011 год Глава департамента Елена Борисовна Чеснокова Семейные традиции семьи Митрофановых
Семейные традиции семьи Митрофановых HADI – это экспериментальный научный метод исследования, переложенный на бизнес
HADI – это экспериментальный научный метод исследования, переложенный на бизнес ПРОЕКТ КНИЖКА-САМОДЕЛКА «Волшебница Зима» 4 класс
ПРОЕКТ КНИЖКА-САМОДЕЛКА «Волшебница Зима» 4 класс