Содержание
- 2. Полногеномный поиск ассоциаций Подходы в исследованиях GWAS Случай - контроль Анализ количественных фенотипических признаков Схема, при
- 3. Алгоритм mtSet программного комплекса Limix
- 4. Цель работы Цель данной работы заключается в том, чтобы разработать алгоритм, который, возможно, с небольшой потерей
- 5. Разработанный алгоритм Алгоритм формирования групп фенотипических признаков Алгоритм быстрого полногеномного поиска ассоциаций Интеграционный модуль Алгоритм полногеномного
- 6. Алгоритм формирования групп фенотипических признаков Количественные фенотипические признаки Решение проблемы неполноты данных Замена средним Метод линейной
- 7. Алгоритм формирования групп фенотипических признаков часть 1 0.95 0.80 0.70
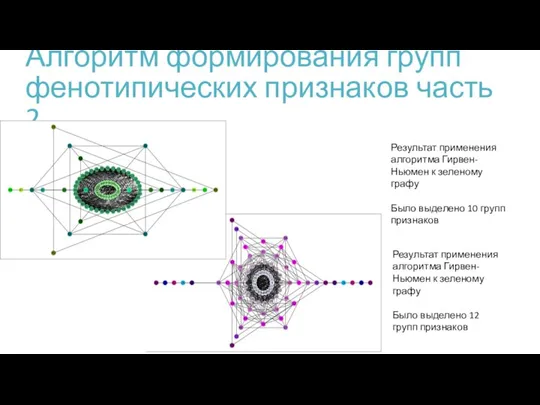
- 8. Алгоритм формирования групп фенотипических признаков часть 2 Результат применения алгоритма Гирвен-Ньюмен к зеленому графу Было выделено
- 9. Алгоритм быстрого полногеномного поиска ассоциаций часть 1 Показатель вклада i-го SNP в данную группу фенотипических признаков
- 10. Алгоритм быстрого полногеномного поиска ассоциаций часть 2 Алгоритм иерархической кластеризации методом ближайшего соседа Получили набор отрезков
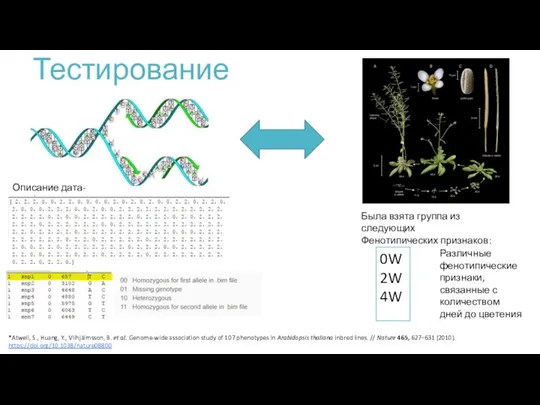
- 11. Тестирование Описание дата-сета* Была взята группа из следующих Фенотипических признаков: 0W 2W 4W Различные фенотипические признаки,
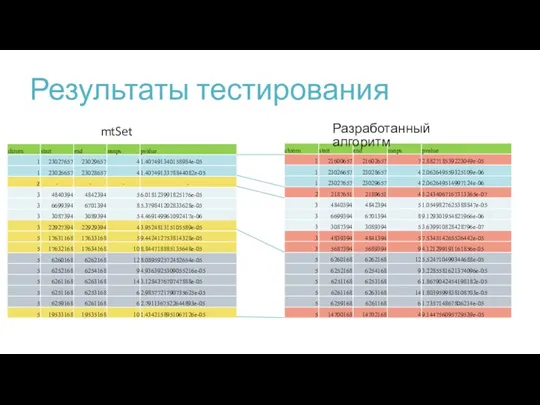
- 12. Результаты тестирования mtSet Разработанный алгоритм
- 13. Дальнейшие шаги Скорректировать кластеризацию координат аллелей для получения более точных отрезков генома для их дальнейшей подачи
- 14. Список литературы Casale, F., Rakitsch, B., Lippert, C. et al. Efficient set tests for the genetic
- 16. Скачать презентацию










 Изобретения, подсмотренные у природы
Изобретения, подсмотренные у природы Презентация на тему Взаимодействие генов
Презентация на тему Взаимодействие генов  Механика сердечного пульса
Механика сердечного пульса Обмен веществ в организме
Обмен веществ в организме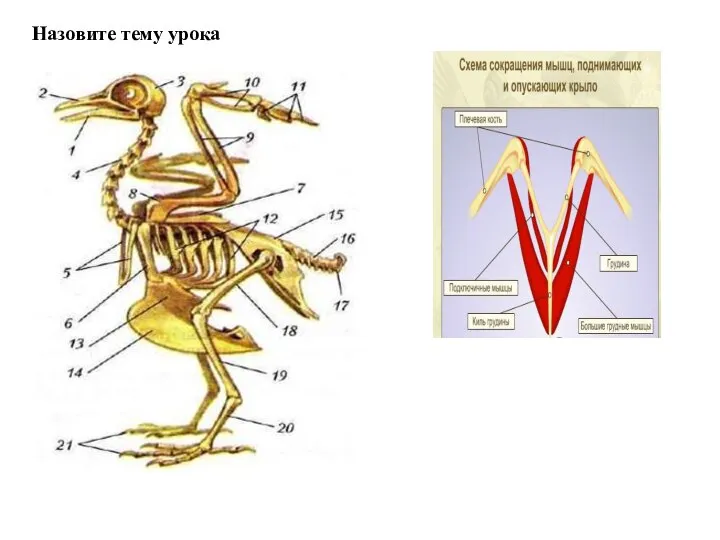 Опорно-двигательная система птиц
Опорно-двигательная система птиц ЭКСПРЕСС диагностика логопеда
ЭКСПРЕСС диагностика логопеда Имена в биологии. 10 класс
Имена в биологии. 10 класс Презентация на тему Таємниці царства Грибів
Презентация на тему Таємниці царства Грибів  Презентация на тему Рост и развитие животных
Презентация на тему Рост и развитие животных  Бесполое размножение
Бесполое размножение урок 16 поведение
урок 16 поведение Почва и ее значение
Почва и ее значение Класс Насекомые (морфология, анатомия, физиология)
Класс Насекомые (морфология, анатомия, физиология) Зимующие птицы
Зимующие птицы Презентация на тему Папоротники
Презентация на тему Папоротники  Рептилии в террариуме. Содержание ящериц
Рептилии в террариуме. Содержание ящериц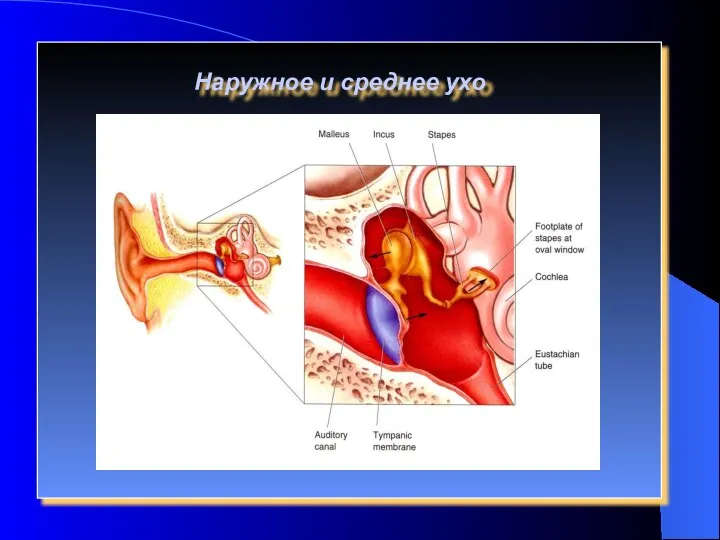 Слуховой анализатор
Слуховой анализатор Презентация на тему Мутации (11 класс)
Презентация на тему Мутации (11 класс)  Презентация на тему Животные Австралии
Презентация на тему Животные Австралии  Презентация на тему Животные маленькие и большие (3 класс)
Презентация на тему Животные маленькие и большие (3 класс)  Терек табигать торлелеге. Патшалыклар
Терек табигать торлелеге. Патшалыклар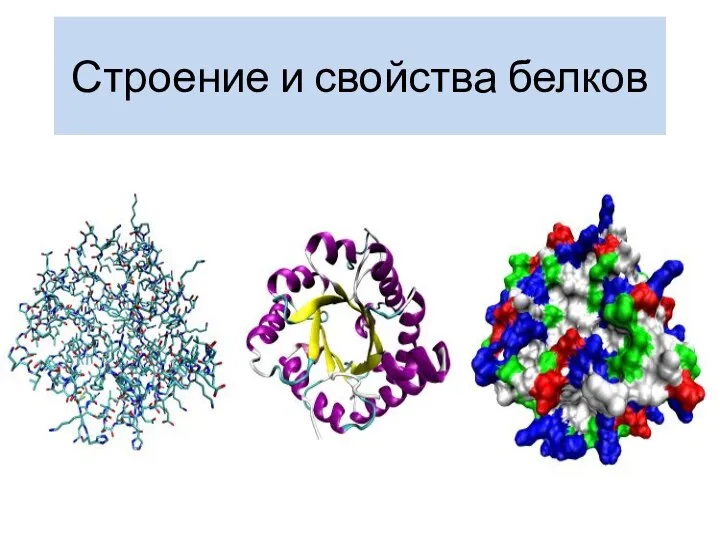 Строение и свойства белков
Строение и свойства белков Веселая биология. Загадки
Веселая биология. Загадки История энтомологии
История энтомологии Физиология и биохимия микроорганизмов
Физиология и биохимия микроорганизмов Урок биологии на тему Соцветия
Урок биологии на тему Соцветия Нуклеиновые кислоты. Мономер-нуклеотид
Нуклеиновые кислоты. Мономер-нуклеотид Презентация на тему Грибы Саратовской области
Презентация на тему Грибы Саратовской области