Содержание
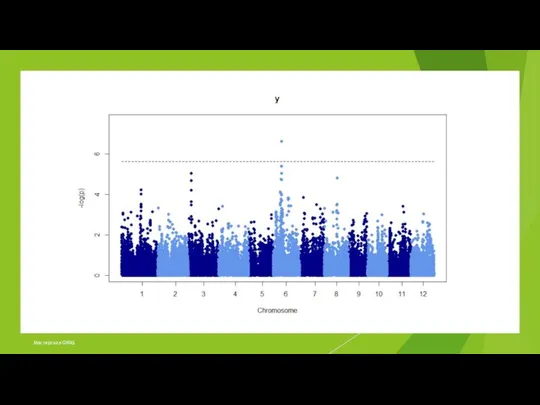
- 2. Исследование полногеномной ассоциации Ищем SNP… связанный с фенотипом. Цель: Объяснять Понимание Механизмы Терапия Предсказывать Вмешательство Профилактика
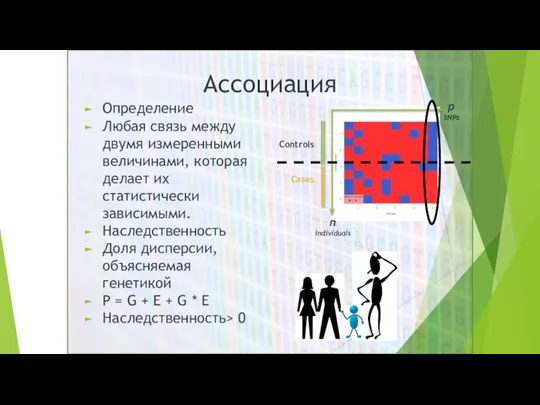
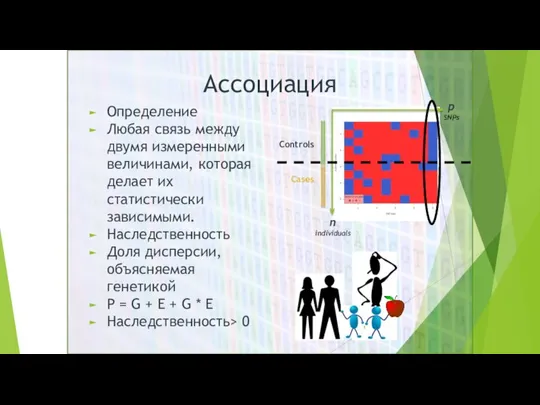
- 3. Определение Любая связь между двумя измеренными величинами, которая делает их статистически зависимыми. Наследственность Доля дисперсии, объясняемая
- 4. Определение Любая связь между двумя измеренными величинами, которая делает их статистически зависимыми. Наследственность Доля дисперсии, объясняемая
- 5. Почему? Окружающая среда, взаимодействие генов и окружающей среды Сложные черты, небольшие эффекты, редкие варианты Уровни экспрессии
- 6. Кейс-контроль Четко определенный «случай» Известная наследственность Вариации Количественные фенотипические данные Например: Рост, концентрация биомаркеров Явные модели
- 7. Процесс Визуализация Филогенетика PCA Коррекция данных Геномный контроль Регрессия по основным компонентам PCA Стратификация населения II
- 8. Что же там, в наших данных? Применим PCA - Метод главных компонент Для начала рассмотрим простые
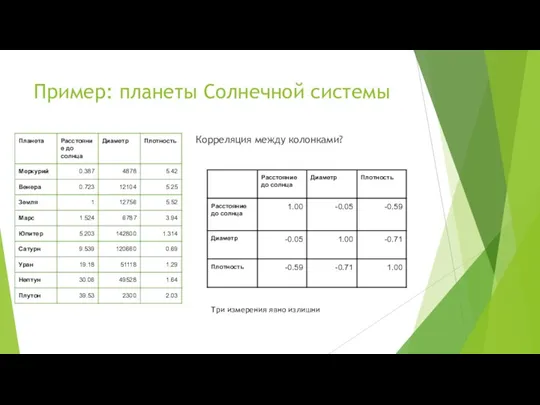
- 9. Пример: планеты Солнечной системы Как вы думаете, есть какая корреляция между колонками?
- 10. Пример: планеты Солнечной системы Корреляция между колонками? Три измерения явно излишни
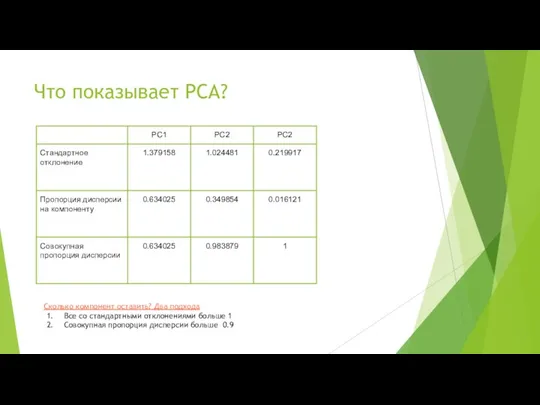
- 11. Что показывает PCA? Сколько компонент оставить? Два подхода Все со стандартными отклонениями больше 1 Совокупная пропорция
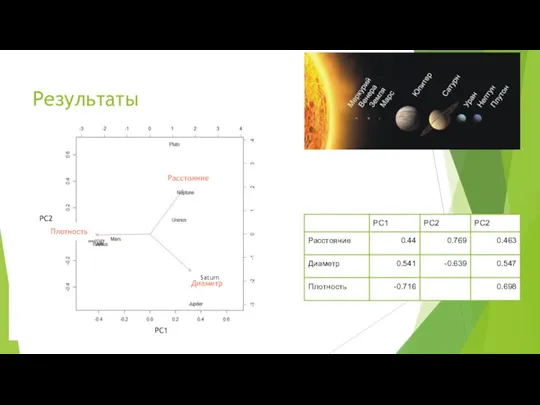
- 12. Результаты Расстояние Плотность Диаметр Saturn PC1 PC2
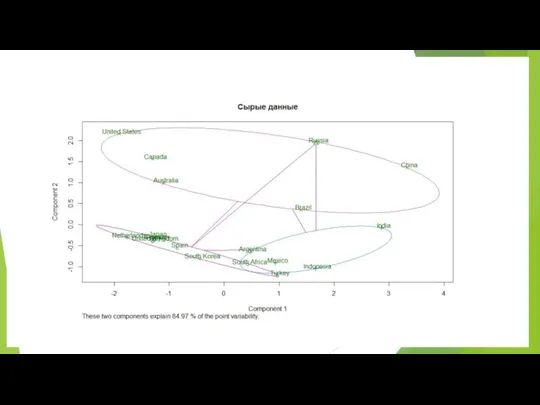
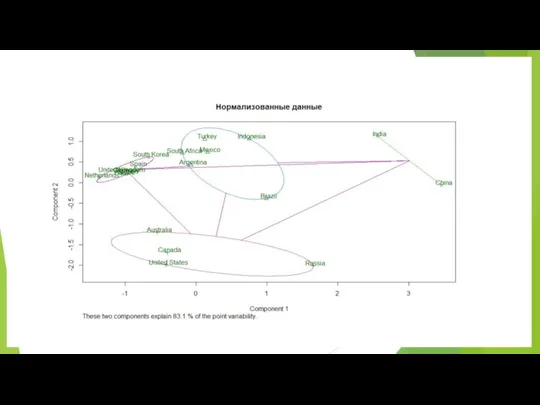
- 13. Эффект нормализации: даем равный шанс разным группам измерений С нормализацией 2 компоненты Без нормализации 1 главная
- 14. Теперь попробуйте сами # загрузите файл с данными планет # planets.csv Planets=read.csv('Planets.csv', row.names = 1); Planets
- 15. Повторим упражнение с данными о странах мира Данные Вопросы: Нужно ли проводить нормализацию? Сколько главных компонент
- 16. Результаты
- 17. Простая карта install.packages(“maptools”) library(maptools) data(wrld_simpl) myCountries = wrld_simpl@data$NAME %in% row.names(Countries) plot(wrld_simpl, col = c(gray(.80), "red")[myCountries+1])
- 18. Не совсем простая карта MAX_INCOME=max(Countries$average.income) # добавили колонку Countries$index=round(10*Countries$average.income/MAX_INCOME); Countries COL=rainbow(10) # задали цвета ALL_COUNTRIES=as.data.frame(cbind( as.character(wrld_simpl@data$NAME
- 19. K-MEANS install.packages(“cluster”) library(cluster) Countries_clusters=kmeans(Countries[,1:3], centers=4, nstart=25) clusplot(Countries, Countries_clusters$cluster,labels=3, color=TRUE) ccs=data.frame(sapply(Countries[,1:3], scale)) ##нормализация rownames(ccs)=rownames(Countries) Countries_clusters_scaled=kmeans(ccs, centers=4, nstart=25)
- 22. Возвращаемся к нуклеотидам Скачайте данные в вашу рабочую директорию sativas413.ped sativas413.fam sativas413.map sativas413.pheno sativas413.csv Мастерская GWAS
- 23. Библиотеки #install packages install.packages(c("poolr","qqman","BGLR","rrBLUP","DT", "dplyr")) install.packages(c("rnaturalearth",'rnaturalearthdata','rgeos','ggspatial')) devtools::install_github("dkahle/ggmap", ref = "tidyup") if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
- 24. Библиотеки library(rrBLUP) library(BGLR) library(DT) library(SNPRelate) library(dplyr) library(qqman) library(poolr) library(OpenStreetMap) library(rjson) library(rgdal) library(RgoogleMaps) library(mapproj) library(sf) library(OpenStreetMap) library(ggplot2)
- 25. Шаг 1. Подготовка данных SNP в R rm (список = ls ()) setwd («# ваш рабочий
- 26. Шаг 2. Считайте данные фенотипа в R ## прочитать фенотип ris.pheno header = TRUE, stringsAsFactors =
- 27. Шаг 3. Фильтрация данных SNP # Здесь мы будем использовать цикл for, чтобы определить недостающие и
- 28. Шаг 4. Структура популяции # Создать файл геноматрицы и назначить имена строк и столбцов из файлов
- 29. Шаг 5. PCA #PCA анализ pca # график результатов PCA pca plot(pca $ EV2, pca $
- 30. Шаг 5а. Визуализация PCA # Извлечь информацию о местоположении и добавить выходной файл PCA pca_country as.numeric(pca$EV4),as.numeric(pca_2$Latitude),
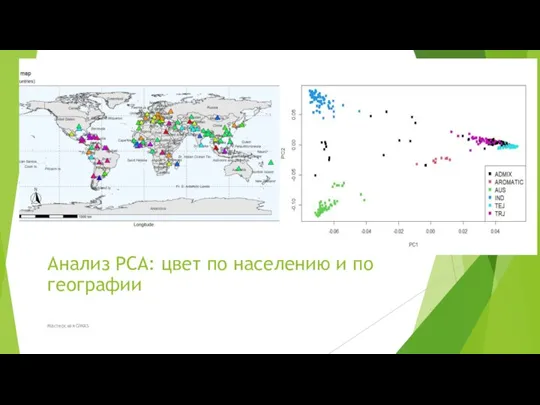
- 31. Анализ PCA: цвет по населению и по географии Мастерская GWAS
- 33. Аллели в отдельных локусах зависимы друг от друга Проблема? Да и нет Слишком много LD -
- 34. Стандартный GWAS Одномерные методы Использования взаимодействий Многовариантные методы Методы штрафной регрессии (LASSO) Факториальные методы (ФС на
- 35. Вариации Тестирование Точный критерий Фишера, критерий тенденции Кохрана-Армитиджа, критерий хи-квадрат, дисперсионный анализ Золотой стандарт - точный
- 36. Сильные стороны Недостатки Простота Вычислительно быстро Консервативный Легко интерпретировать Многомерная система, одномерная структура Размер эффекта отдельных
- 37. А как насчет взаимодействия? Тестирование на ассоциацию
- 38. Многовариантные методы Тестирование на ассоциацию Штрафная регрессия LASSO Регрессия хребта (ридж Регрессия) Нейронные сети Штрафная регрессия
- 39. GWAS Ридж регрессия (Метод регуляризации Тихонова) с использованием пакета rrBLUP Ридж регрессия BLUP (Meuwissen et al.
- 40. Код R: установите и загрузите необходимые библиотеки install.packages (c («poolr», «qqman», «BGLR», «rrBLUP», «DT», «SNPRelate», «dplyr»,
- 41. GWAS # создаем файл geno для анализа GWAS пакета rrBLUP geno_final dim(Geno1) # создаем фенофайл pheno_final
- 42. Мастерская GWAS
- 43. Использовать поиск по SNP https://snp-seek.irri.org/ Мастерская GWAS
- 44. Есть много новых инструментов Однако, если вы освоите старый добрый rrBLUP, это поможет понять другие. Теперь
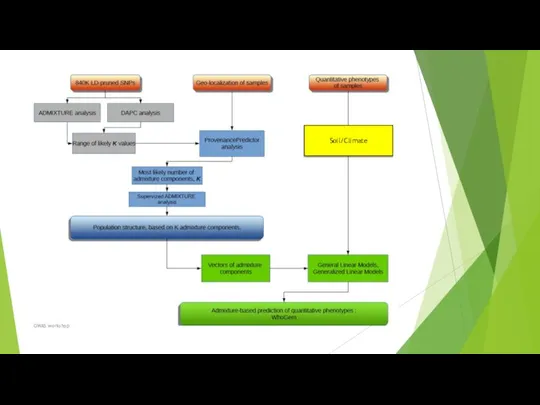
- 45. Подход к прогнозированию географической структуры населения (GPS) Сделать вывод о происхождении человека из полногеномной коллекции маркеров,
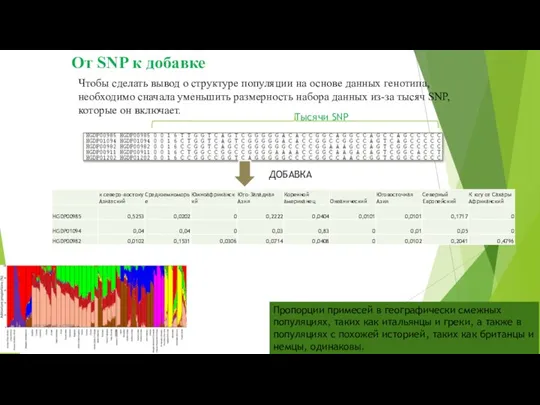
- 46. Чтобы сделать вывод о структуре популяции на основе данных генотипа, необходимо сначала уменьшить размерность набора данных
- 47. Мастерская GWAS
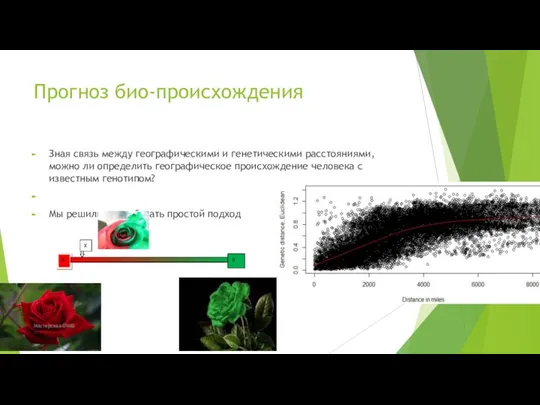
- 48. Прогноз био-происхождения Зная связь между географическими и генетическими расстояниями, можно ли определить географическое происхождение человека с
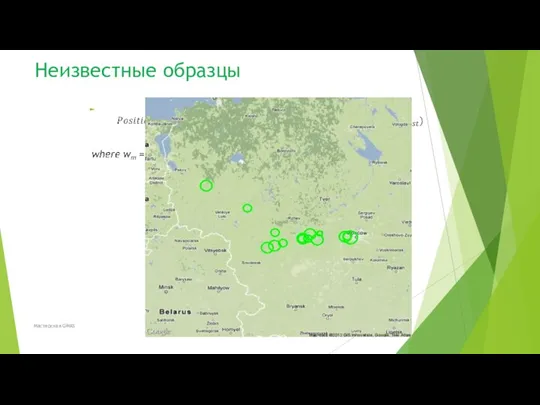
- 49. Неизвестные образцы Мастерская GWAS
- 50. GPS точно назначен ~ 100% всех людей в свои континентальные регионы 80% всех людей в страну
- 51. Моделирующая добавка
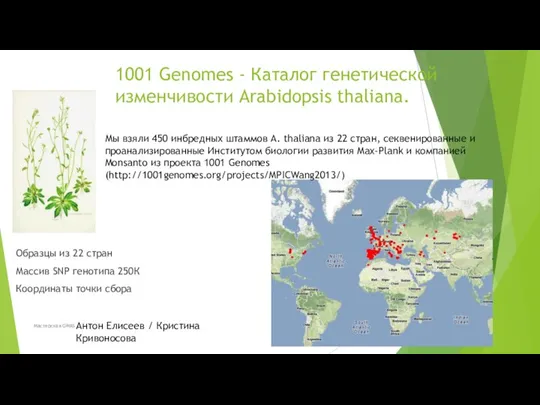
- 52. 1001 Genomes - Каталог генетической изменчивости Arabidopsis thaliana. Образцы из 22 стран Массив SNP генотипа 250К
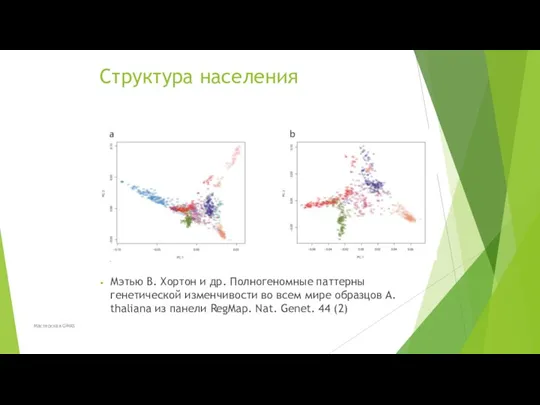
- 53. Структура населения Мэтью В. Хортон и др. Полногеномные паттерны генетической изменчивости во всем мире образцов A.
- 54. Фильтрация SNP Мы взяли файлы вариантов, доступные на http://1001genomes.org/data/MPI/MPICWang2013/releases/current/. отфильтрованы инделы и неаутосомные варианты. Обнаружено около
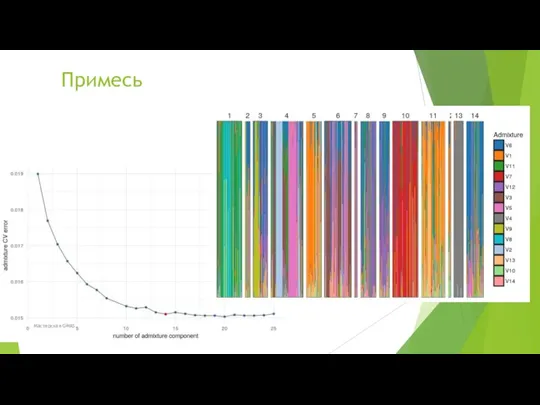
- 55. ДОБАВКА Мы выполнили несколько анализов ADMIXTURE с числом предковых популяций K от 3 до 20, а
- 56. Примесь Мастерская GWAS
- 57. Проверка GPS по одному разу Процент популяций, которые точно нанесены на карту 60% Среднее расстояние до
- 58. Исходная гипотеза Географическое положение Arabidopsis должно быть связано с холодоустойчивостью. Более холодный климат в Северном полушарии
- 59. GPS-анализ O. sativa Мастерская GWAS
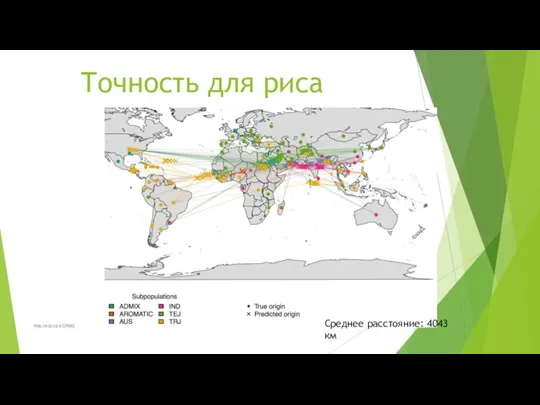
- 60. Точность для риса Среднее расстояние: 4043 км Мастерская GWAS
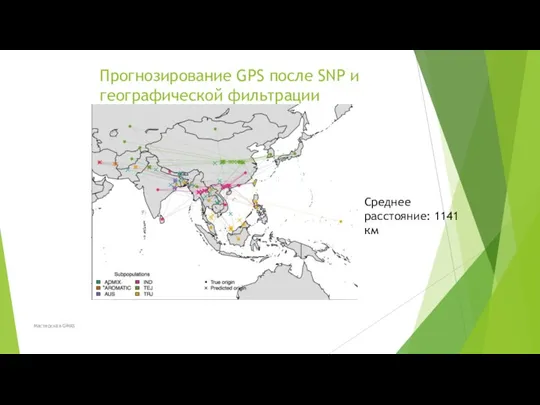
- 61. Прогнозирование GPS после SNP и географической фильтрации Среднее расстояние: 1141 км Мастерская GWAS
- 62. Почему не работает с рисом? Рис не может выбрать свою вторую половинку, а арабидопсис может. Мастерская
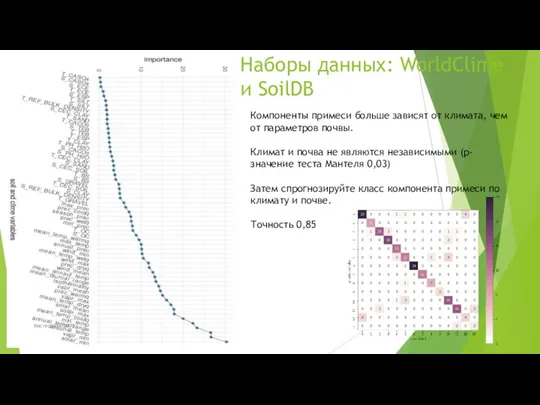
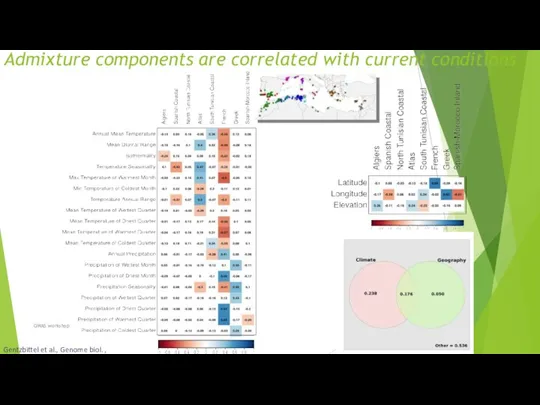
- 63. Наборы данных: WorldClime и SoilDB Компоненты примеси больше зависят от климата, чем от параметров почвы. Климат
- 64. Часть 1 заключение Мы умеем моделировать дикие виды - не так уж и много с одомашненными
- 65. Семейство бобовых (бобовых) растений Возможность установления клубенькового симбиоза Высокая синтения к бобовым культурам Небольшой диплоидный геном
- 66. Проект NSF HapMap Текущее состояние: 288 последовательных образцов. Итого: 16.516.721 SNP 30 строк при> 20X Остальные
- 67. 12.11.2020 Зерновые бобовые Кормовые бобовые Люцерна Клевер Соя Нута Фасоль Высокая сельскохозяйственная ценность Горох Арахис Чечевица
- 68. Данные HapMap для M. truncatula Текущее состояние: 288 последовательных образцов. # SNP Chr1: 1,508,346 Chr2: 1.964.419
- 69. M. truncatula спонтанно встречается по всему Средиземноморскому бассейну. Доступно несколько коллекций: DZ, TN, FR, AU, US,….
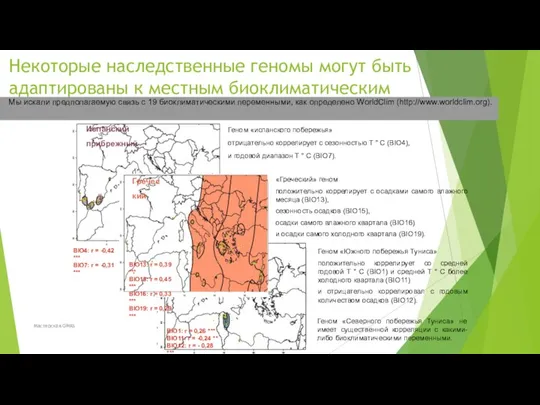
- 70. Некоторые наследственные геномы могут быть адаптированы к местным биоклиматическим переменным. Мы искали предполагаемую связь с 19
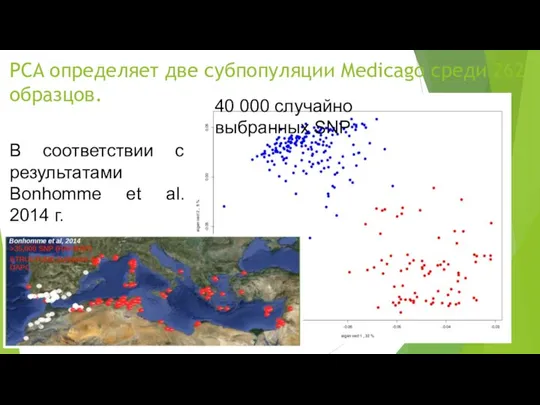
- 71. PCA определяет две субпопуляции Medicago среди 262 образцов. 40 000 случайно выбранных SNP В соответствии с
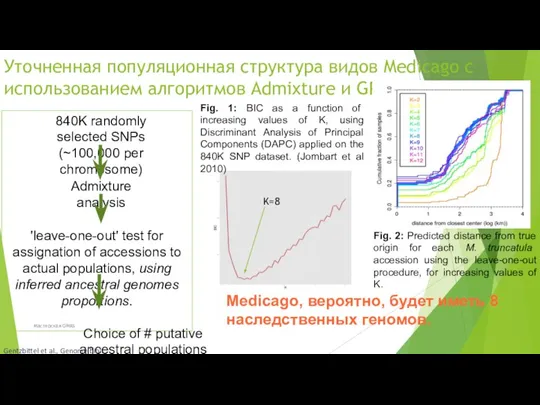
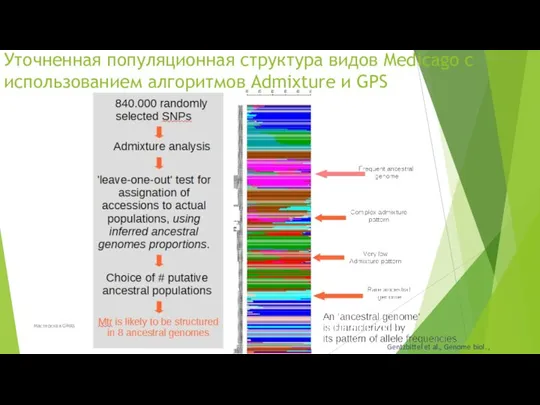
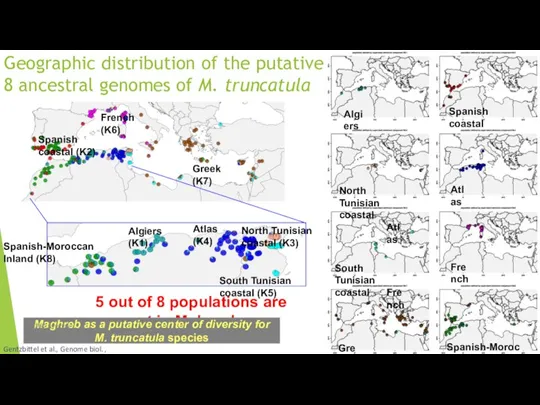
- 72. Уточненная популяционная структура видов Medicago с использованием алгоритмов Admixture и GPS Medicago, вероятно, будет иметь 8
- 73. Уточненная популяционная структура видов Medicago с использованием алгоритмов Admixture и GPS Gentzbittel et al., Genome biol.
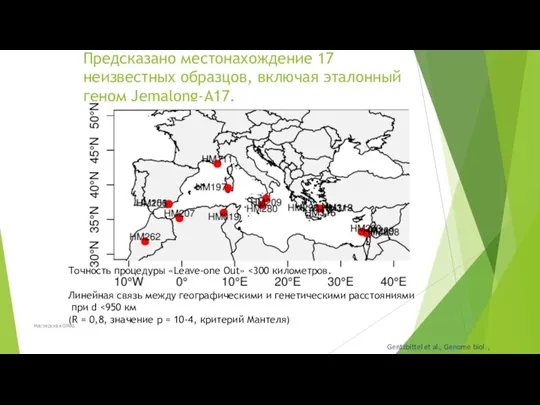
- 74. Предсказано местонахождение 17 неизвестных образцов, включая эталонный геном Jemalong-A17. Точность процедуры «Leave-one Out» Линейная связь между
- 75. Уточненная популяционная структура видов Medicago с использованием алгоритмов Admixture и GPS Мастерская GWAS
- 76. Geographic distribution of the putative 8 ancestral genomes of M. truncatula Spanish coastal Algiers North Tunisian
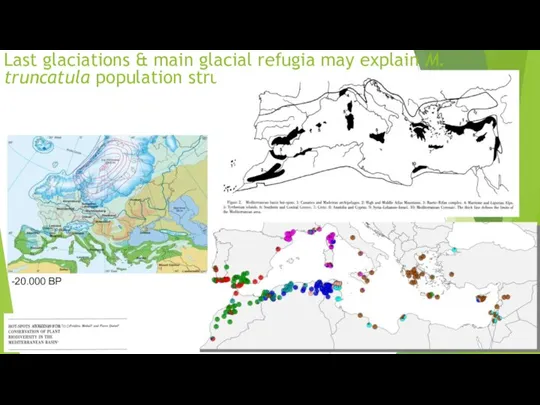
- 77. Last glaciations & main glacial refugia may explain M. truncatula population structure -20.000 BP GWAS workshop
- 78. Admixture components are correlated with current conditions Gentzbittel et al., Genome biol. , 2019 GWAS workshop
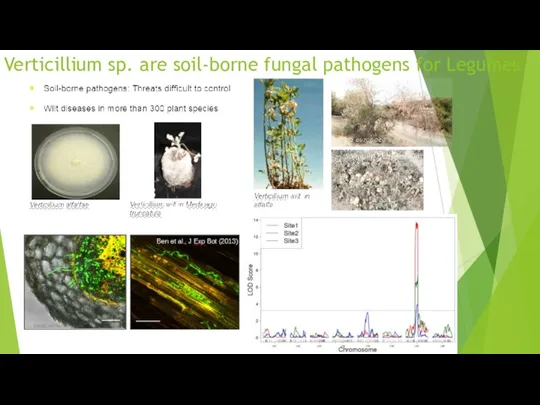
- 79. Verticillium sp. are soil-borne fungal pathogens for Legumes GWAS workshop
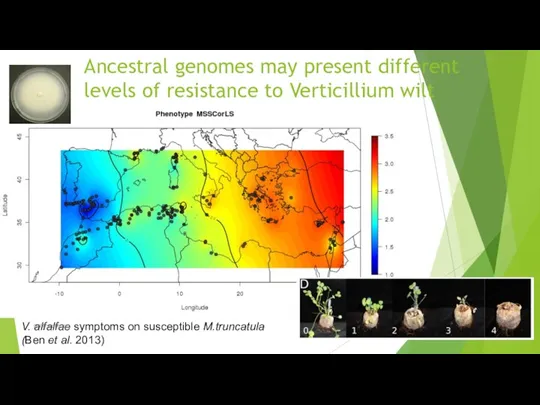
- 80. Ancestral genomes may present different levels of resistance to Verticillium wilt V. alfalfae symptoms on susceptible
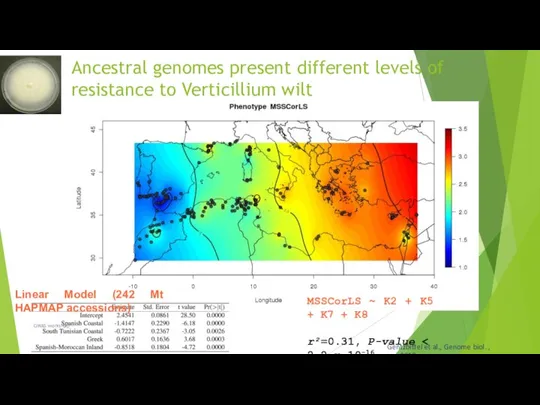
- 81. Ancestral genomes present different levels of resistance to Verticillium wilt Linear Model (242 Mt HAPMAP accessions)
- 82. Can phenotype be predicted using WhoGem? Use plant model (Medicago truncatula) to test applicability of admixture
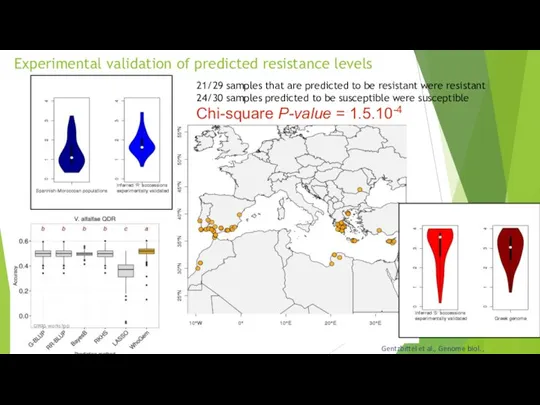
- 83. Experimental validation of predicted resistance levels 21/29 samples that are predicted to be resistant were resistant
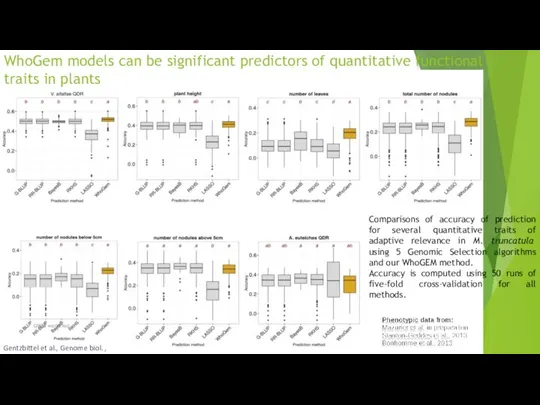
- 84. WhoGem models can be significant predictors of quantitative functional traits in plants Comparisons of accuracy of
- 85. What is missing? Distance between two points should not be just geometric distance. Add: Mode of
- 86. GWAS workshop
- 87. GWAS workshop
- 88. GWAS workshop
- 89. This methodology may serve as a basis for analyses in other plant species and for other
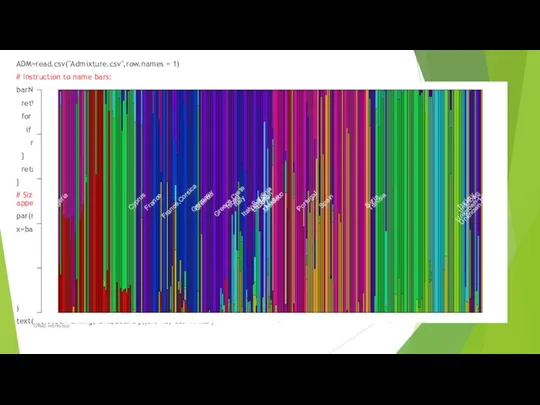
- 90. Practice Admixture.csv Pheno.csv GWAS workshop
- 91. ADM=read.csv("Admixture.csv",row.names = 1) # Instruction to name bars: barNaming retVec for (k in 2:length(vec)) { if
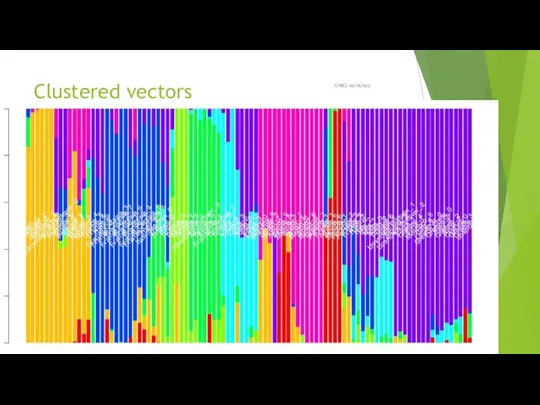
- 92. Clustered vectors GWAS workshop
- 93. Merge datasets ADM=read.csv('Admixture.csv',row.names = 1); TEST=subset(ADM, is.na(ADM$Lat)) REF=subset(ADM, !is.na(ADM$Lat)); Phen=read.csv('pheno.csv',row.names = 1); head(Phen) #merge reference adm
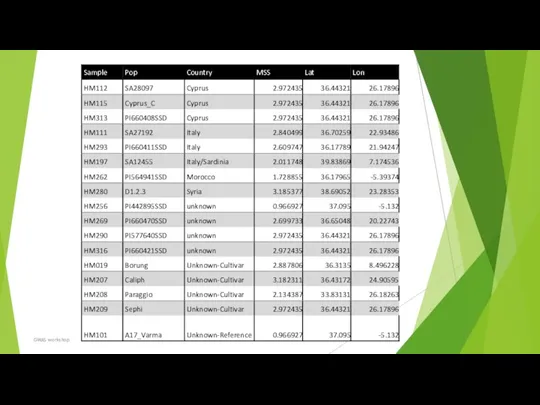
- 94. DATA=AMDPH[,c(4:6,8:15,17)]; cor(DATA) GWAS workshop
- 95. ##GPS M=25; for(i in 1:dim(TEST)[1]){ Y=TEST[i,7:14];Y DIST=c() for(j in 1:dim(AMDPH)[1]){ Z=AMDPH[j,8:15];Z d=sum((Y-Z)^2);d DIST=c(DIST,d) } I=order(DIST)[1:M];I Ph=AMDPH$MaxSymptomScore[I];Ph
- 96. Results GWAS workshop
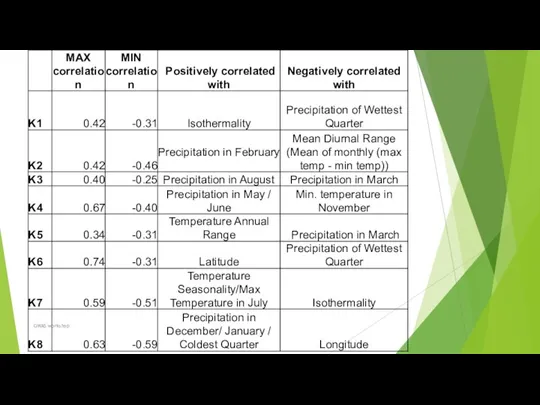
- 97. #GLM library(lmtest); library(dplyr); library(car); library(rcompanion) #set up formulas formula=formula(MaxSymptomScore~K1+K2+K3+K4+K5+K6+K7+K8) formula0=MaxSymptomScore ~ 1 #initialize models model.null =
- 98. Laurent Gentzbittel, Ecolab, Toulouse, France/Skoltech, Moscow, Russia Nevin Young's lab, Univ. of Minnesota, USA Sergey Nuzdhin's
- 100. Скачать презентацию










![Простая карта install.packages(“maptools”) library(maptools) data(wrld_simpl) myCountries = wrld_simpl@data$NAME %in% row.names(Countries) plot(wrld_simpl, col = c(gray(.80), "red")[myCountries+1])](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1062822/slide-16.jpg)

![K-MEANS install.packages(“cluster”) library(cluster) Countries_clusters=kmeans(Countries[,1:3], centers=4, nstart=25) clusplot(Countries, Countries_clusters$cluster,labels=3, color=TRUE) ccs=data.frame(sapply(Countries[,1:3], scale)) ##нормализация](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1062822/slide-18.jpg)











































![DATA=AMDPH[,c(4:6,8:15,17)]; cor(DATA) GWAS workshop](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1062822/slide-93.jpg)
![##GPS M=25; for(i in 1:dim(TEST)[1]){ Y=TEST[i,7:14];Y DIST=c() for(j in 1:dim(AMDPH)[1]){ Z=AMDPH[j,8:15];Z d=sum((Y-Z)^2);d](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1062822/slide-94.jpg)


 Кровообращение, лимфообращение
Кровообращение, лимфообращение Условно-рефлекторное поведение и научение. Концепция И.П. Павлова
Условно-рефлекторное поведение и научение. Концепция И.П. Павлова Карнотавр - род хищных динозавров из семейства абелизаврид
Карнотавр - род хищных динозавров из семейства абелизаврид Селекция сортов перекрёстноопыляющихся культур
Селекция сортов перекрёстноопыляющихся культур Давайте внимательно посмотрим вокруг. Онлайн-экскурсия в природу
Давайте внимательно посмотрим вокруг. Онлайн-экскурсия в природу Ассимиляционная и диссимиляционная нитратредуктации
Ассимиляционная и диссимиляционная нитратредуктации Haemophilus influenzae. Таксономия
Haemophilus influenzae. Таксономия Жизнь растений осенью
Жизнь растений осенью Адаптации млекопитающих к образу жизни на примере речного бобра
Адаптации млекопитающих к образу жизни на примере речного бобра Витамины и их значение для организма
Витамины и их значение для организма Ботаника (ОГЭ, часть 2)
Ботаника (ОГЭ, часть 2) Сердце - это жизнь
Сердце - это жизнь Насекомые
Насекомые Общая характеристика грибов
Общая характеристика грибов Мышцы груди и живота
Мышцы груди и живота Клеточный цикл. Деление клетки. Классическая генетика
Клеточный цикл. Деление клетки. Классическая генетика Женская половая система - 1. Яичник. Жёлтое тело
Женская половая система - 1. Яичник. Жёлтое тело Ткани, органы, системы органов
Ткани, органы, системы органов Направление цепей белков и нуклеиновых кислот. Репликация. Транскрипция
Направление цепей белков и нуклеиновых кислот. Репликация. Транскрипция Синдром Дауна
Синдром Дауна Дивні майстри маскування
Дивні майстри маскування Комнатные растения
Комнатные растения Черепные, или позвоночные. Внешнее и внутреннее строение рыб
Черепные, или позвоночные. Внешнее и внутреннее строение рыб Материалы к экзамену: Митоз в клетках кончика корешка лука. Генетика. Онто-филогенз. Имбриогенеза у различных классов животных
Материалы к экзамену: Митоз в клетках кончика корешка лука. Генетика. Онто-филогенз. Имбриогенеза у различных классов животных Технология рекомбинантной ДНК
Технология рекомбинантной ДНК Аномалии развития позвоночника
Аномалии развития позвоночника Различие в содержании вдыхаемого и выдыхаемого воздуха. Кислород. Окисление сложных органических веществ
Различие в содержании вдыхаемого и выдыхаемого воздуха. Кислород. Окисление сложных органических веществ Вегетативная нервная система
Вегетативная нервная система