Содержание
- 2. Что такое множественное выравнивание? Несколько гомологичных последовательностей, написанных друг под другом оптимальным способом: Гомологичные остатки один
- 3. Какое выравнивание интереснее?
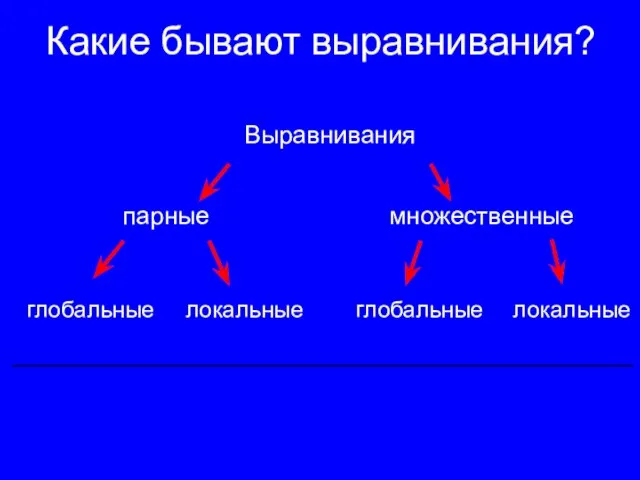
- 4. Какие бывают выравнивания? локальные глобальные локальные глобальные множественные парные Выравнивания
- 5. Зачем нужно множественное выравнивание? Перенос аннотации Предсказание функции каждого остатка (например, выявление остатков, составляющих активный центр
- 6. Как выбрать последовательности для множественного выравнивания? Выравнивайте белки, а не ДНК, если есть выбор Последовательностей лучше
- 7. Изучая новую последовательность Выборка на основе BLAST Подробно охарактеризованные последовательности - аннотация Совсем неохарактеризованные (hypothetical proteins)
- 8. Подготовка выборки BLAST => сохранить все последовательности разом в FASTA формате или сразу на выравнивание Имена
- 9. Как можно строить глобальное множественное выравнивание? Построение множественного выравнивания N последовательностей t =LN !!! Можно пытаться
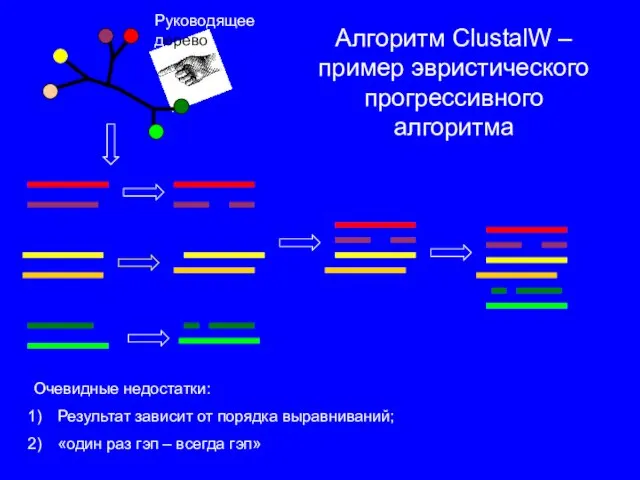
- 10. Алгоритм ClustalW – пример эвристического прогрессивного алгоритма Руководящее дерево Очевидные недостатки: Результат зависит от порядка выравниваний;
- 11. Современные методы построения множественного выравнивания (MSA, multiple sequence alignment): Алгоритм ClustalW (реализации ClustalX, emma из EMBOSS)
- 12. Использование ClustalW
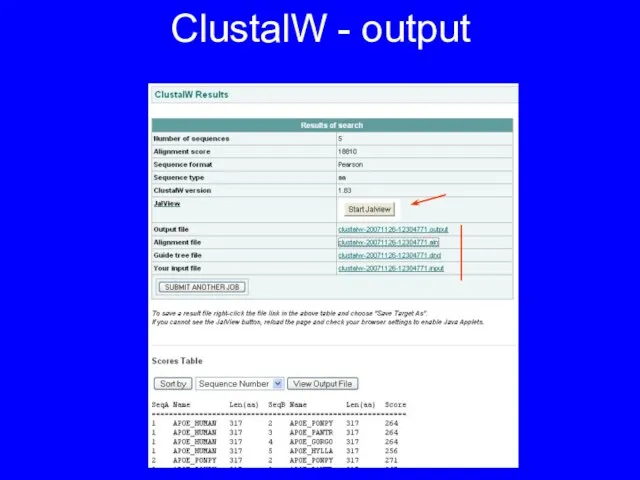
- 13. Какие output-форматы бывают Post-script, pdf, html – только графика FASTA – последовательности отдельно, но с пробелами
- 14. Перевод форматов: READSEQ (http://www-bimas.cit.nih.gov/molbio/readseq/) Аналогично: SEQCHECK
- 15. ClustalW - output
- 16. JalView – редактирование выравниваний Другие программы для редактирования выравниваний (stand-alone): GeneDoc; CINEMA; Seaview; Belvu; Bioedit; DCSE
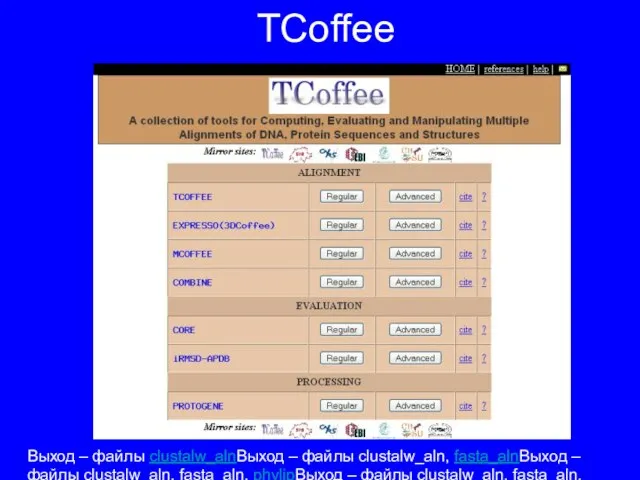
- 17. TCoffee Построение множественных выравниваний Оценка достоверности существующего выравнивания Использование 3-D структуры при построении выравнивания Сравнение и
- 18. TCoffee Выход – файлы clustalw_alnВыход – файлы clustalw_aln, fasta_alnВыход – файлы clustalw_aln, fasta_aln, phylipВыход – файлы
- 19. Как использовать TCoffee для других целей Множественное выравнивание на основе 3D-структуры (Expresso): надо заменить 1 или
- 20. Как “читать” множественное выравнивание? Хорошее выравнивание – высоко-консервативные блоки, перемежающиеся блоками с инсерциями/делециями ДНК – консервативные
- 21. Если консервативны только отдельные столбцы W, Y, F – консервативное гидрофобное ядро, стабилизирующая роль в ядре.
- 22. Локальное множественное выравнивание – постановка задачи Ряд последовательностей, в каждой из которых есть интересное слово (либо
- 23. dnaN ACATTATCCGTTAGGAGGATAAAAATG gyrA GTGATACTTCAGGGAGGTTTTTTAATG serS TCAATAAAAAAAGGAGTGTTTCGCATG bofA CAAGCGAAGGAGATGAGAAGATTCATG csfB GCTAACTGTACGGAGGTGGAGAAGATG xpaC ATAGACACAGGAGTCGATTATCTCATG metS ACATTCTGATTAGGAGGTTTCAAGATG gcaD AAAAGGGATATTGGAGGCCAATAAATG
- 24. Gibbs sampler Let’s A be a signal (set of sites), and I(A) be its information content.
- 25. Соответствующие программы
- 26. Представление результатов таких программ – Logos Программы построения – http://www-lmmb.ncifcrf.gov/~toms/sequencelogo.html; http://www.cbs.dtu.dk/~gorodkin/appl/plogo.html
- 27. Greedy algorithms (MEME) Find a signal among all k-words (assuming that we know the length signal).
- 28. Greedy algorithms. Cont’d Select the k-word with maximal information content Problem. We considered only k-words from
- 29. Limitation of greedy algorithms Started from k-words in our sequences and increase the information content at
- 31. Скачать презентацию






















 Сайт коммерческой фирмы. Макет сайта
Сайт коммерческой фирмы. Макет сайта Разработка факсимильного шрифта на основе собственного почерка. Программы для создания собственного шрифта
Разработка факсимильного шрифта на основе собственного почерка. Программы для создания собственного шрифта Способы управления. Клавиатура и мышь
Способы управления. Клавиатура и мышь Библиотека конвертеров данных eCAD-КОМПАС
Библиотека конвертеров данных eCAD-КОМПАС Новый модуль электронного сервиса по регистрации: выбор типового устава
Новый модуль электронного сервиса по регистрации: выбор типового устава Разработка авторулевого для морских и речных судов
Разработка авторулевого для морских и речных судов Cети ЦВМ - системообразующий элемент сложных технических систем
Cети ЦВМ - системообразующий элемент сложных технических систем Нормализация баз данных
Нормализация баз данных Остросоциальные компьютерные игры
Остросоциальные компьютерные игры Создатели Viber
Создатели Viber Кибернетические системы. Лекция 3
Кибернетические системы. Лекция 3 Kurilka Gutenberga
Kurilka Gutenberga Мониторинг и статистика PS Core на основе Perfomance Report System (PRS)
Мониторинг и статистика PS Core на основе Perfomance Report System (PRS) Построение линейных базовых примитивов в AutoCAD
Построение линейных базовых примитивов в AutoCAD Конструирование информационного ресурса создание собственной альтернативной повестки
Конструирование информационного ресурса создание собственной альтернативной повестки Информационные носители
Информационные носители Изучение модели СМО с повторными заявками в Rockwell Arena
Изучение модели СМО с повторными заявками в Rockwell Arena Красота, вопросы по ТОРу. Правильные ответы
Красота, вопросы по ТОРу. Правильные ответы World Of Tanks. Мир танков
World Of Tanks. Мир танков JavaScript История развития
JavaScript История развития Проектирование системы сбора и корреляции событий информационной безопасности
Проектирование системы сбора и корреляции событий информационной безопасности Обчислювальні кластери в корпоративній мережі. Їх архітектура
Обчислювальні кластери в корпоративній мережі. Їх архітектура Кодирование информации
Кодирование информации Электронные таблицы
Электронные таблицы База данных Access
База данных Access Приложение Кошелёк: история борьбы с пластиком
Приложение Кошелёк: история борьбы с пластиком Информация и информационные процессы
Информация и информационные процессы Составление адреса URL из частей
Составление адреса URL из частей