Слайд 2 ДОСТУПНЫЕ ПАКЕТЫ для ДОКИНГА :
DOCK4.0, FlexX1.8, GOLD1.2, AutoDock4

находят структуры комплексов белок-лиганд
- Успешный докинг RMSD 2.0 A ~
вероятность 40-60 %
[Chen et al, J Comp.Chem. 2007, 28:612-623;
Предсказание абсолютной энергии связывания ??
нет надежных методов
Слайд 3ДОКИНГ:
ПОИСК ОПТИМАЛЬНОГО МЕСТА (СТРУКТУРЫ) СВЯЗЫВАНИЯ
ЛИГАНДА С МОЛЕКУЛОЙ БЕЛКА
DOCKING – GLOBAL

OPTIMIZATION PROBLEM
для структуры комплекса белок-лиганд
Фазовое пространство задачи (степени свободы):
- позиция, ориентация, конформация лиганда
- конформация белка – индуцированная подстройка
- Метод Глобальной оптимизации ??
Целевая функция
Свободная энергия комплекса – не вычисляема
- Разумные аппроксимации
reliable scoring function ?
должна отличать нативный комплекс от неправильного
Слайд 4Scoring function - two groups:
Empirical scoring function – weighted sum of

terms or descriptors, i.e. different energy terms,
weights are estimated on training set -
LIMITED TRANSFERABILYTY beyond of training P/L set
2) PMF – atom-atom potentials derived from 3D-dataset for P/L complexes, as a probability to find atom pair at a given distance – WEEK STAT.MECH. FOUNDATION – limited accuracy, SR ~ 55-70%
3) physics based scoring functions, i.e. Force Field used in atom-atom simulation of protein structure,
- highest rate of transferability
Наиболее надежны аппроксимации свободной энергии связывания =
потенциальная энергия + энергия сольватации + энтропия
Слайд 5We present the docking method MdDOCK which relay on:
1) physics

based approaches
2) USE exhaustive hierarchical search for binding sites
on protein surface
3) USE physics based scoring function =
BINDING ENERGY : atom-atom potentials +
electrostatics + solvation model
4) global optimization for ligand and receptor conformations via molecular dynamics coupled with simulated annealing and force field deformation
Слайд 6Глобальная оптимизация в задаче ДОКИНГА
Подход в лоб не продуктивен –
известные

методы используют:
- ручное ограничение исследуемого фазового
пространства - ОГРАНИЧИВАЕТСЯ область докинга
на поверхности белка -
НЕ ПРИЕМЛЕМО ДЛЯ СЛЕПОГО ДОКИНГА
Варианты генетического метода глобальной
оптимизации + локальные методы
Слайд 7
ДОКИНГ – подобен самоорганизации
поверхность потенциальной энергии – ВОРОНОЧНАЯ-
Область низкой энергии

в фазовом
пространстве – сайт связывания
в низком разрешении
Точная структура комплекса
белок-лиганд
- сайт в высоком разрешении
Слайд 8
РАЗРАБАТЫВАЕТСЯ-
метод слепого иерархического ДОКИНГА
1) исчерпывающий анализ молекулярной поверхности молекулы белка

– поиск всех полостей, карманов, складок
- локализация позиций связывания в низком разрешении ~ 3 Å
2) Глобальная оптимизация позиции, ориентации,
конформации лиганда,
- оптимизация индуцированной подстройки белка
Глобальная оптимизация на основе
- метода Молекулярной Динамики
- деформация поверхности потенциальной энергии +
отжиг по температуре,
множественный старт из разных ориентаций(конформаций) лиганда из сайта низкого разрешения
Слайд 9Определение сайтов связывания низкого разрешения:
1 – Расчет поверхности молекулы белка

ДОСТУПНОЙ сфере радиуса 1.4
2 – анализ и определение позиций центров
полостей, карманов и складок
на поверхности молекулы белка =
сайтов низкого разрешения
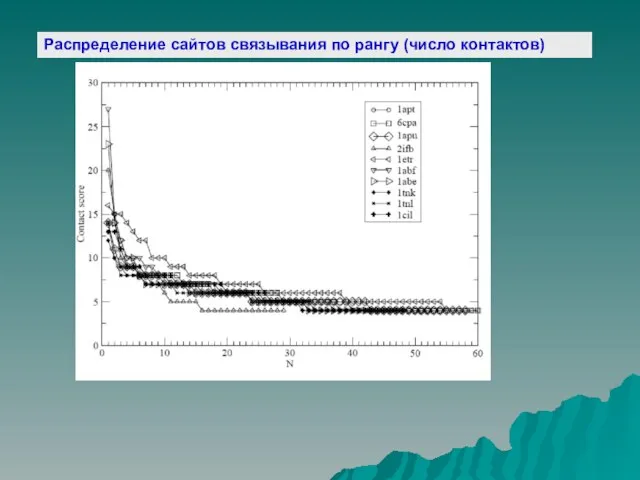
3 – Расчет ранга (числа контактов) сайтов низкого разрешения
4 - Определение сайтов связывания низкого разрешения с
наибольшим рангом
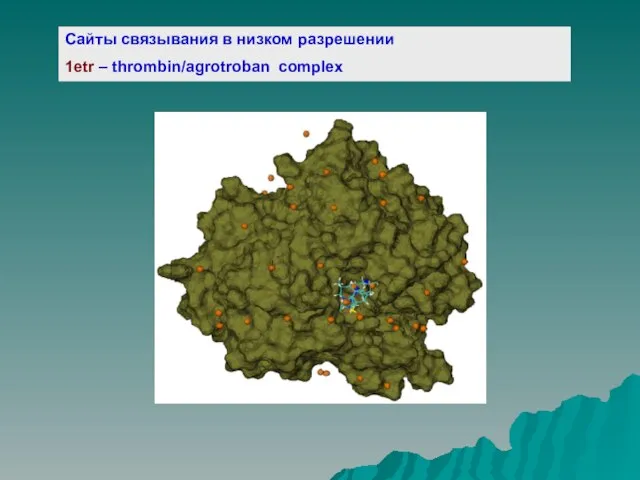
Слайд 10Сайты связывания в низком разрешении
1etr – thrombin/agrotroban complex
Слайд 11Распределение сайтов связывания по рангу (число контактов)
Слайд 12Определение приближенной ориентации лиганда:
Точечный образ лиганда ? натягивается на сайты связывания низкого
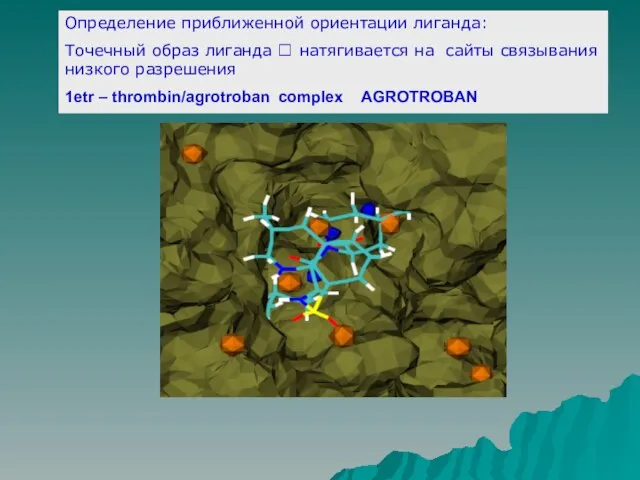
разрешения
1etr – thrombin/agrotroban complex AGROTROBAN
Слайд 13МД глобальная оптимизация:
- молекулярная динамика для лиганда в окрестности сайта

связывания низкого разрешения
- температурный отжиг + деформация поверхности
потенциальной энергии (стимуляция конформационных
переходов)
- Force Field – AMBER99 for VDW +
+ modified electrostatics
+ explicit HydrogenBonds
+ Solvation
Слайд 14Тrypsine/ benzamidine complex.
A – ранг низкого разрешения для сайтов связывания
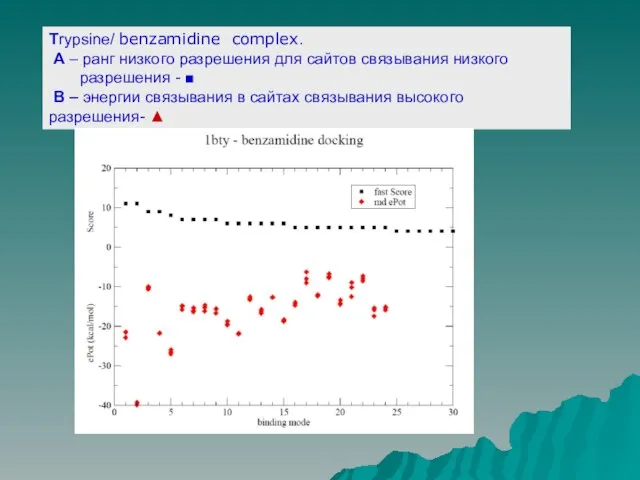
низкого
разрешения - ■
B – энергии связывания в сайтах связывания высокого разрешения- ▲
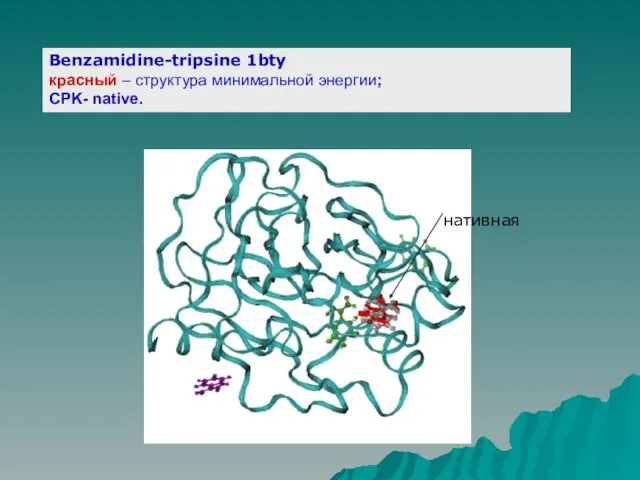
Слайд 15Benzamidine-tripsine 1bty
красный – структура минимальной энергии;
CPK- native.
нативная
Слайд 161dwb : thrombin/ benzamidin complex
A – ранг низкого разрешения для сайтов связывания
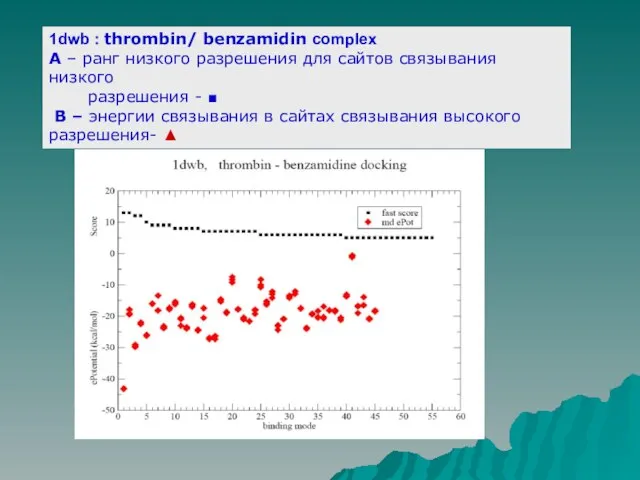
низкого
разрешения - ■
B – энергии связывания в сайтах связывания высокого разрешения- ▲
Слайд 17Docking results
1dwb : thrombin + benzamidine complex
Структура минимальной энергии - синий;
НАТИВНЫЙ-CPK-
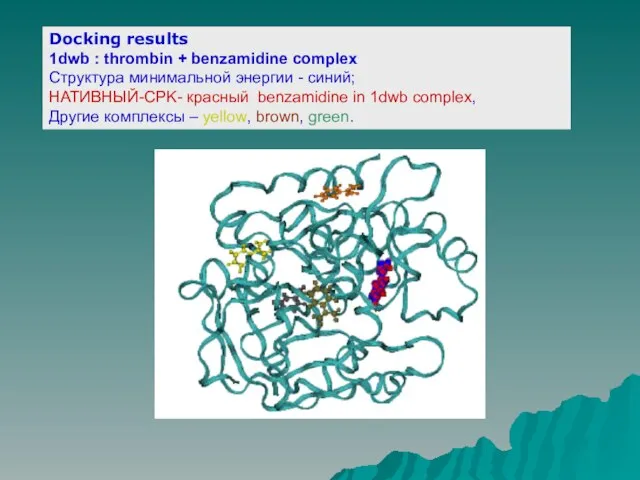
красный benzamidine in 1dwb complex,
Другие комплексы – yellow, brown, green.
















 Мир геометрических фигур
Мир геометрических фигур Презентация на тему Организация и содержание физминутки на уроке
Презентация на тему Организация и содержание физминутки на уроке Мы за Новый мир! Движение за Новый Коммунизм
Мы за Новый мир! Движение за Новый Коммунизм Как выполнить выгрузкуотчетности в электронном виде из программ системы 1С:Предприятие
Как выполнить выгрузкуотчетности в электронном виде из программ системы 1С:Предприятие Неиспользуемые здания в коммунальной собственности Мостовского района, планируемые к продаже в 2012 году
Неиспользуемые здания в коммунальной собственности Мостовского района, планируемые к продаже в 2012 году СПП нерасчлененной структуры
СПП нерасчлененной структуры Курсовая работа
Курсовая работа Транспортное средство. Характеристика непрофильного актива
Транспортное средство. Характеристика непрофильного актива «Определение характерных черт портрета успешного предпринимателя »Проект Подготовили: Любченко Алёна и Няргинен ВикторияРуков
«Определение характерных черт портрета успешного предпринимателя »Проект Подготовили: Любченко Алёна и Няргинен ВикторияРуков И.И. Шишкин
И.И. Шишкин Круг обучения. Готовим профессионалов.
Круг обучения. Готовим профессионалов. Конституция Тыва
Конституция Тыва «Дело-КС» Автоматизация делопроизводства
«Дело-КС» Автоматизация делопроизводства Охрана материнства и детства в свете приоритетных правительственных программ Директор Департамента медико-социальных проблем с
Охрана материнства и детства в свете приоритетных правительственных программ Директор Департамента медико-социальных проблем с Тема проекта:«Здоровьесберегающие компоненты математического образования младшего школьника в свете стандарта второго поколен
Тема проекта:«Здоровьесберегающие компоненты математического образования младшего школьника в свете стандарта второго поколен Тургенев и Виардо
Тургенев и Виардо Менеджер по компенсациям и льготам. Профпереподготовка
Менеджер по компенсациям и льготам. Профпереподготовка 7 период развития соц.работы
7 период развития соц.работы Рынок труда: состав, состояние и перспективы развития.
Рынок труда: состав, состояние и перспективы развития. Игровые технологии в младшем школьном возрасте
Игровые технологии в младшем школьном возрасте Открытки с 9 мая
Открытки с 9 мая О введении комплексного курса
О введении комплексного курса Презентация на тему Народы России
Презентация на тему Народы России  Изменчивость. Мутации
Изменчивость. Мутации Презентация на тему Мастерство педагога
Презентация на тему Мастерство педагога Сирень 1945 года
Сирень 1945 года Методы составления и анализа семантического ядра
Методы составления и анализа семантического ядра Презентация на тему Одиночество
Презентация на тему Одиночество