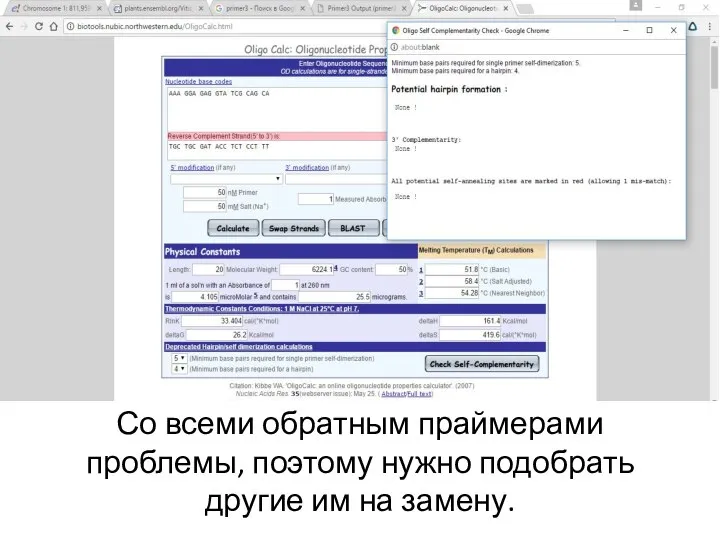
Слайд 10Со всеми обратным праймерами проблемы, поэтому нужно подобрать другие им на замену.
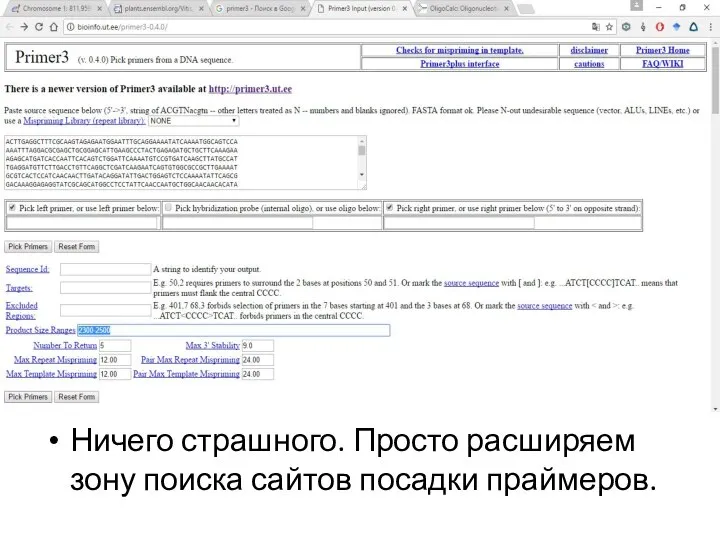
Слайд 11Ничего страшного. Просто расширяем зону поиска сайтов посадки праймеров.
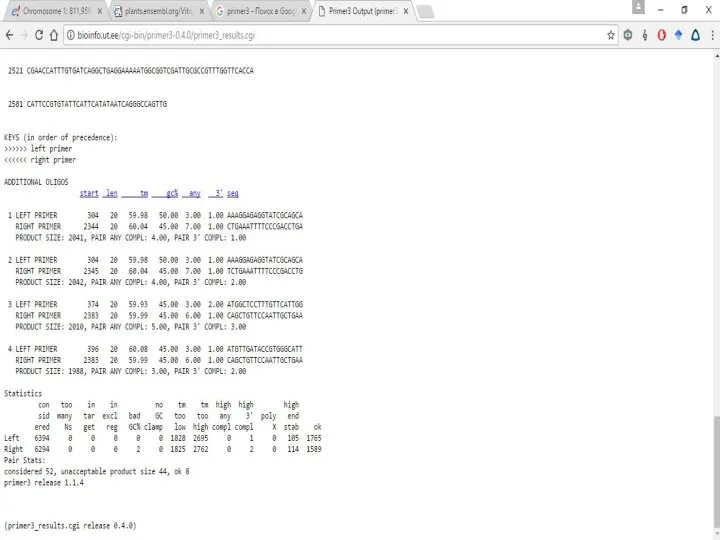
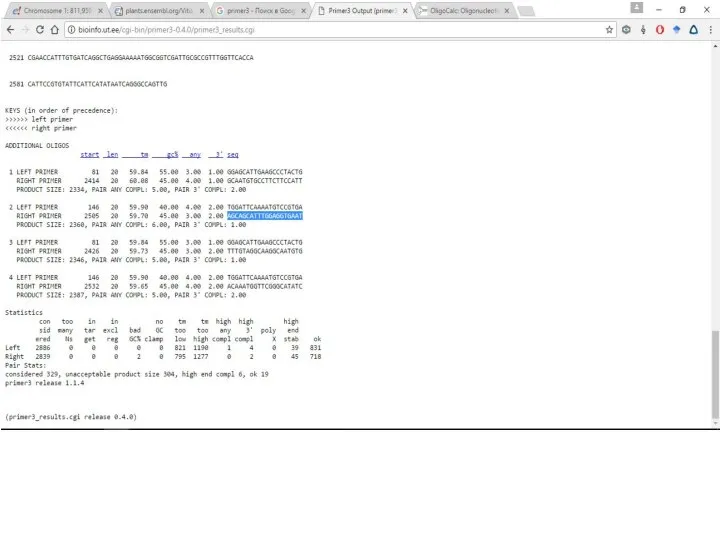
Слайд 13Остальные два праймера тоже хорошо подходят, но тут нужно выбирать в зависимости
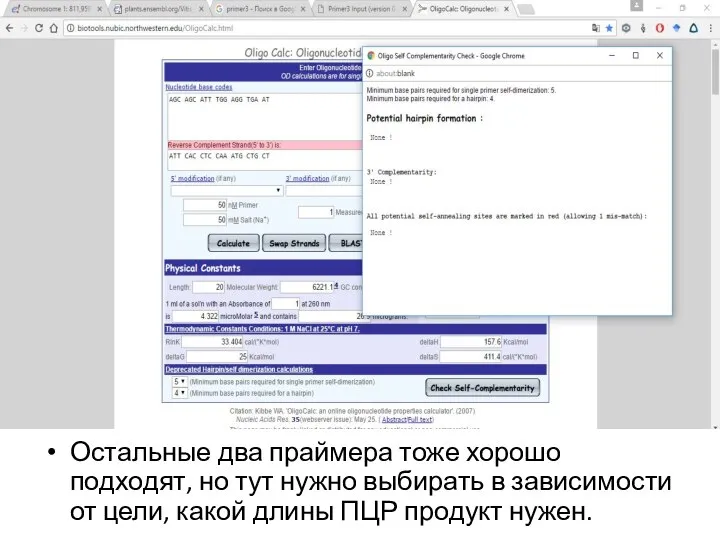
от цели, какой длины ПЦР продукт нужен.
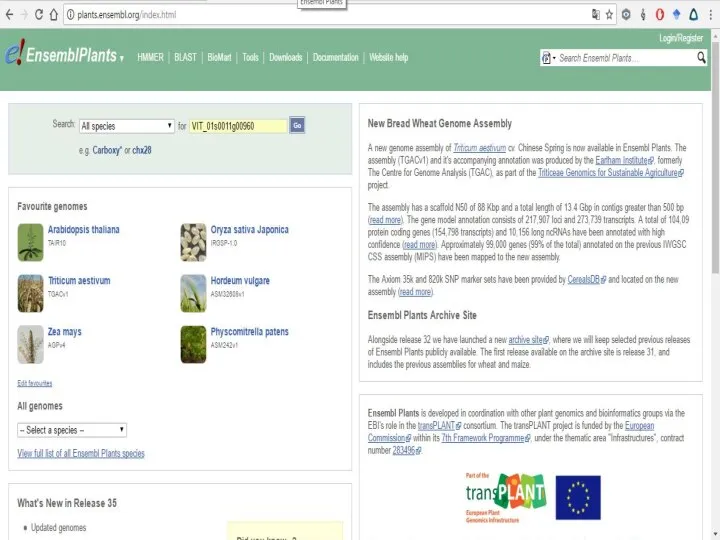
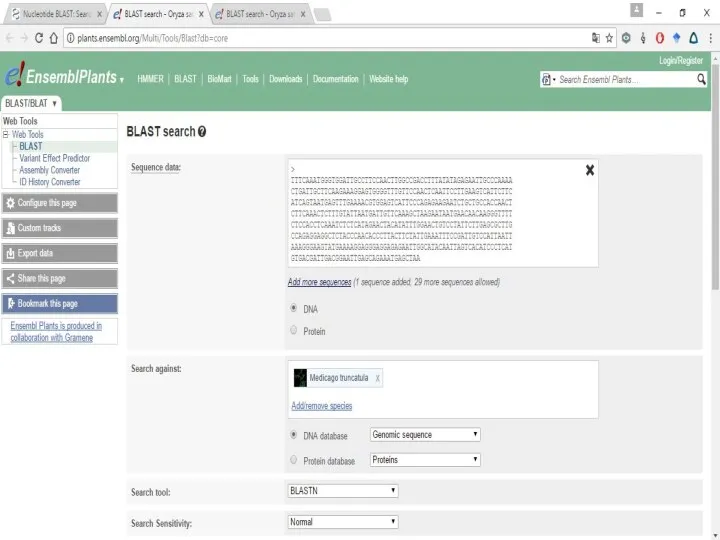
Слайд 16Когда есть «неизвестная» последовательность ДНК
На примере гена NBS-LRR гена детекции патогенов у

люцерны (давайте сделаем вид, что сиквенс действительно случайный)
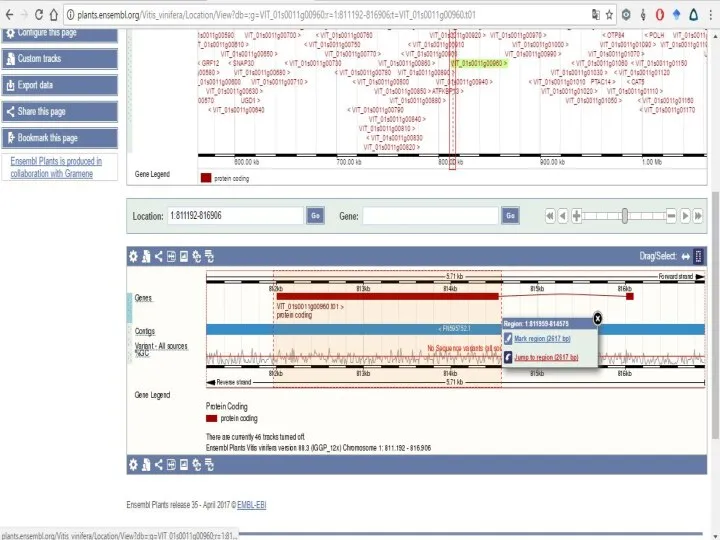
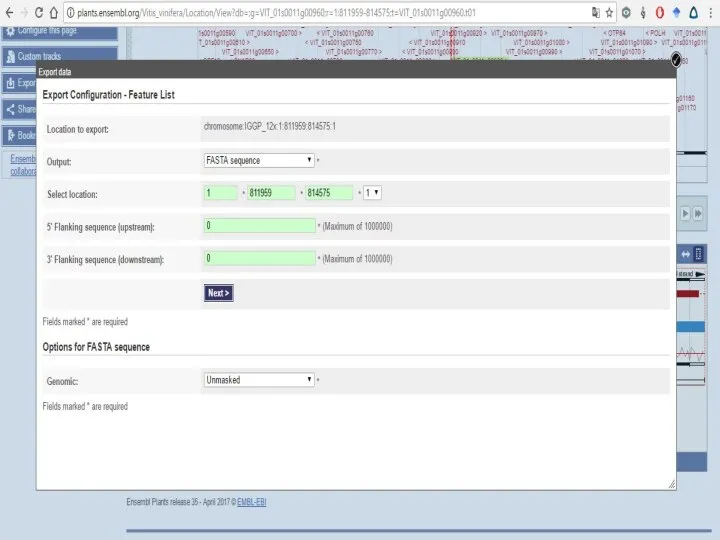
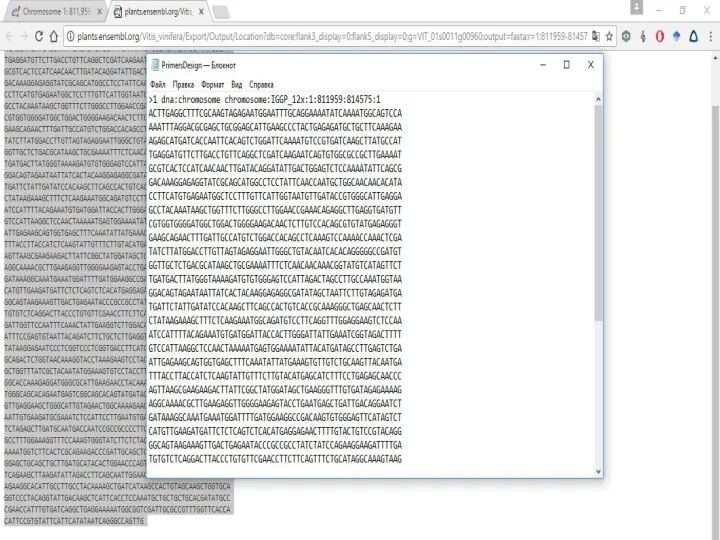
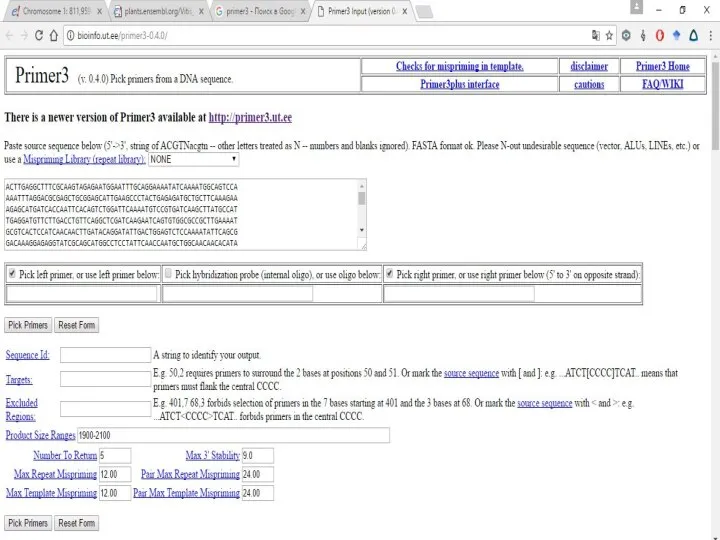
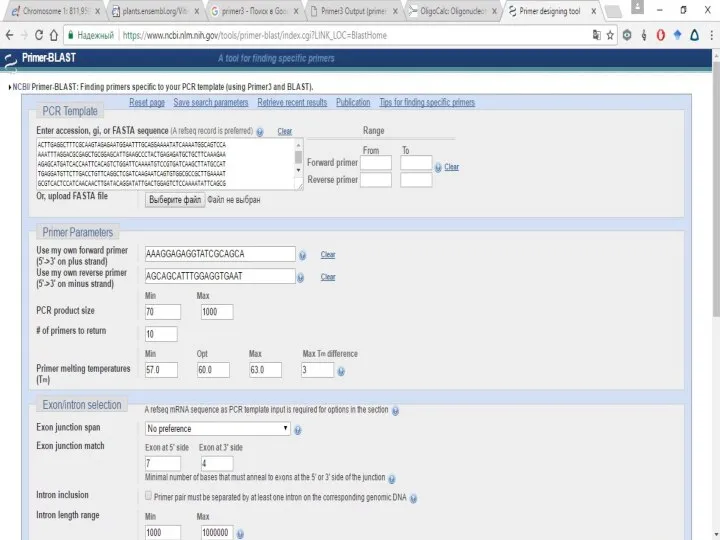
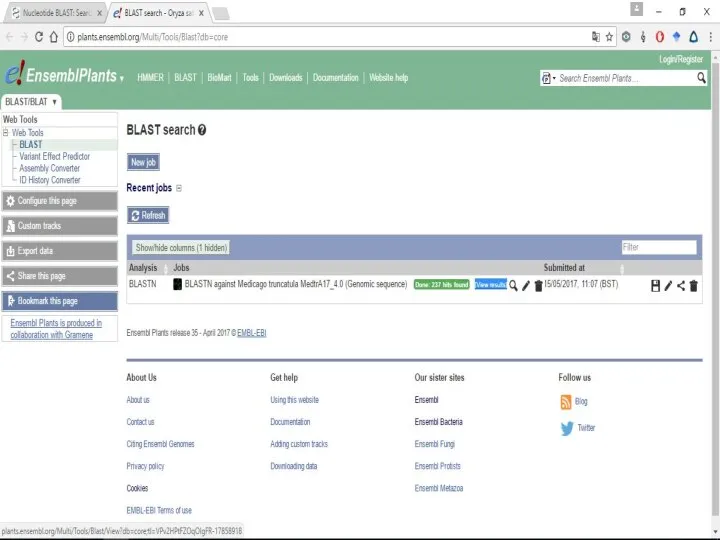
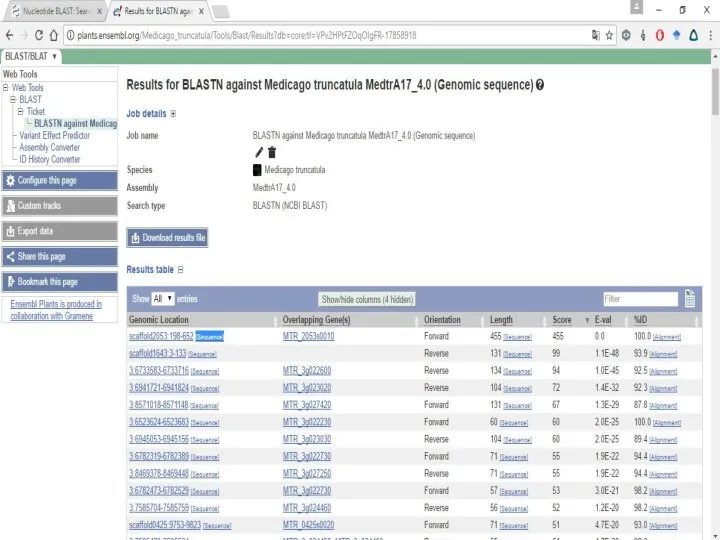
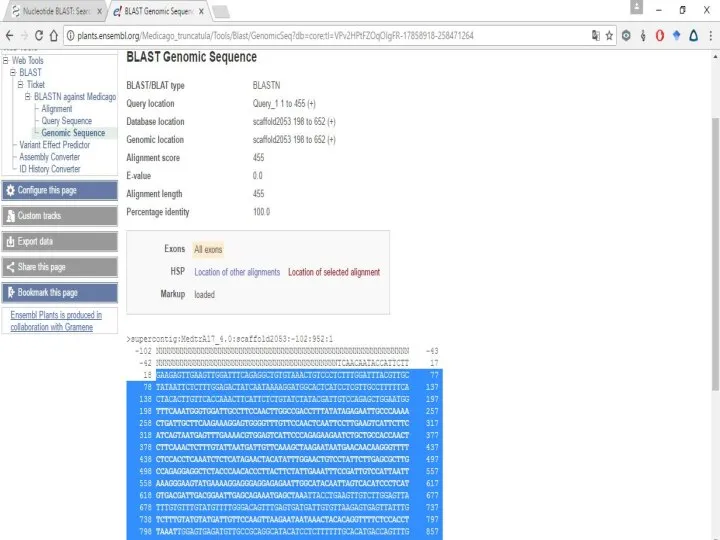
Слайд 21Далее возвращаемся к разделу Primer3 и делаем как делали ранее.
То есть все

то же самое, только если бы у Вас не было названия гена, а был какой-то его сиквенс.
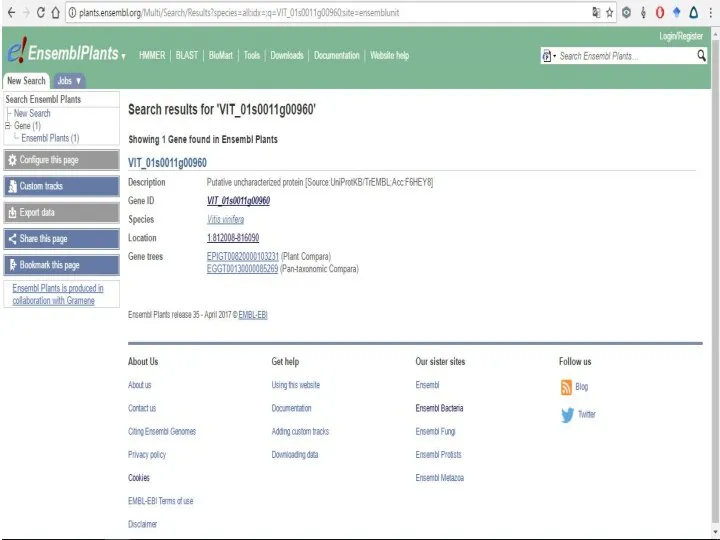
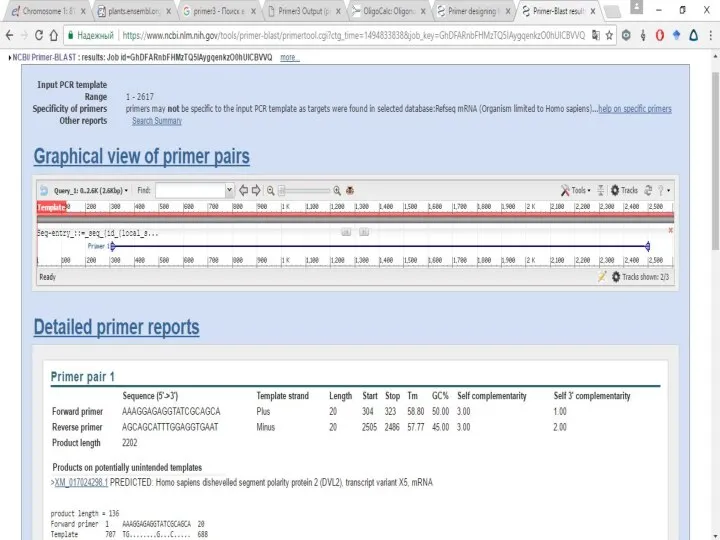
Слайд 22Но. Если есть название гена, который пересекается с нашим сиквенсом, то можно
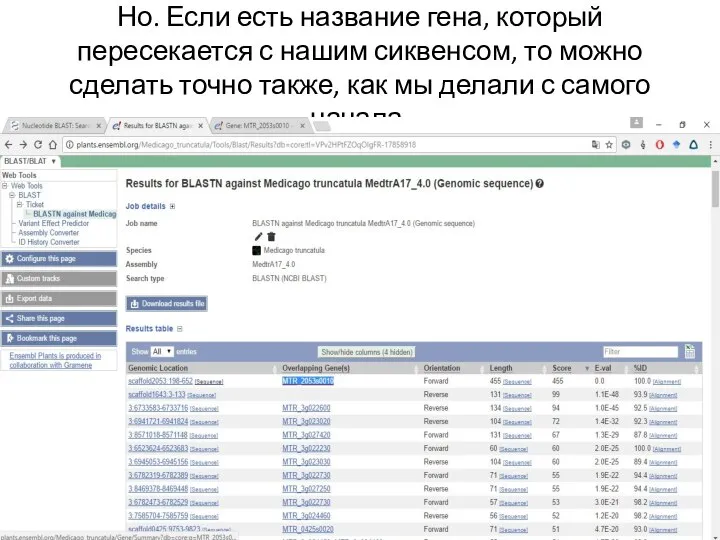
сделать точно также, как мы делали с самого начала.






 Пятнистый леопардовый эублефар (Eublepharis macularius)
Пятнистый леопардовый эублефар (Eublepharis macularius) Свиристель. 4 класс
Свиристель. 4 класс Презентация на тему Скаты
Презентация на тему Скаты  Клетка растения. Ткань растения
Клетка растения. Ткань растения Изучаем растения
Изучаем растения Образование. Фотосинтез. Симметрия. Хиральность
Образование. Фотосинтез. Симметрия. Хиральность Домашние животные
Домашние животные Зимние птицы
Зимние птицы Презентация на тему "Ткани человека" - презентации по Биологии
Презентация на тему "Ткани человека" - презентации по Биологии Что такое цветок?
Что такое цветок? 2. ТЗ 4 Вирусы. Бактериофаги
2. ТЗ 4 Вирусы. Бактериофаги Об амфибиях и рептилиях, которых многие совсем не любят. Земноводные и пресмыкающиеся Спасского района Нижегородской области
Об амфибиях и рептилиях, которых многие совсем не любят. Земноводные и пресмыкающиеся Спасского района Нижегородской области Царство грибов
Царство грибов Ребусы о растениях
Ребусы о растениях Ткани человека
Ткани человека Живая и неживая природа
Живая и неживая природа Митоз и мейоз
Митоз и мейоз Біологічні основи запліднення та розвитку організму
Біологічні основи запліднення та розвитку організму Белки: строение и функции. Лекция 2
Белки: строение и функции. Лекция 2 Комнатные растения в интерьере квартиры
Комнатные растения в интерьере квартиры Растения России
Растения России Молекулярная биология. Размножения растений. Лекция 1. Введение + методы
Молекулярная биология. Размножения растений. Лекция 1. Введение + методы osnovy_genetikiPrezentatsia_Microsoft_PowerPoint_2 (1)
osnovy_genetikiPrezentatsia_Microsoft_PowerPoint_2 (1) Пищеварение в ротовой полости и в желудке
Пищеварение в ротовой полости и в желудке В гостях у дикой природы. Лоси
В гостях у дикой природы. Лоси Однодольные и Двудольные растения
Однодольные и Двудольные растения Класс Земноводные
Класс Земноводные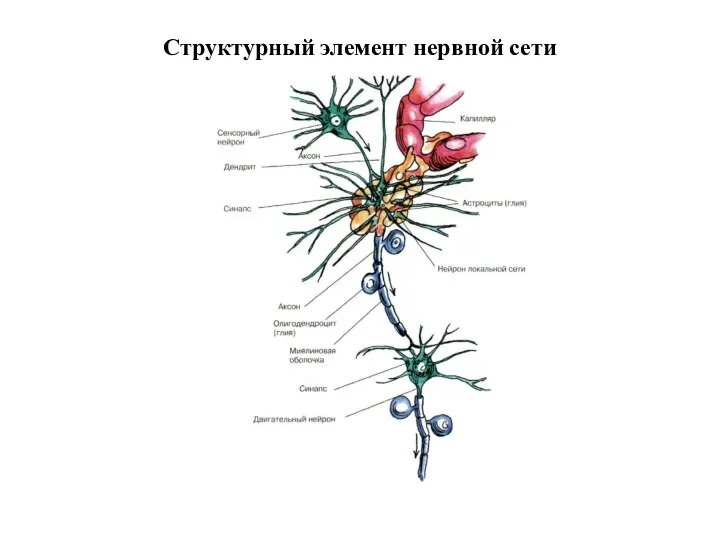 Структурный элемент нервной сети
Структурный элемент нервной сети