Содержание
- 2. Некоторые параметры молекул ДНК и белка: Один шаг это полный виток спирали ДНК-поворот на 3600 Один
- 3. Выравнивание генетических последовательностей В эволюции генетических последовательностей происходят как замены, так и вставки и делеции. Первым
- 4. Выравнивание генетических последовательностей
- 5. Выравнивание генетических последовательностей
- 6. Выравнивание генетических последовательностей Clustal -- это одна из самых широко используемых компьютерных программ для множественного выравнивания
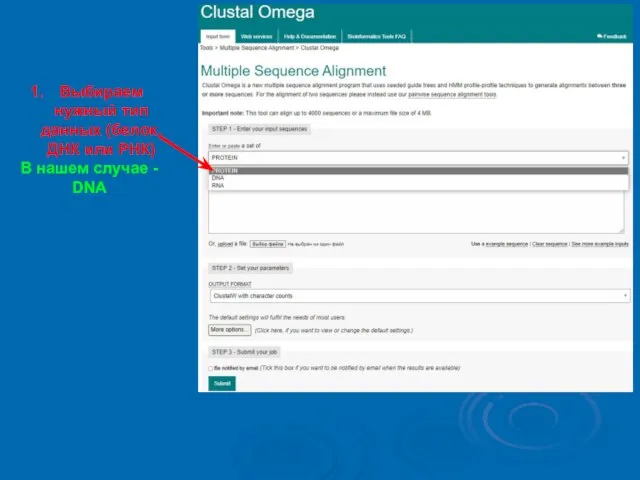
- 7. Выбираем нужный тип данных (белок, ДНК или РНК) В нашем случае - DNA
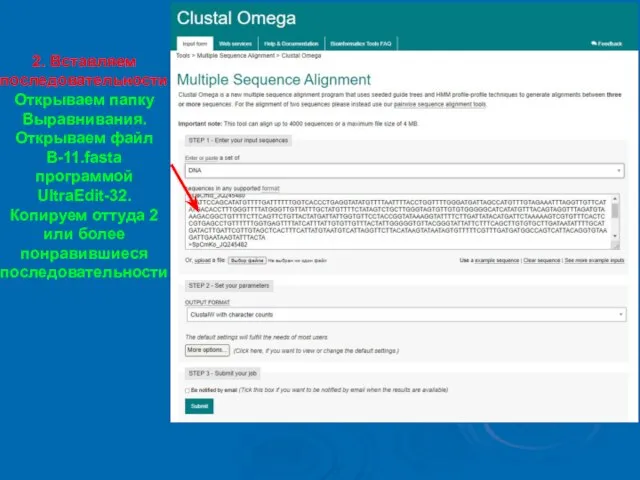
- 8. 2. Вставляем последовательности Открываем папку Выравнивания. Открываем файл В-11.fasta программой UltraEdit-32. Копируем оттуда 2 или более
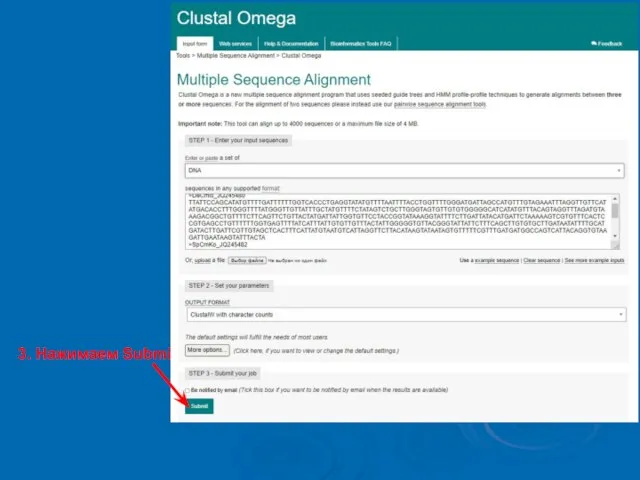
- 9. 3. Нажимаем Submit
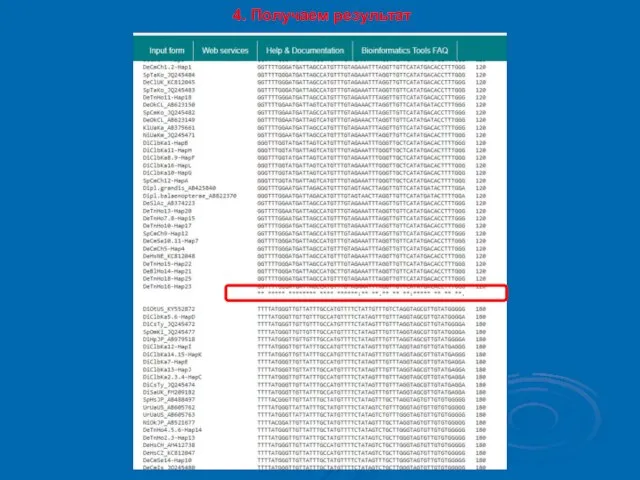
- 10. 4. Получаем результат
- 11. 5. Интерпретация Звездочка (*) – различия по данной позиции (нуклеотид или аминокислота) отсутствуют между разными последовательностями
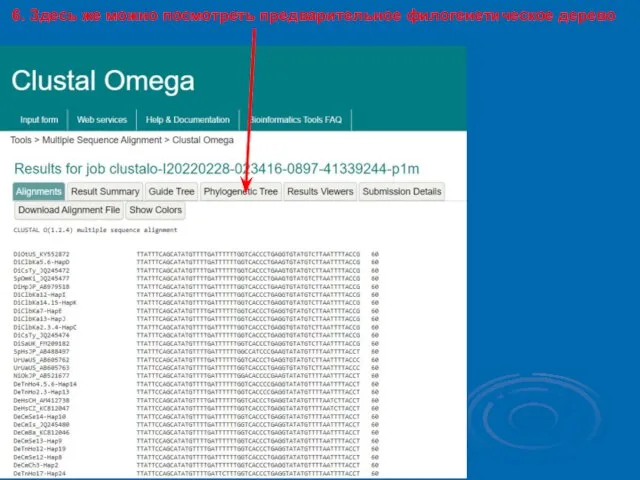
- 12. 6. Здесь же можно посмотреть предварительное филогенетическое дерево
- 14. BLAST BLAST (англ. Basic Local Alignment Search Tool — средство поиска основного локального выравнивания) — семейство
- 15. Классификация программ серии BLAST Нуклеотидные предназначены для сравнения изучаемой нуклеотидной последовательности с базой данных секвенированных нуклеиновых
- 16. Классификация программ серии BLAST Белковые предназначены для сравнения изучаемой аминокислотной последовательности белка с имеющейся базой данных
- 17. Классификация программ серии BLAST Транслирующие способны транслировать нуклеотидные последовательности в аминокислотные: blastx — переводит изучаемую нуклеотидную
- 18. Классификация программ серии BLAST Геномные предназначены для сравнения изучаемой нуклеотидной последовательности с базой данных секвенированного генома
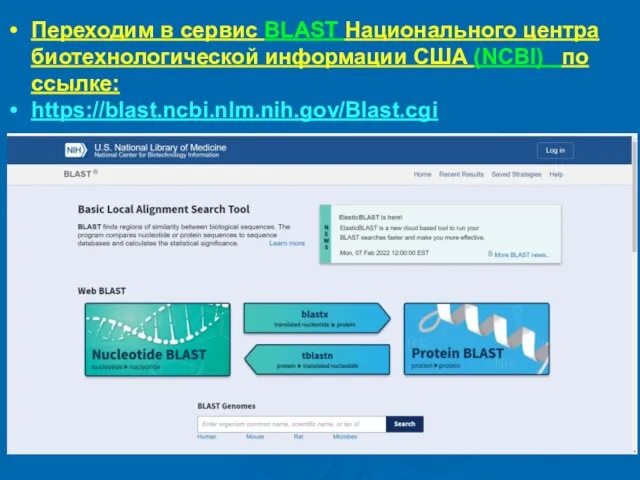
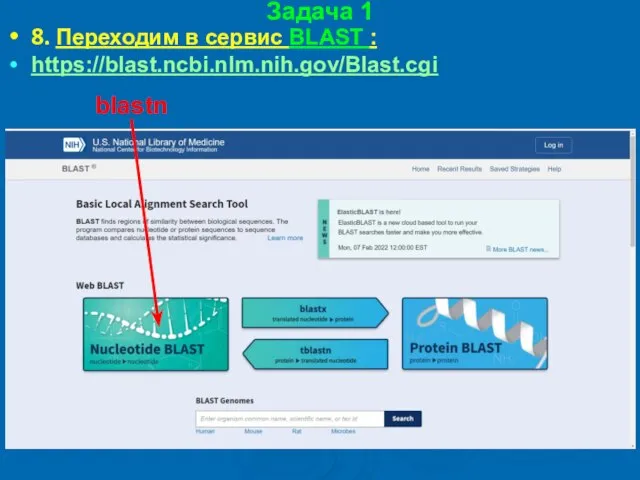
- 19. Переходим в сервис BLAST Национального центра биотехнологической информации США (NCBI) по ссылке: https://blast.ncbi.nlm.nih.gov/Blast.cgi
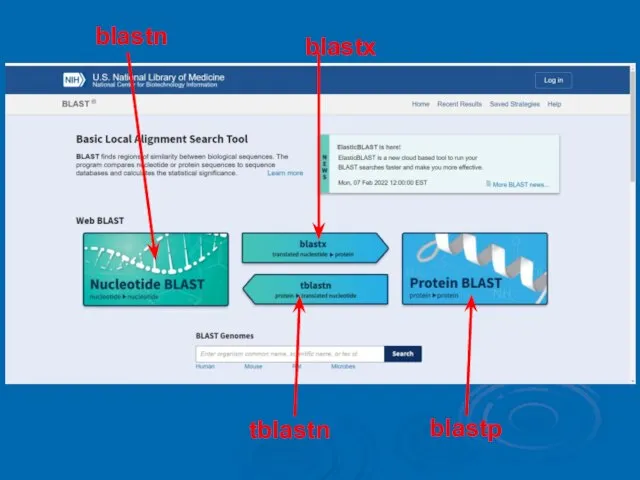
- 20. blastn tblastn blastx blastp
- 21. Задача 1. Форма отчета Каждый лично на своем компьютере делает скриншот/фото (так, чтобы было видно номер
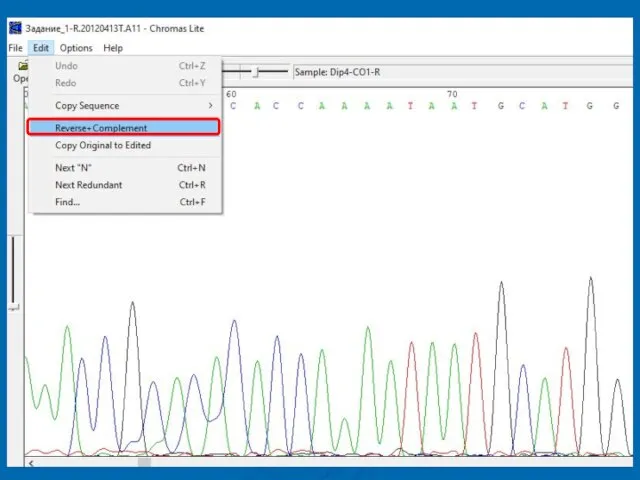
- 22. Задача 1 Открыть с помощью программы Chromas файл с хроматограммой Задание_1-R.20120413T.A11 из папки Задания Это последовательность
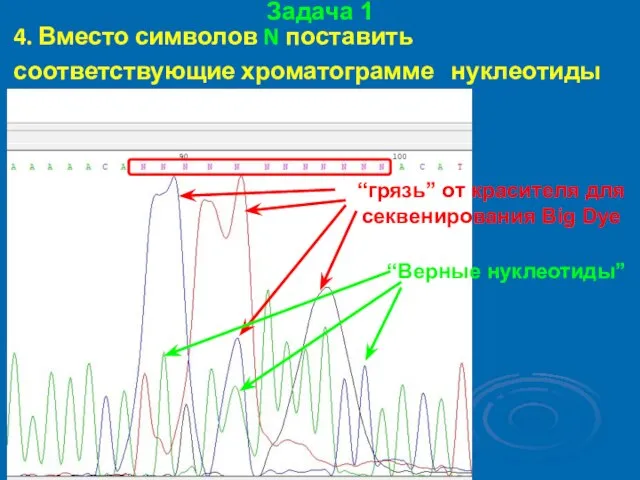
- 24. Задача 1 4. Вместо символов N поставить соответствующие хроматограмме нуклеотиды (A, T, G или С): “грязь”
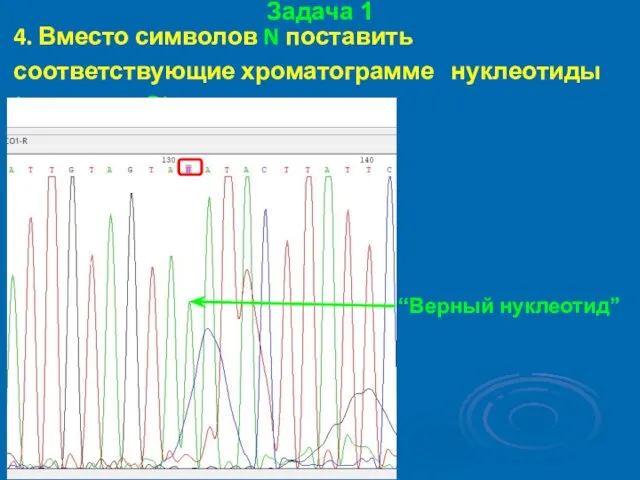
- 25. Задача 1 4. Вместо символов N поставить соответствующие хроматограмме нуклеотиды (A, T, G или С): “Верный
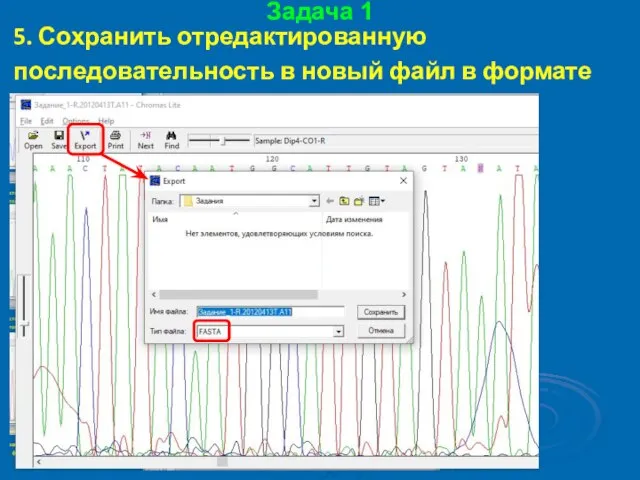
- 26. Задача 1 5. Сохранить отредактированную последовательность в новый файл в формате FASTA:
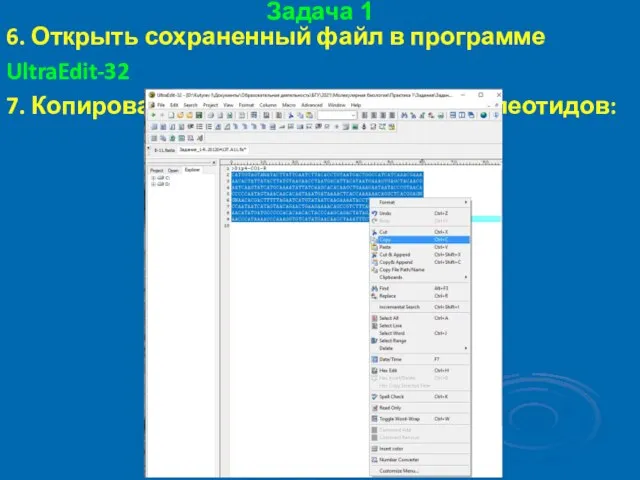
- 27. Задача 1 6. Открыть сохраненный файл в программе UltraEdit-32 7. Копировать последовательность нуклеотидов:
- 28. Задача 1 8. Переходим в сервис BLAST : https://blast.ncbi.nlm.nih.gov/Blast.cgi blastn
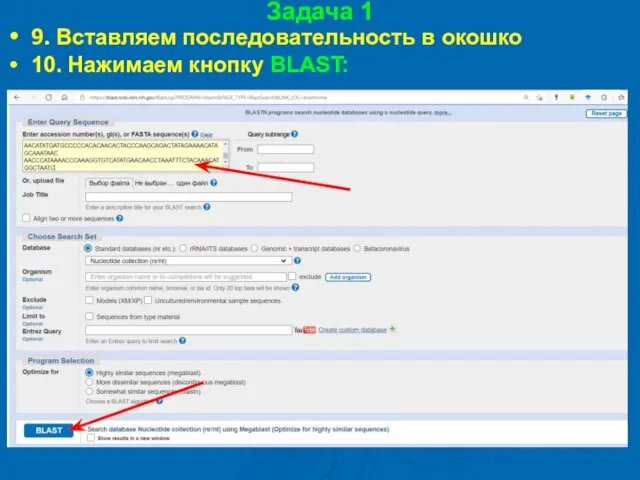
- 29. Задача 1 9. Вставляем последовательность в окошко 10. Нажимаем кнопку BLAST:
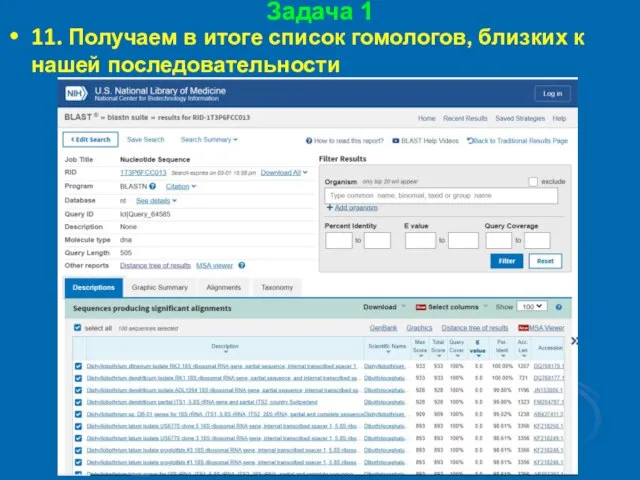
- 30. Задача 1 11. Получаем в итоге список гомологов, близких к нашей последовательности
- 31. Задача 1. Форма отчета Каждый лично на своем компьютере делает скриншот/фото (так, чтобы было видно номер
- 32. Задача 2. Форма отчета Каждый лично отправляет мне на почту [email protected] : 1) файл insulin.fasta 2)
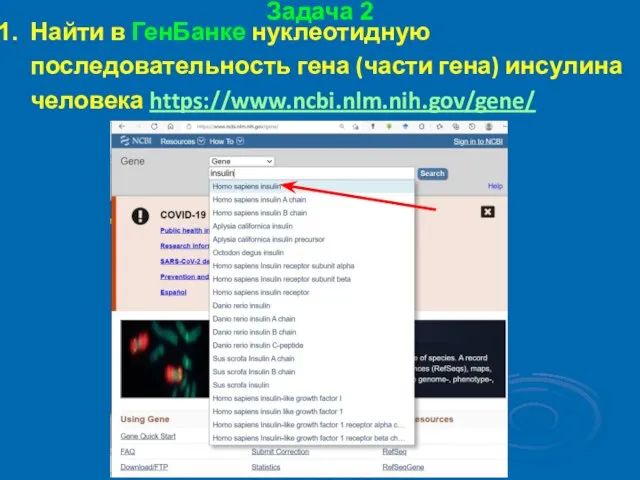
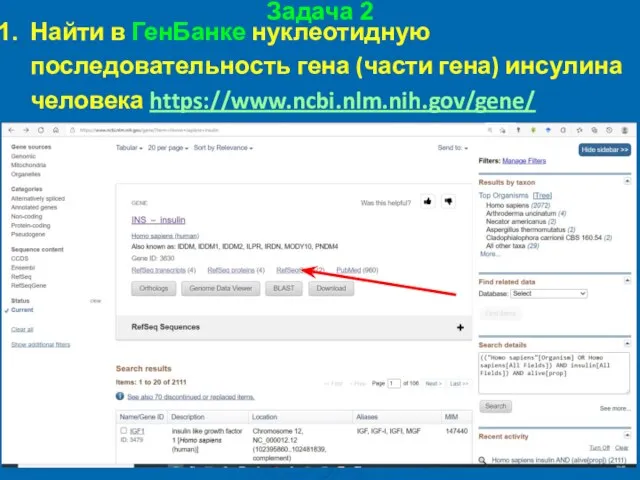
- 33. Задача 2 Найти в ГенБанке нуклеотидную последовательность гена (части гена) инсулина человека https://www.ncbi.nlm.nih.gov/gene/
- 34. Задача 2 Найти в ГенБанке нуклеотидную последовательность гена (части гена) инсулина человека https://www.ncbi.nlm.nih.gov/gene/
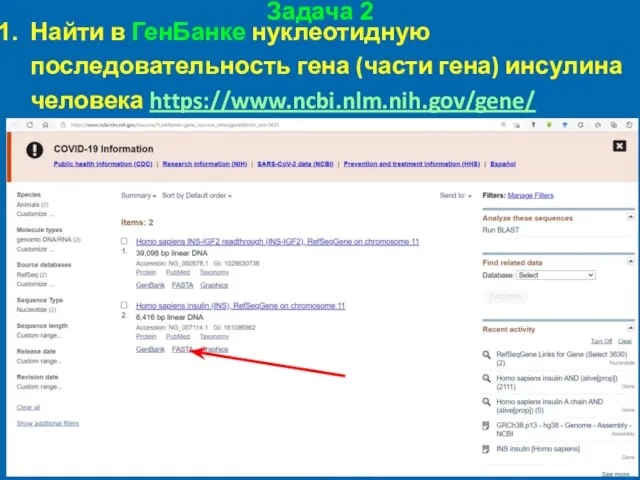
- 35. Задача 2 Найти в ГенБанке нуклеотидную последовательность гена (части гена) инсулина человека https://www.ncbi.nlm.nih.gov/gene/
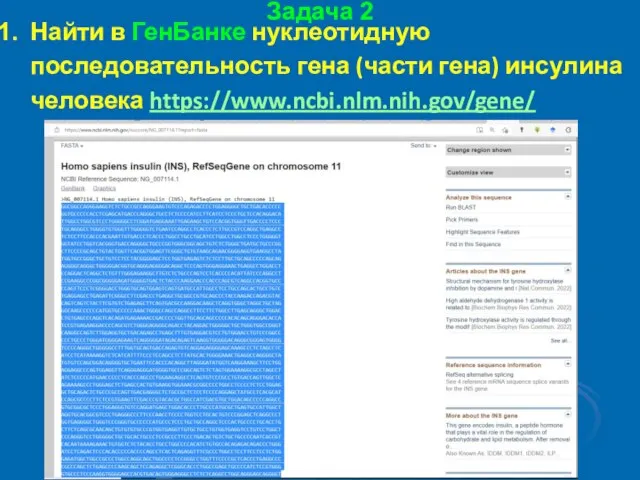
- 36. Задача 2 Найти в ГенБанке нуклеотидную последовательность гена (части гена) инсулина человека https://www.ncbi.nlm.nih.gov/gene/
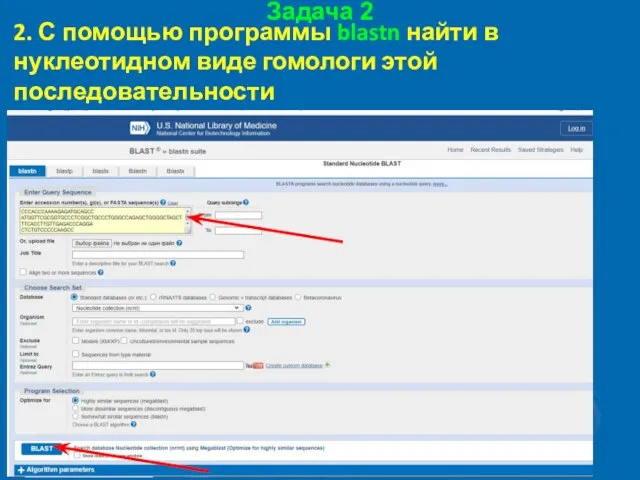
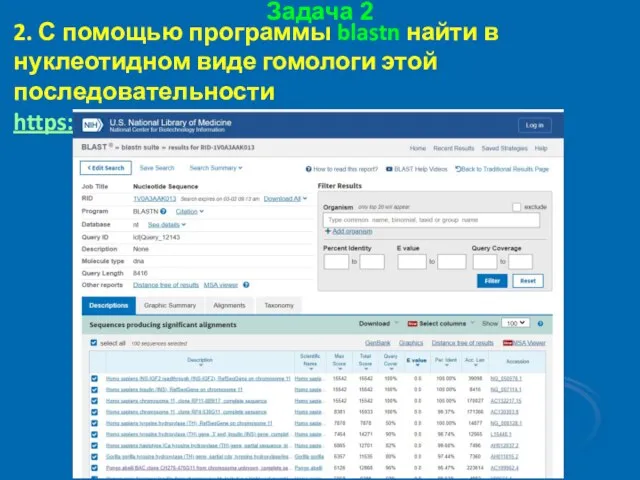
- 37. Задача 2 2. С помощью программы blastn найти в нуклеотидном виде гомологи этой последовательности https://blast.ncbi.nlm.nih.gov/Blast.cgi
- 38. Задача 2 2. С помощью программы blastn найти в нуклеотидном виде гомологи этой последовательности https://blast.ncbi.nlm.nih.gov/Blast.cgi
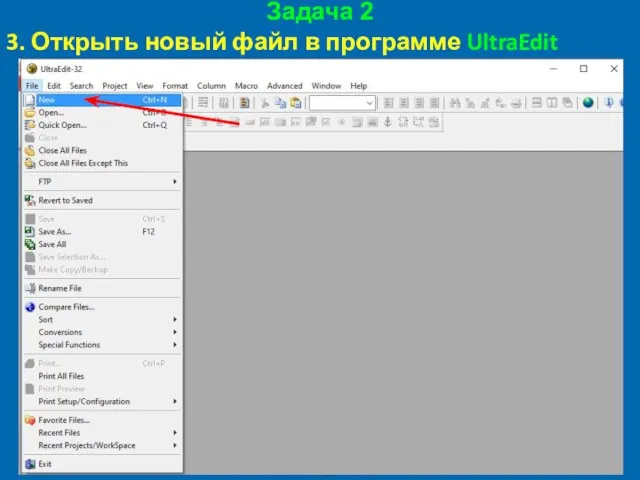
- 39. Задача 2 3. Открыть новый файл в программе UltraEdit
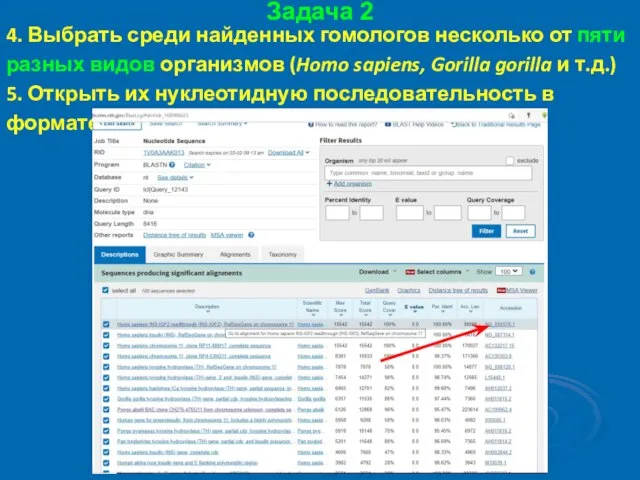
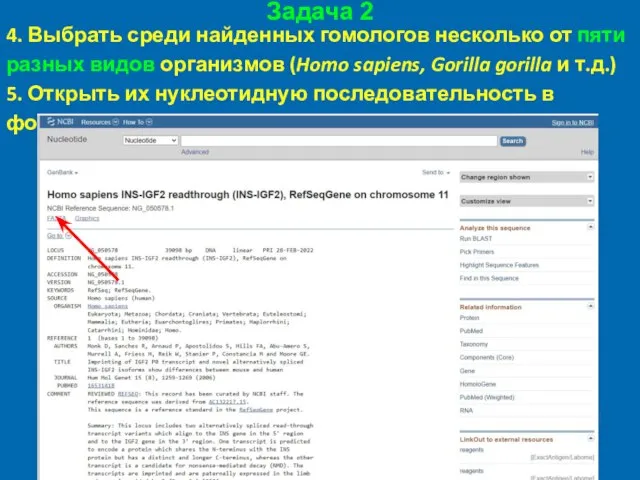
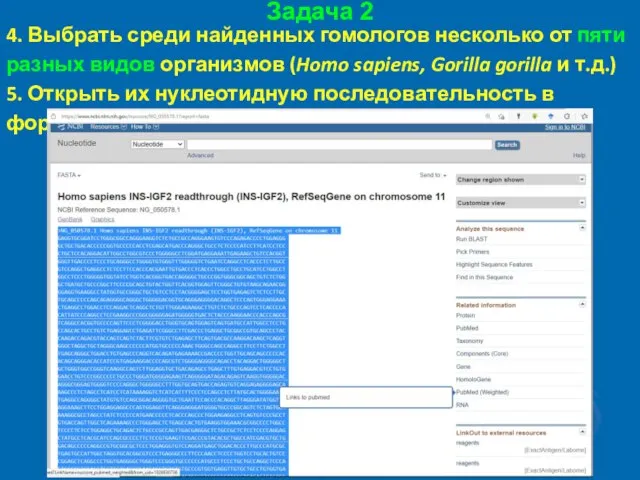
- 40. Задача 2 4. Выбрать среди найденных гомологов несколько от пяти разных видов организмов (Homo sapiens, Gorilla
- 41. Задача 2 4. Выбрать среди найденных гомологов несколько от пяти разных видов организмов (Homo sapiens, Gorilla
- 42. Задача 2 4. Выбрать среди найденных гомологов несколько от пяти разных видов организмов (Homo sapiens, Gorilla
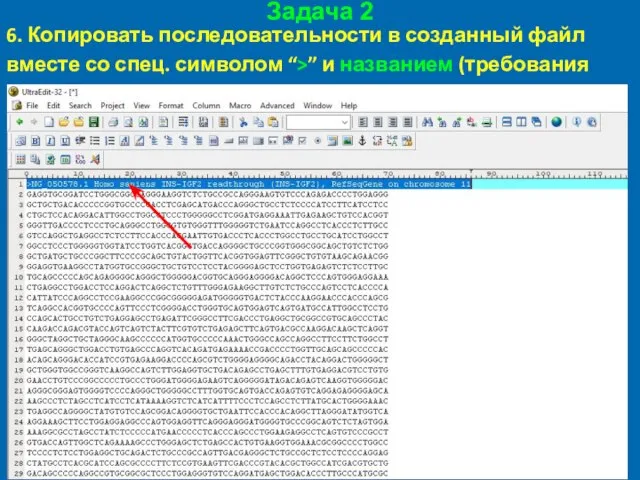
- 43. Задача 2 6. Копировать последовательности в созданный файл вместе со спец. символом “>” и названием (требования
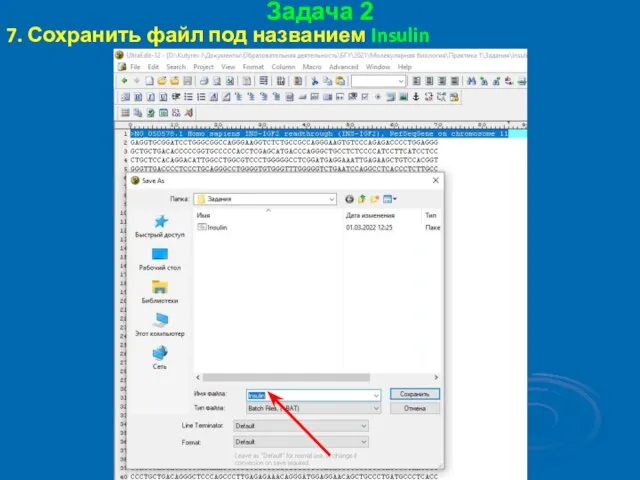
- 44. Задача 2 7. Сохранить файл под названием Insulin
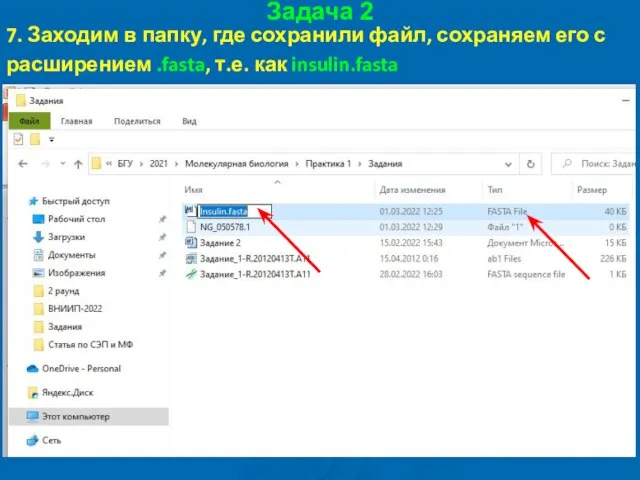
- 45. Задача 2 7. Заходим в папку, где сохранили файл, сохраняем его с расширением .fasta, т.е. как
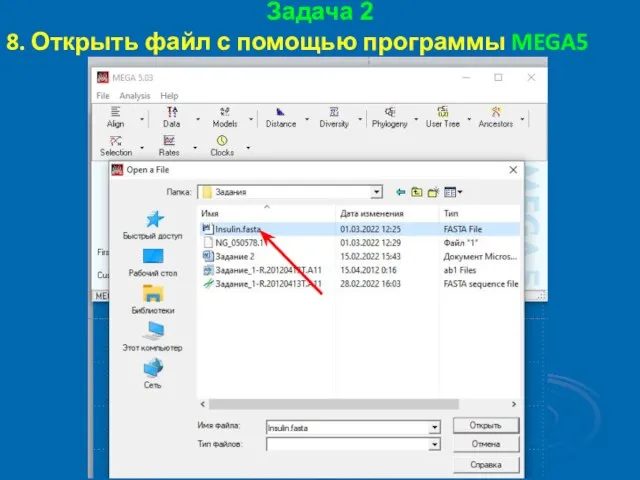
- 46. Задача 2 8. Открыть файл с помощью программы MEGA5
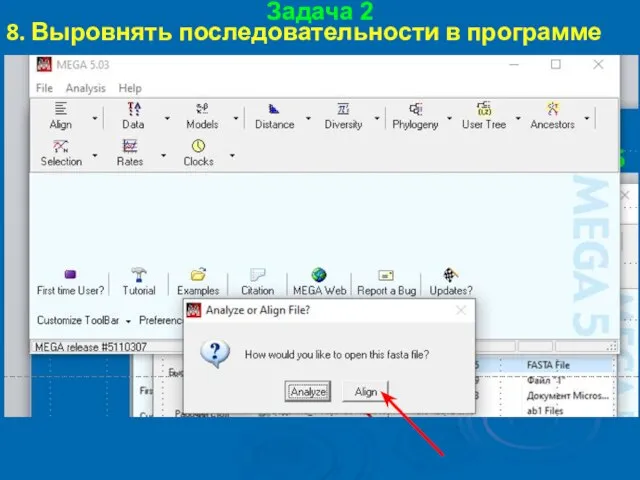
- 47. Задача 2 8. Выровнять последовательности в программе MEGA5
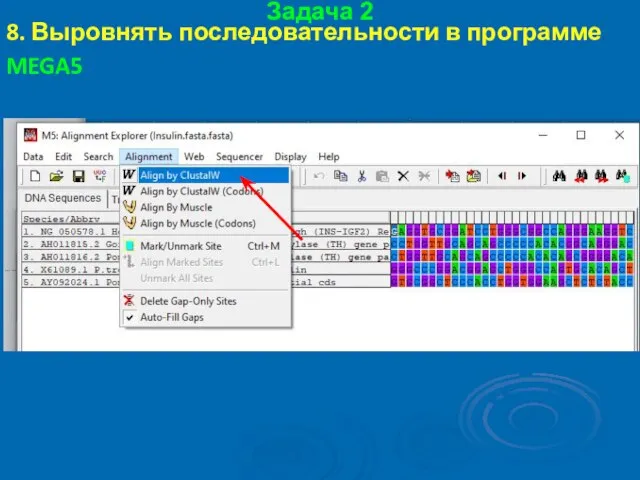
- 48. Задача 2 8. Выровнять последовательности в программе MEGA5
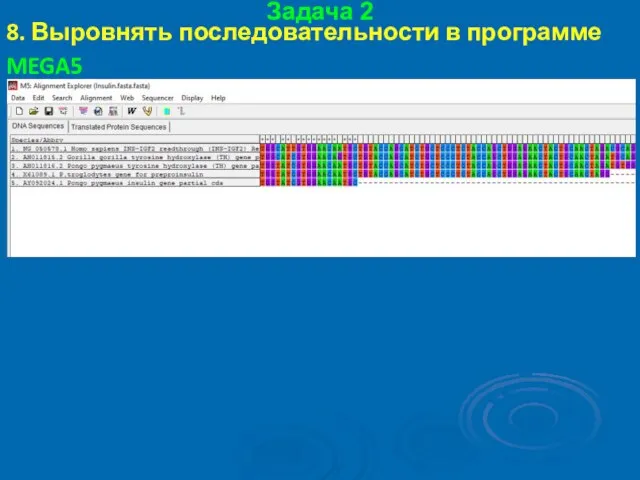
- 49. Задача 2 8. Выровнять последовательности в программе MEGA5
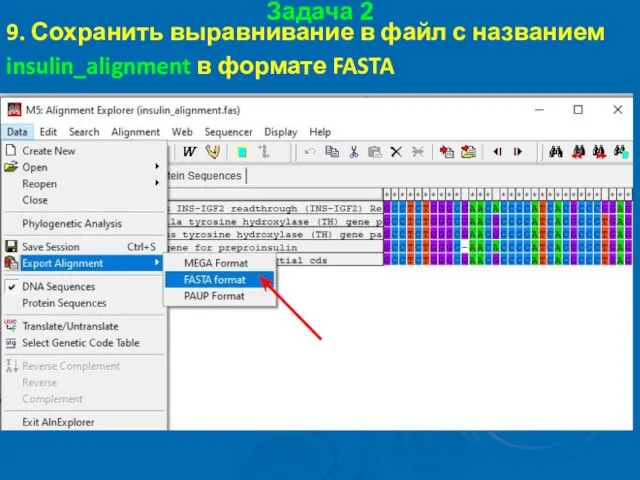
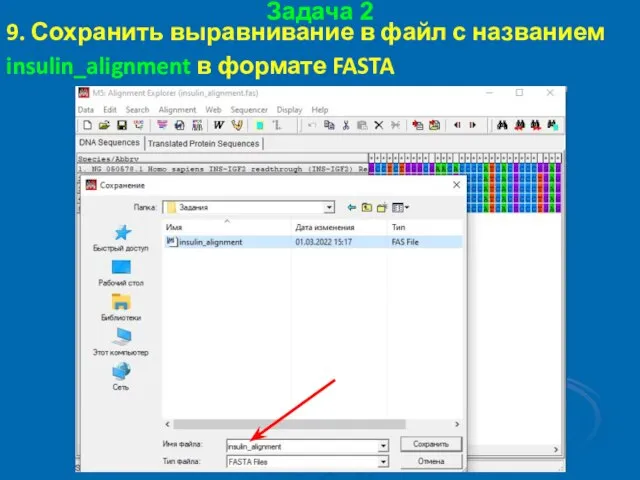
- 50. Задача 2 9. Сохранить выравнивание в файл с названием insulin_alignment в формате FASTA
- 51. Задача 2 9. Сохранить выравнивание в файл с названием insulin_alignment в формате FASTA
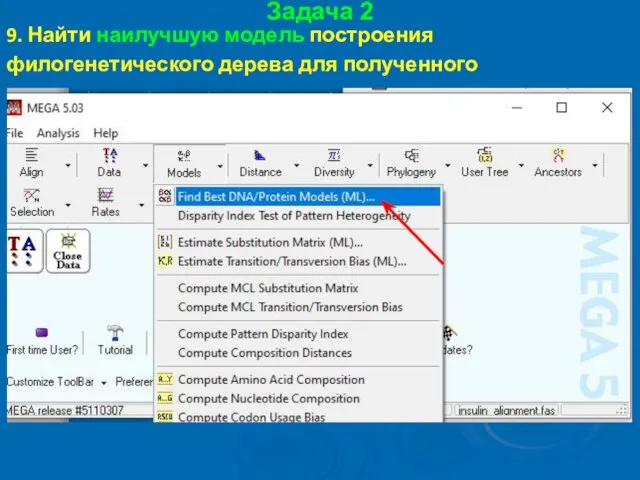
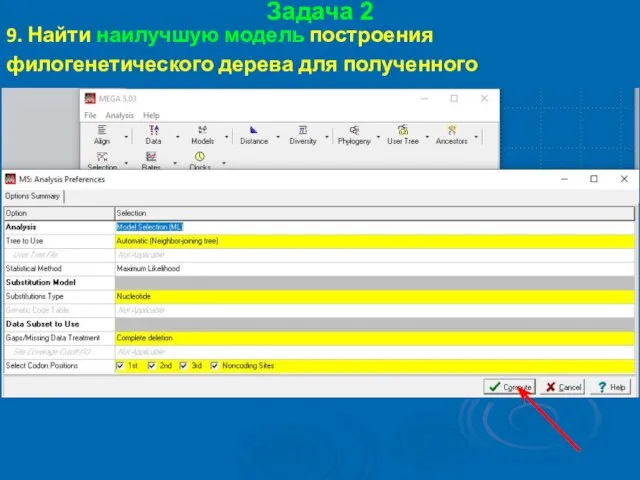
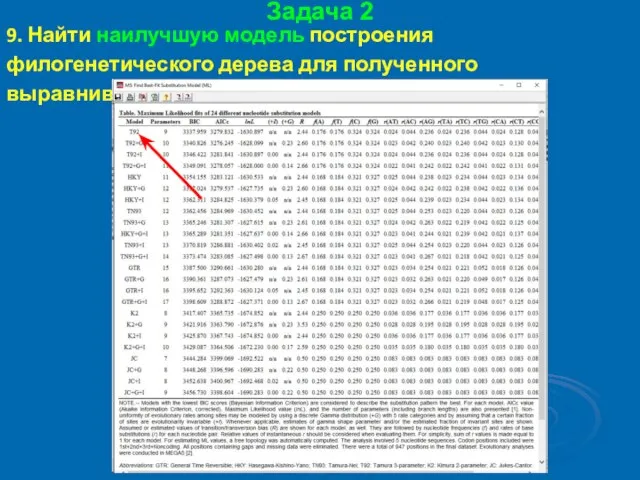
- 52. Задача 2 9. Найти наилучшую модель построения филогенетического дерева для полученного выравнивания
- 53. Задача 2 9. Найти наилучшую модель построения филогенетического дерева для полученного выравнивания
- 54. Задача 2 9. Найти наилучшую модель построения филогенетического дерева для полученного выравнивания
- 55. Задача 2 10. Смотрим расшифровку наилучшей модели внизу таблицы, необходимую для построения филогенетического дерева:
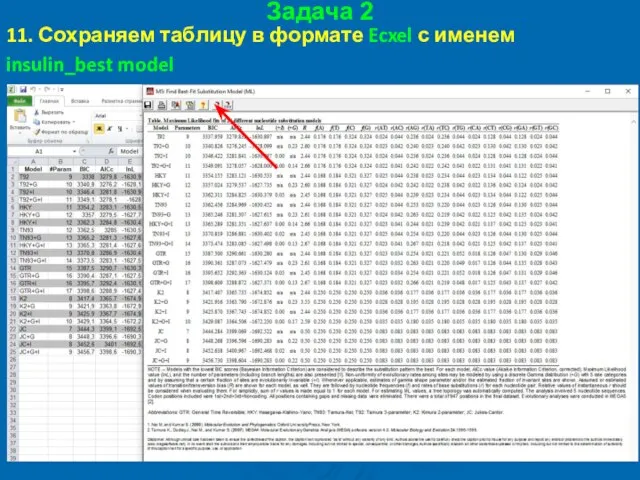
- 56. Задача 2 11. Сохраняем таблицу в формате Ecxel с именем insulin_best model
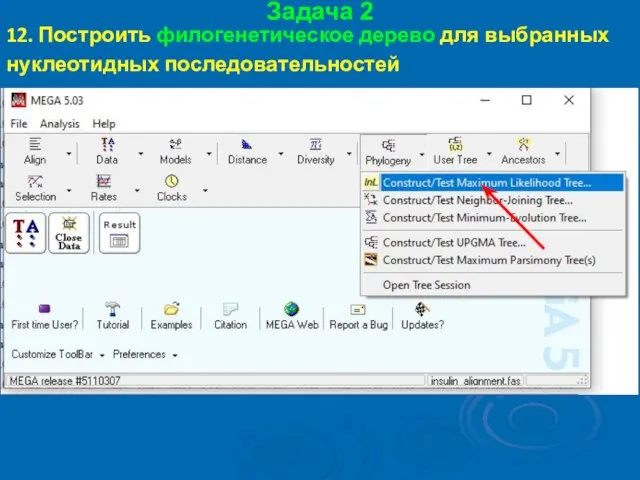
- 57. Задача 2 12. Построить филогенетическое дерево для выбранных нуклеотидных последовательностей
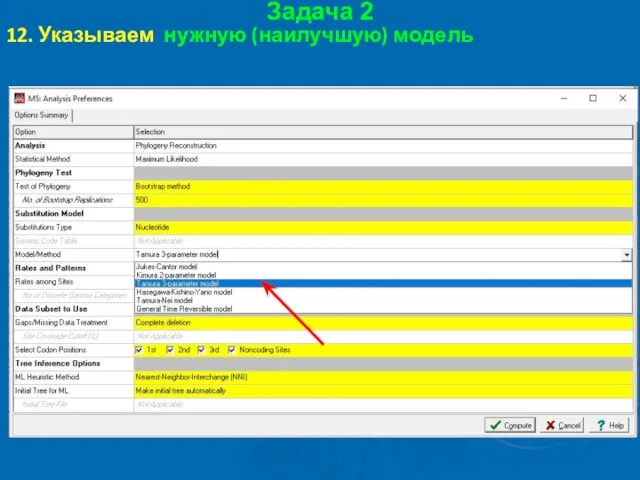
- 58. Задача 2 12. Указываем нужную (наилучшую) модель
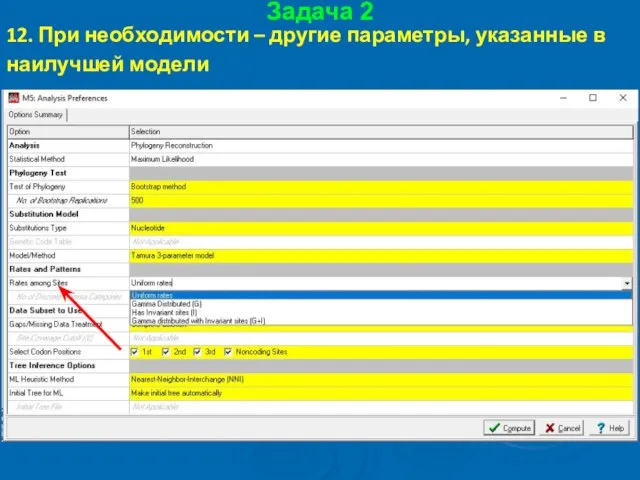
- 59. Задача 2 12. При необходимости – другие параметры, указанные в наилучшей модели
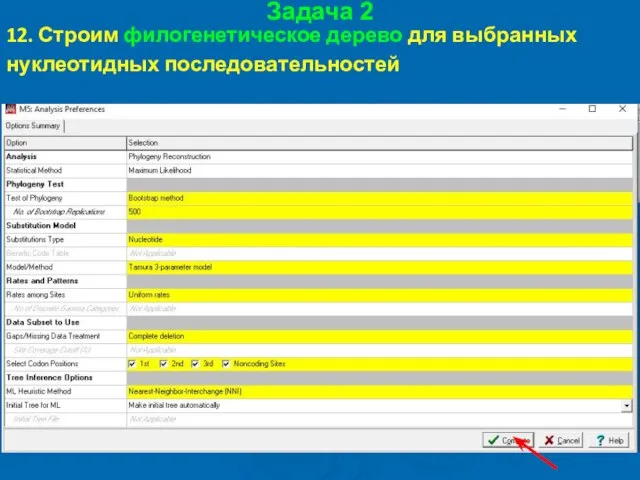
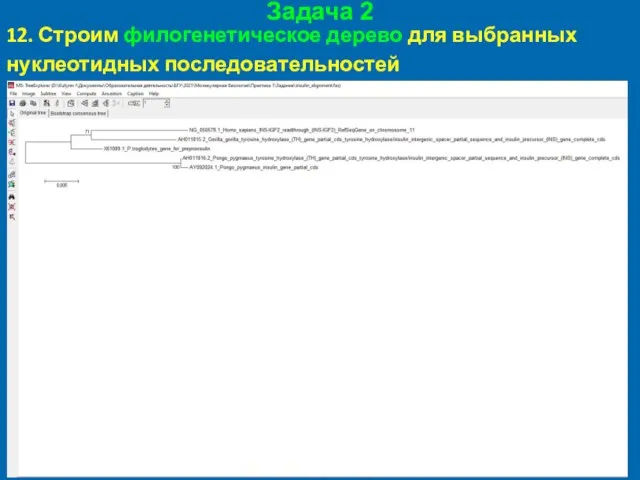
- 60. Задача 2 12. Строим филогенетическое дерево для выбранных нуклеотидных последовательностей
- 61. Задача 2 12. Строим филогенетическое дерево для выбранных нуклеотидных последовательностей
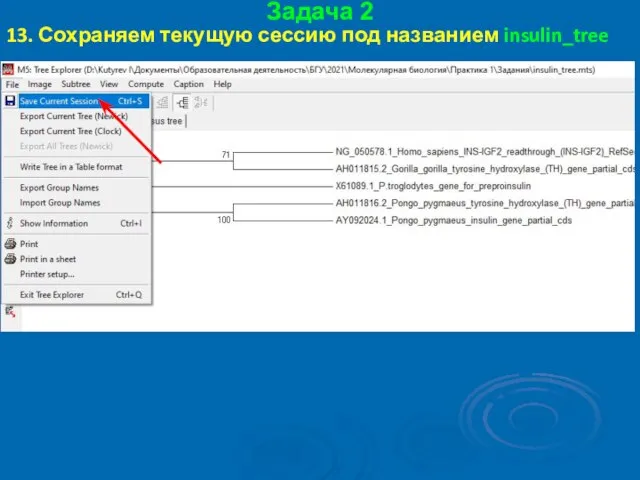
- 62. Задача 2 13. Сохраняем текущую сессию под названием insulin_tree
- 64. Скачать презентацию

















 Генетика тарауын кайталау
Генетика тарауын кайталау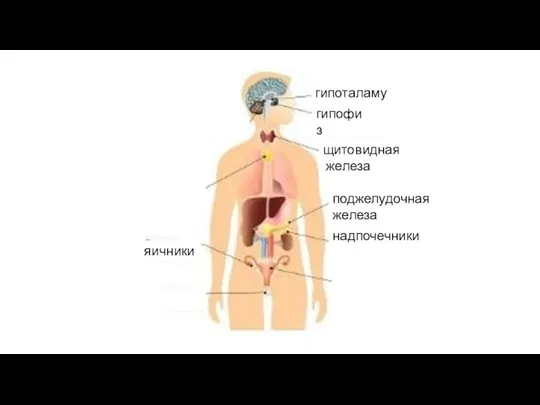 Гормональная регуляция
Гормональная регуляция Великие естествоиспытатели
Великие естествоиспытатели Метаболизм и питание микроорганизмов
Метаболизм и питание микроорганизмов Клиникалық диагностикалық зерттеу-биологиялық субстраттардың физико-химиялық қасиеттерін анықтайды
Клиникалық диагностикалық зерттеу-биологиялық субстраттардың физико-химиялық қасиеттерін анықтайды Эндокринная система
Эндокринная система Предмет и задачи микробиологии. История развития микробиологии
Предмет и задачи микробиологии. История развития микробиологии Методы регистрации клеточного цикла
Методы регистрации клеточного цикла Птицы. Картинки
Птицы. Картинки Теория эндосимбиоза
Теория эндосимбиоза Виды корней и типы корневых систем
Виды корней и типы корневых систем Самонесовместимость. Лекция 8
Самонесовместимость. Лекция 8 Характеристика грибов. Питание грибов
Характеристика грибов. Питание грибов День добра. Животные
День добра. Животные Биотехнология - использование живых систем, клеток, организмов для практических нужд человека
Биотехнология - использование живых систем, клеток, организмов для практических нужд человека Лиственные деревья России
Лиственные деревья России Изучение строения растительной и животной клеток под микроскопом Лабораторная работа
Изучение строения растительной и животной клеток под микроскопом Лабораторная работа Мышление – сложная деятельность коры больших полушарий головного мозга
Мышление – сложная деятельность коры больших полушарий головного мозга Плакуча верба-дерево з легенд
Плакуча верба-дерево з легенд Пищеварение. Функции пищеварительного тракта
Пищеварение. Функции пищеварительного тракта Дыхательная система
Дыхательная система Алекс Флемінг
Алекс Флемінг Hayvanlar
Hayvanlar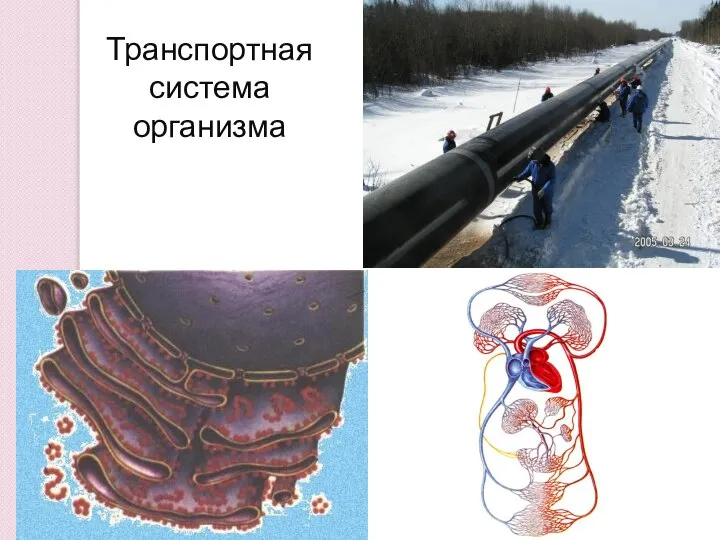 Транспортная система организма
Транспортная система организма Своя игра. Наследственность, изменчивость, клетка (9 класс)
Своя игра. Наследственность, изменчивость, клетка (9 класс) Прекрасный мир цветов
Прекрасный мир цветов Лиса-всему миру краса
Лиса-всему миру краса Половые клетки. Гаметогенез. Урок №63
Половые клетки. Гаметогенез. Урок №63