Содержание
- 2. Подходы к анализу в протеомной масс-спектрометрии I. “Восходящий” анализ – Bottom-up гидролиз белка/белков, масс-спектрометрия малых пептидных
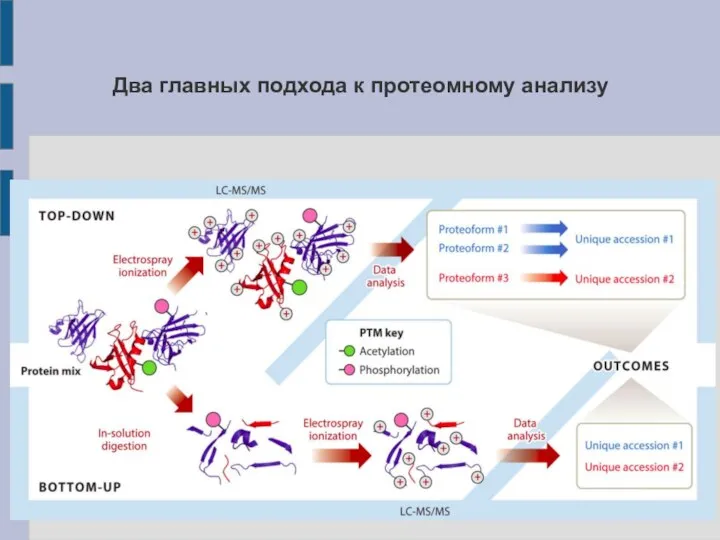
- 3. Два главных подхода к протеомному анализу
- 4. Top-down: изучение интактных белков 1). Позволяет изучать индивидуальные протеины. 2). Является низкопроизводительным подходом. Исключение: изучение белкового
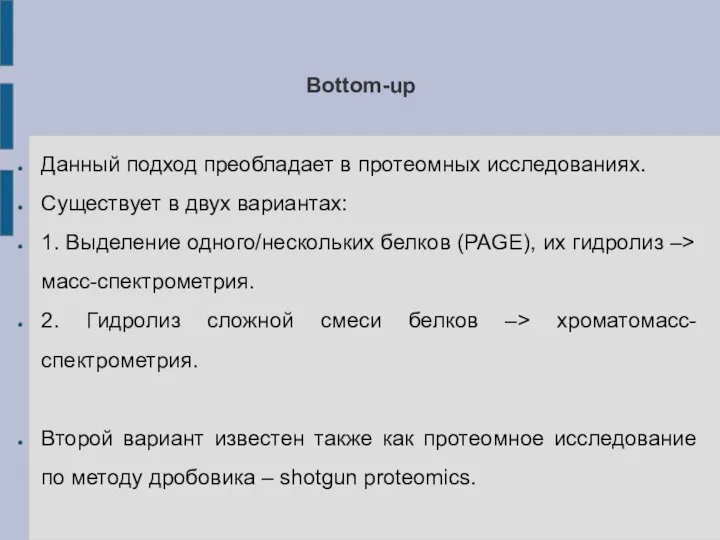
- 5. Bottom-up Данный подход преобладает в протеомных исследованиях. Существует в двух вариантах: 1. Выделение одного/нескольких белков (PAGE),
- 6. Типы протеомного исследования 1. Определение наличия в образце белков с известной последовательностью (“ресеквенирование” белков). 2. Определение
- 7. Определение белков de novo Применяется на основе bottom-up подхода (анализ пептидных спектров). В данном случае требуется
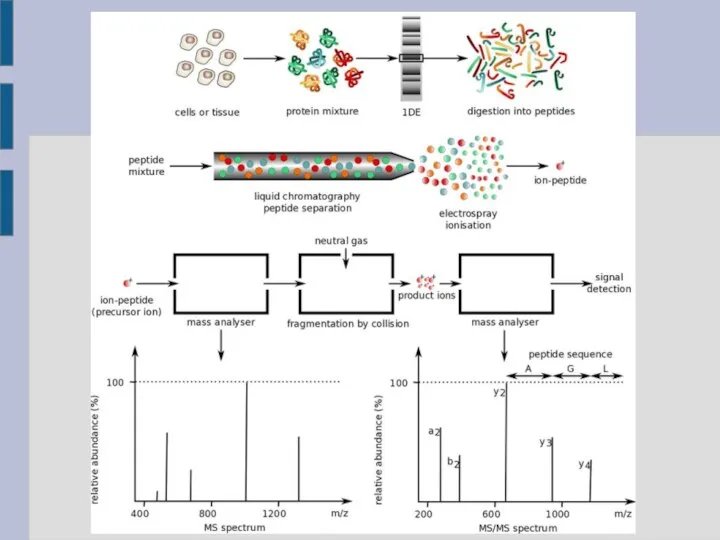
- 9. Основа анализа масс-спектров – определение типов образующихся ионов В подавляющем большинстве методов МС в результате ионизации
- 10. Главные типы ионов, образующихся в протеомной МС Существует три вида связей в пептидах, по которым происходит
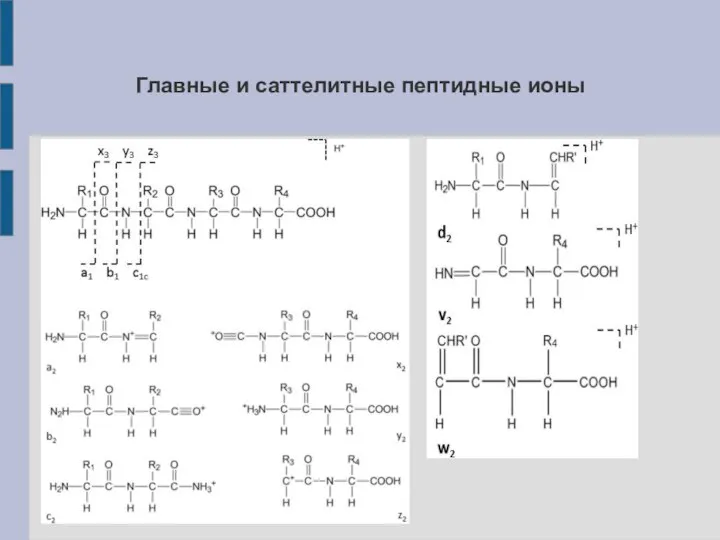
- 11. Дополнительные пептидные ионы При более жёсткой ионизации наряду с главными ионами происходит образование саттелитных ионов (d-,
- 12. Главные и саттелитные пептидные ионы
- 13. Связь между массой главного иона и массами аминокислотных остатков иона Масса образующихся главных ионов определяется суммой
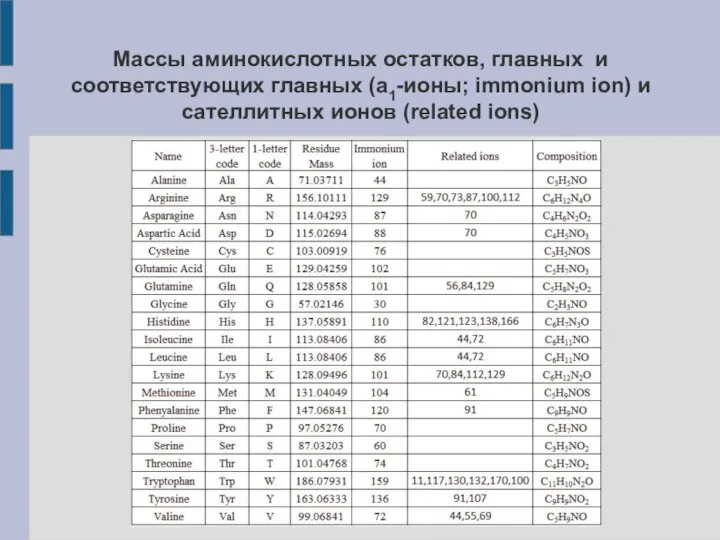
- 14. Массы аминокислотных остатков, главных и соответствующих главных (a1-ионы; immonium ion) и сателлитных ионов (related ions)
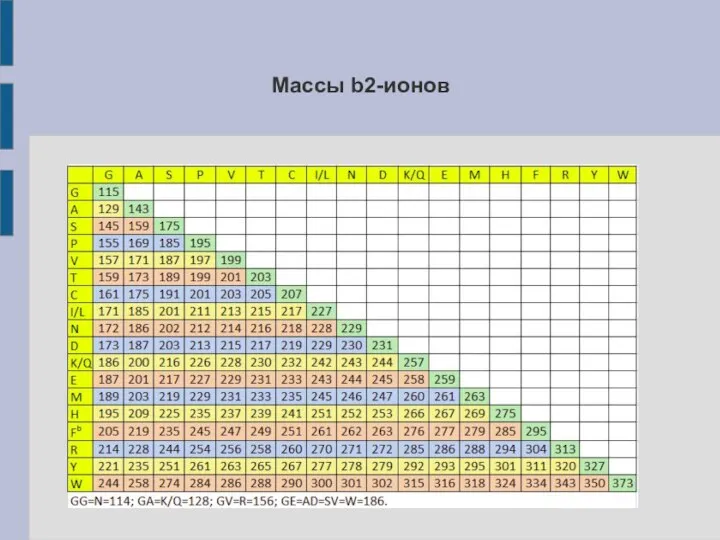
- 15. Массы b2-ионов
- 16. “Правила” ионизации пептидов 1). Подавляющее большинство ионов приходится на b-, y- и a-ионы. а-Ионы образуются из
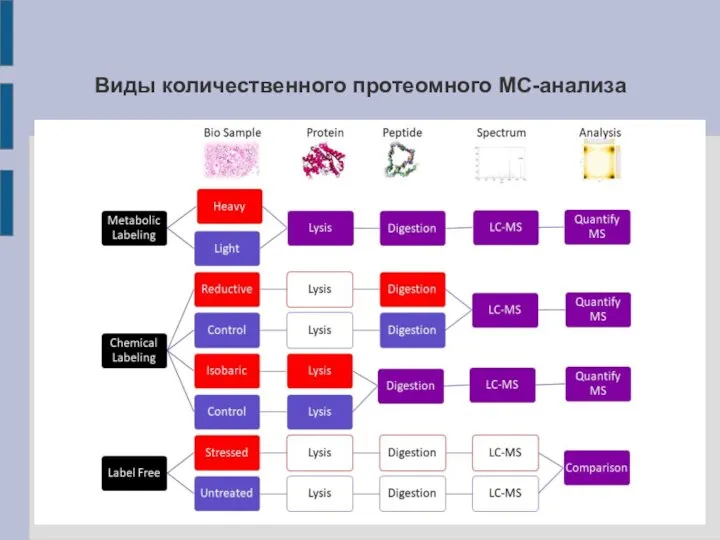
- 17. Количественный протеомный анализ Позволяет оценивать относительные количества одинаковых белков их разных источников в результате анализа смешанного
- 18. Виды количественного протеомного МС-анализа
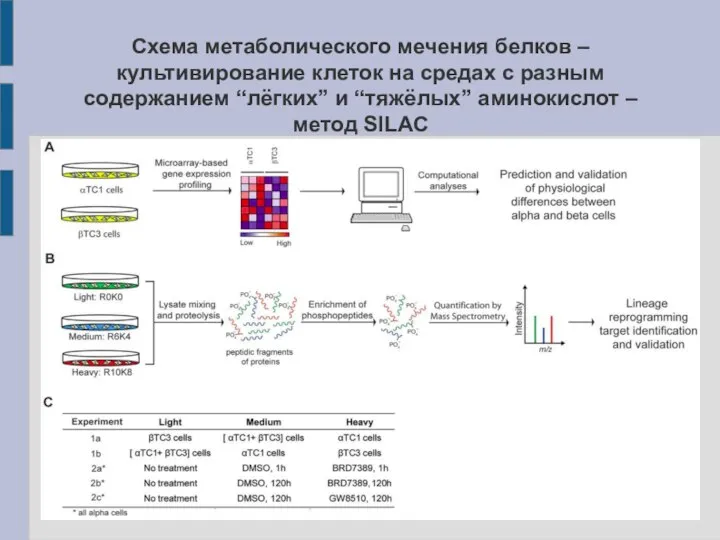
- 19. Схема метаболического мечения белков – культивирование клеток на средах с разным содержанием “лёгких” и “тяжёлых” аминокислот
- 20. Форматы представления МС-данных 1. “Родные” форматы приборов TDF (Bruker), t2d (ABI), lcd (Shimadzu), tdc (Physical Electronics)
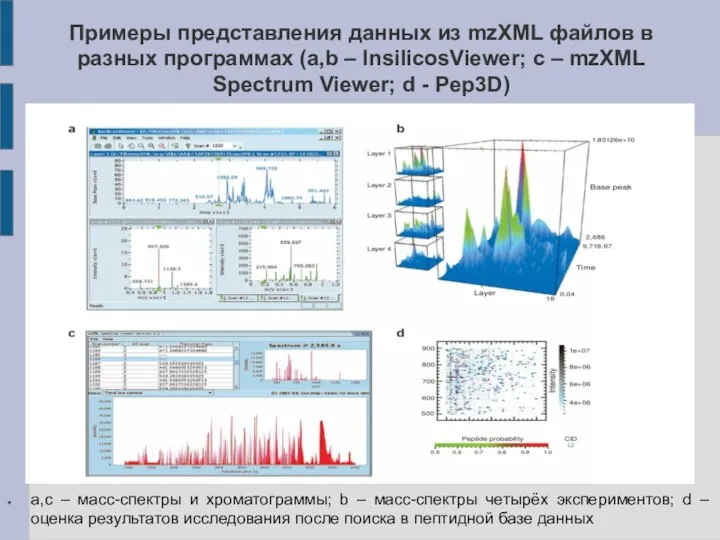
- 21. Примеры представления данных из mzXML файлов в разных программах (a,b – InsilicosViewer; c – mzXML Spectrum
- 22. Анализ результатов МС-эксперимента 1. Поиск по базам данных масс-спектров пептидных фрагментов записей, отвечающих (с учётом посттрансляционных
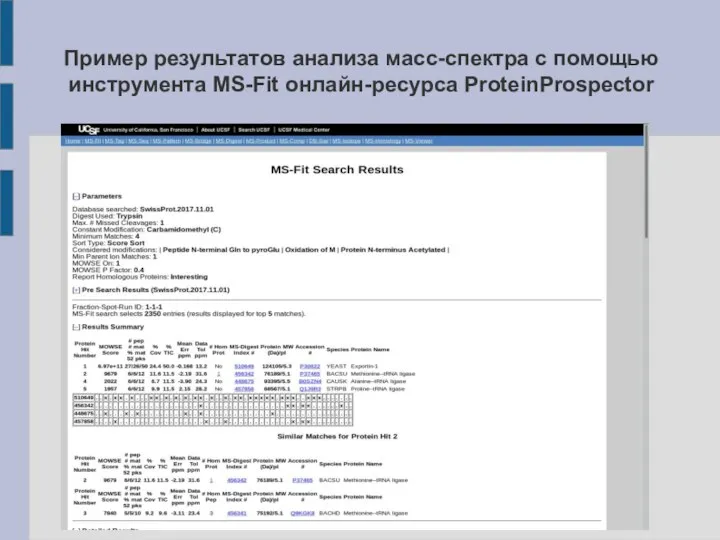
- 23. Пример результатов анализа масс-спектра с помощью инструмента MS-Fit онлайн-ресурса ProteinProspector
- 24. FASTA – универсальный формат представления первичной структуры белков и нуклеиновых кислот >BBB04705.1 M1 protein [Influenza A
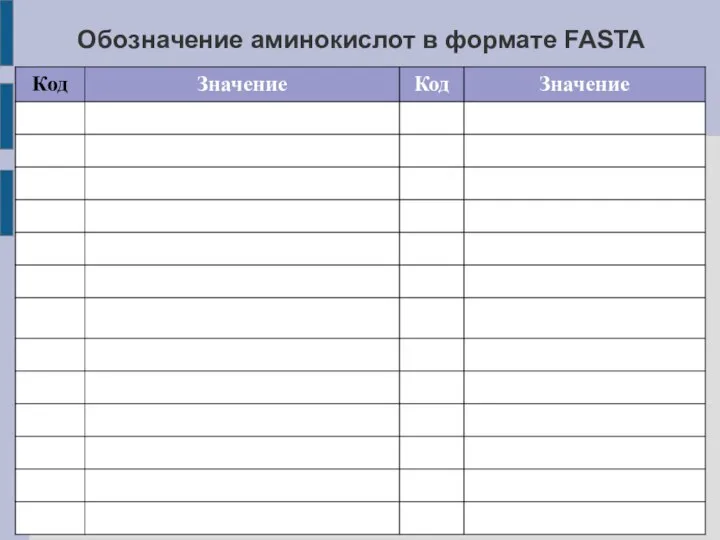
- 25. Обозначение аминокислот в формате FASTA
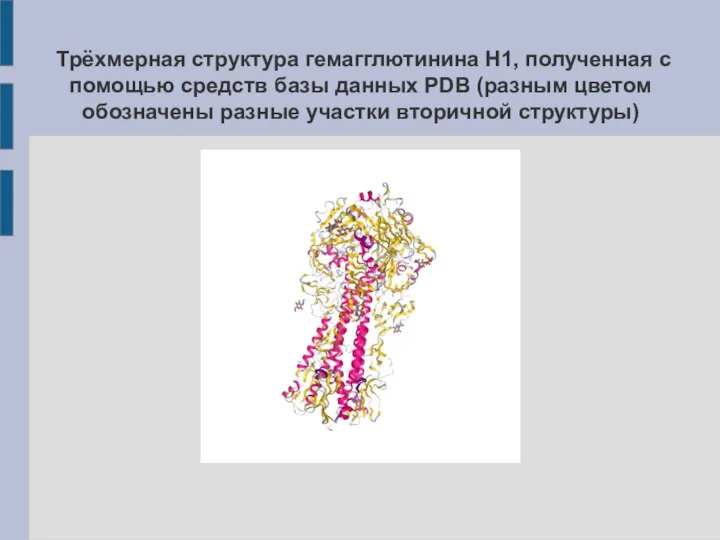
- 26. Трёхмерная структура гемагглютинина H1, полученная с помощью средств базы данных PDB (разным цветом обозначены разные участки
- 28. Скачать презентацию













 Растения Нерюнгринского района
Растения Нерюнгринского района Анастомоз по типу Бок в бок
Анастомоз по типу Бок в бок Сравнение типов нервной системы
Сравнение типов нервной системы Грегор Йоган Мендель
Грегор Йоган Мендель Будова та функції кінцевого мозку
Будова та функції кінцевого мозку Japanese cherry flowers
Japanese cherry flowers Уровни организации живой материи
Уровни организации живой материи Презентация на тему ПРИРОДНЫЕ СООБЩЕСТВА
Презентация на тему ПРИРОДНЫЕ СООБЩЕСТВА  подготовка к впр
подготовка к впр Карл Лінней (1707-1778)
Карл Лінней (1707-1778) ЕГЭ по биологии
ЕГЭ по биологии Жизненные формы растений
Жизненные формы растений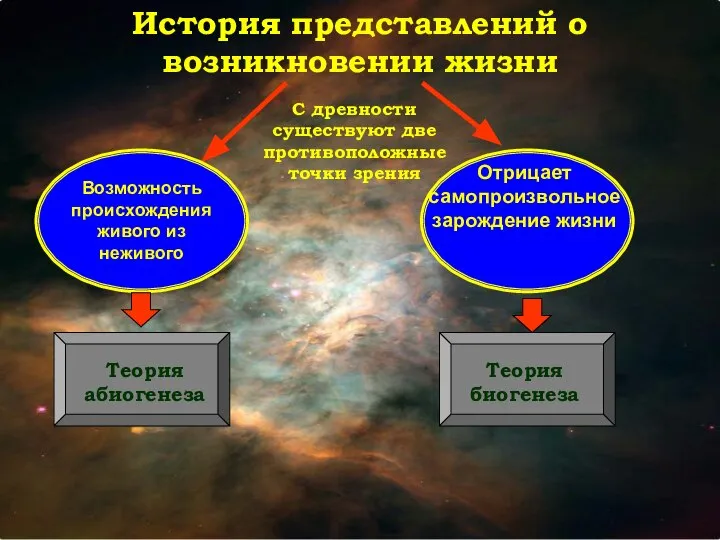 История представлений о возникновении жизни. Возможность происхождения живого из неживого
История представлений о возникновении жизни. Возможность происхождения живого из неживого Животный мир Ленинградской области
Животный мир Ленинградской области Класс Паукообразные. Арахнология
Класс Паукообразные. Арахнология Эволюция земноводных и пресмыкающихся
Эволюция земноводных и пресмыкающихся Органы дыхания и газообмен
Органы дыхания и газообмен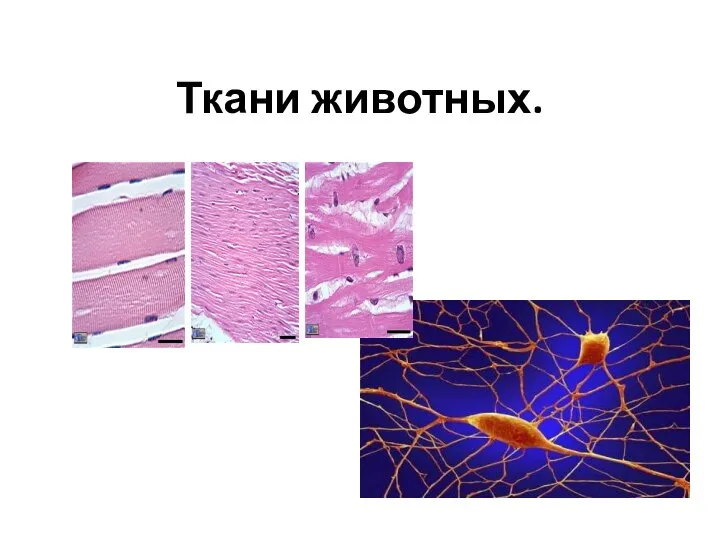 Ткани животных. 6 класс
Ткани животных. 6 класс Растения тоже воевали. 75-летию Победы посвящается
Растения тоже воевали. 75-летию Победы посвящается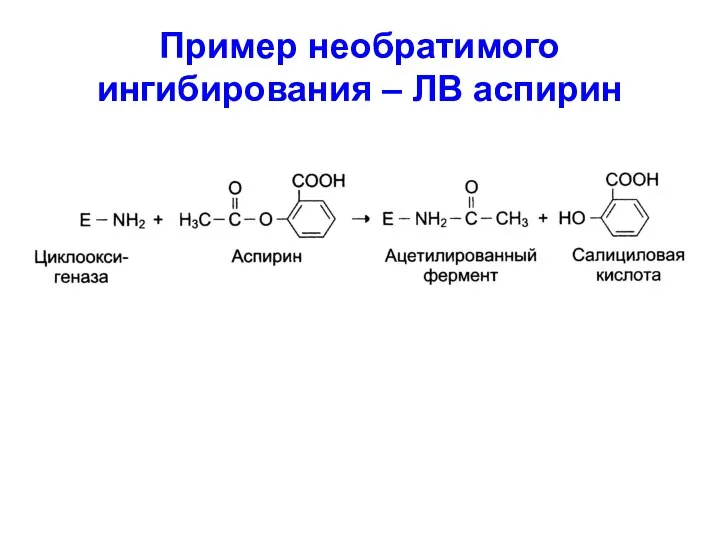 Пример необратимого ингибирования – ЛВ аспирин
Пример необратимого ингибирования – ЛВ аспирин Мочевыделительная система
Мочевыделительная система Коза – это звучит гордо!
Коза – это звучит гордо! Kletochnaya_teoria
Kletochnaya_teoria Закономірності мінливості
Закономірності мінливості Поиск оптимальных условий непосредственно в процессе выращивания микроорганизмов
Поиск оптимальных условий непосредственно в процессе выращивания микроорганизмов 6a4c38ba-5a1d-449b-a6cd-bde09d79dd3e
6a4c38ba-5a1d-449b-a6cd-bde09d79dd3e Закон Геккеля-Мюллера и теория филэмбриогенезов
Закон Геккеля-Мюллера и теория филэмбриогенезов Сцепление генов. Хромосомная теория наследственности. Генетика пола. Сцепленное с полом наследование
Сцепление генов. Хромосомная теория наследственности. Генетика пола. Сцепленное с полом наследование