Содержание
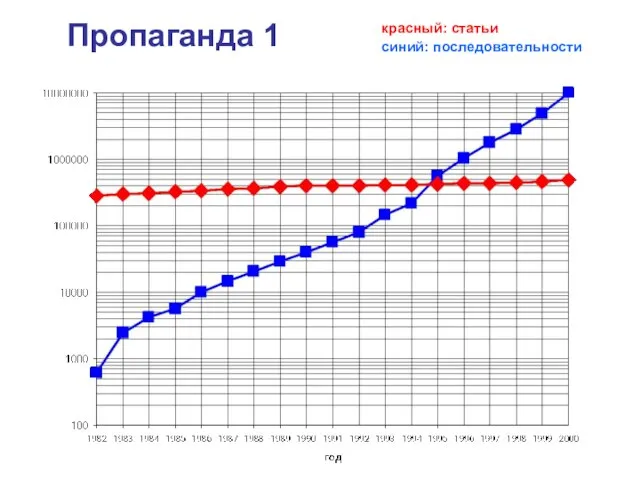
- 2. Пропаганда 1 красный: статьи синий: последовательности
- 3. Анализ индивидуальных генов Поиск родственных белков в банках последовательностей – перенос функции от гомологов Функциональные сайты
- 4. Анализ на уровне индивидуальных генов даёт возможность охарактеризовать 50-75% генов в новом геноме Но: ~100 универсально
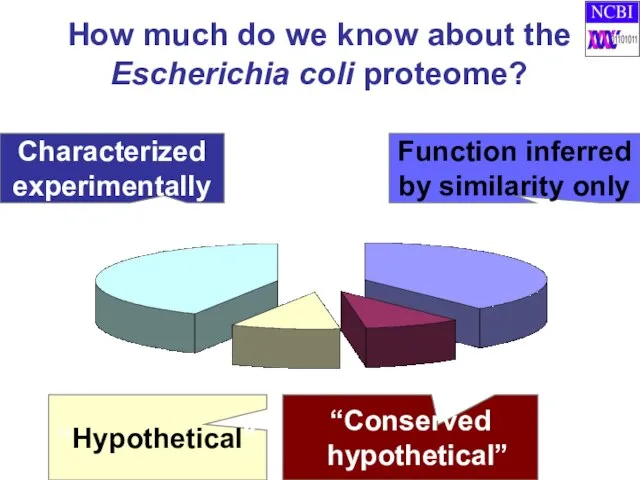
- 5. Characterized experimentally “Hypothetical” Function inferred by similarity only “Conserved hypothetical” How much do we know about
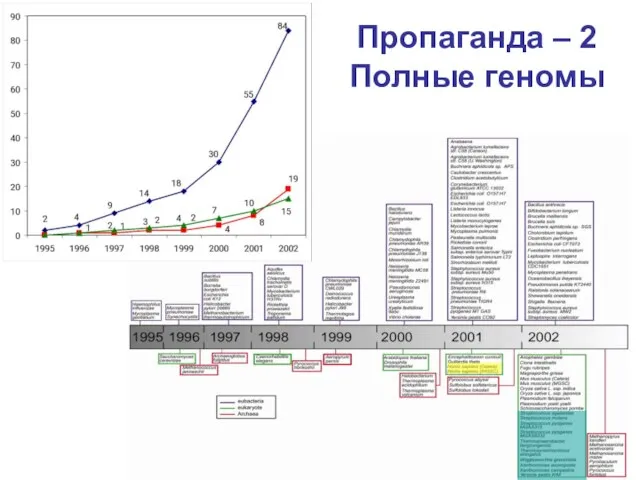
- 6. Пропаганда – 2 Полные геномы
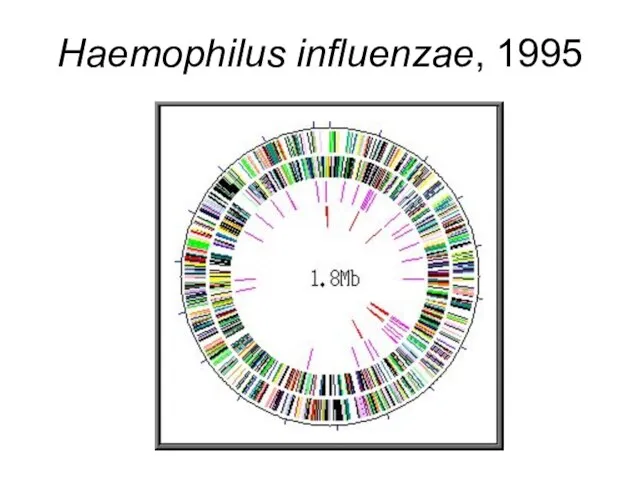
- 7. Haemophilus influenzae, 1995
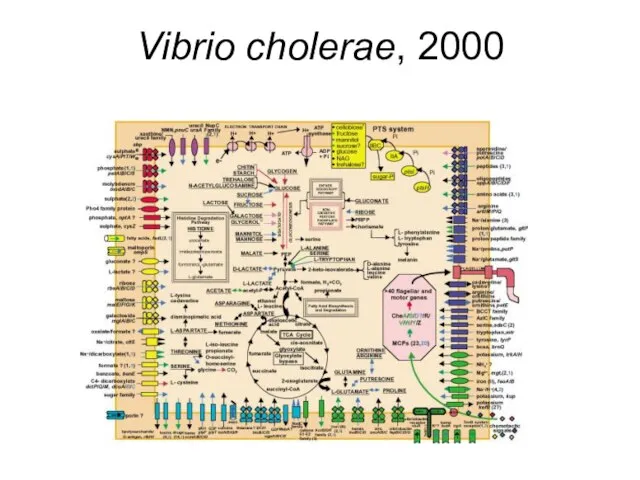
- 8. Vibrio cholerae, 2000
- 9. Сравнительно-геномные подходы Positional clustering Phylogenetic profiling Gene fusions
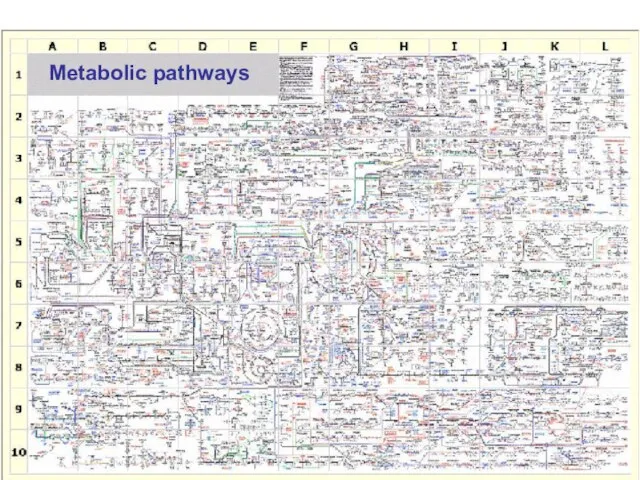
- 10. Metabolic pathways
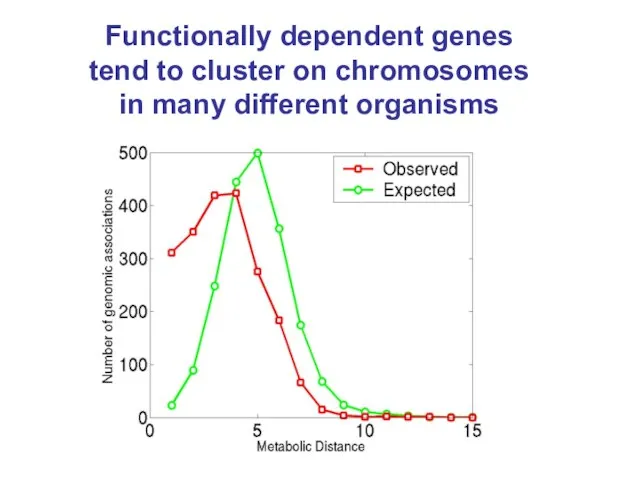
- 11. Functionally dependent genes tend to cluster on chromosomes in many different organisms
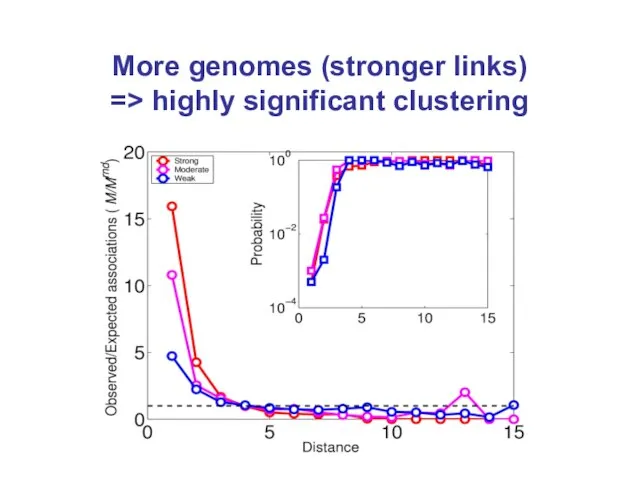
- 12. More genomes (stronger links) => highly significant clustering
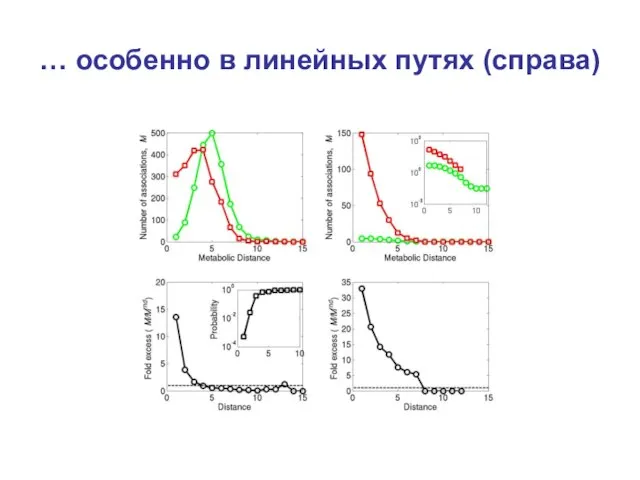
- 13. … особенно в линейных путях (справа)
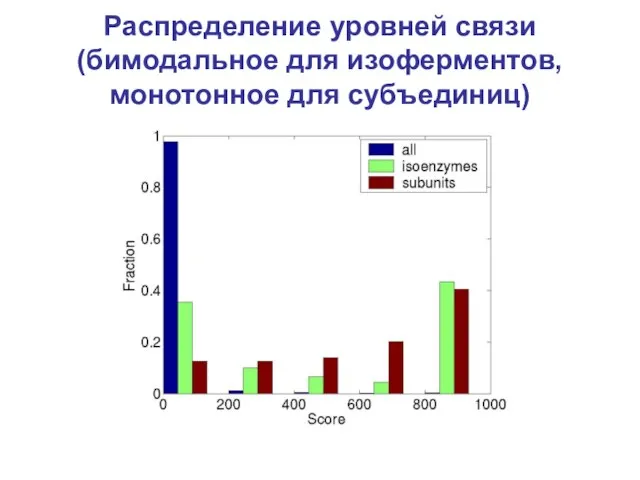
- 14. Распределение уровней связи (бимодальное для изоферментов, монотонное для субъединиц)
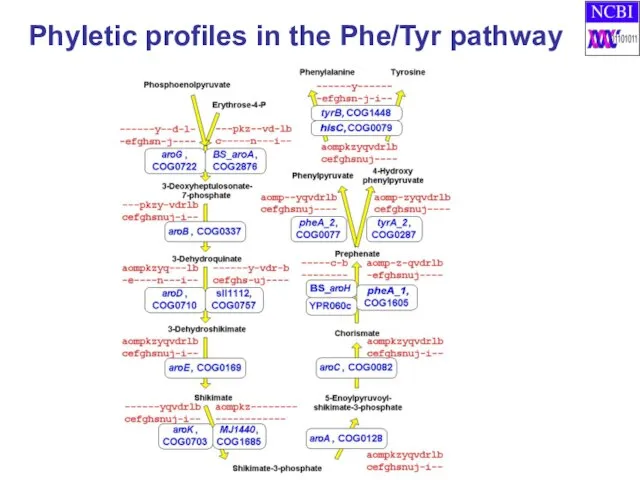
- 15. Phyletic profiles in the Phe/Tyr pathway
- 16. Arithmetics of phyletic patterns 3-dehydroquinate dehydratase (EC 4.2.1.10): Class I (AroD) COG0710 aompkzyq---lb-e----n---i-- Class II (AroQ)
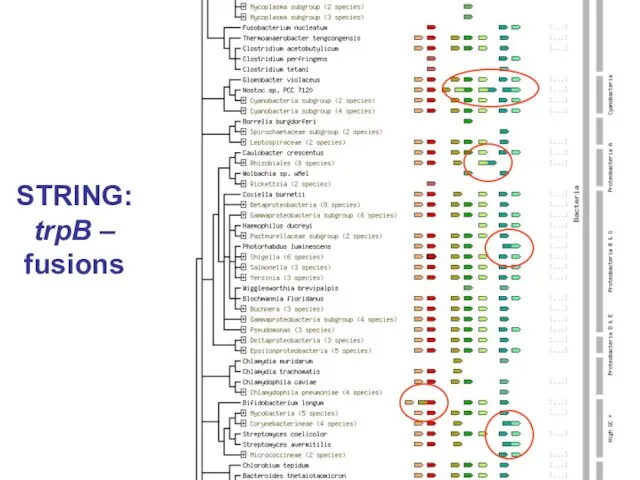
- 17. STRING: trpB – fusions
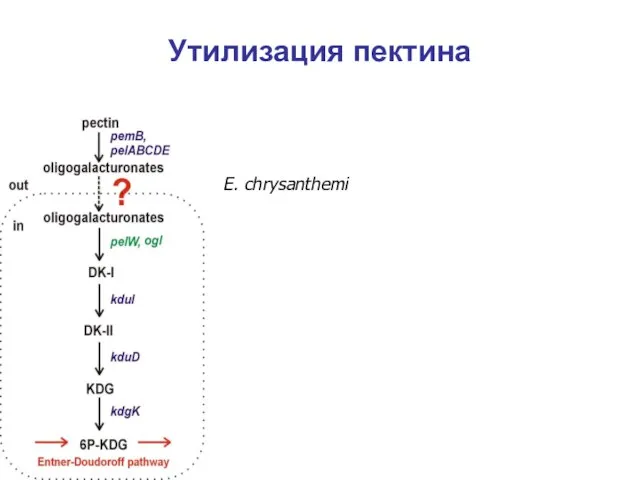
- 18. Утилизация пектина E. chrysanthemi
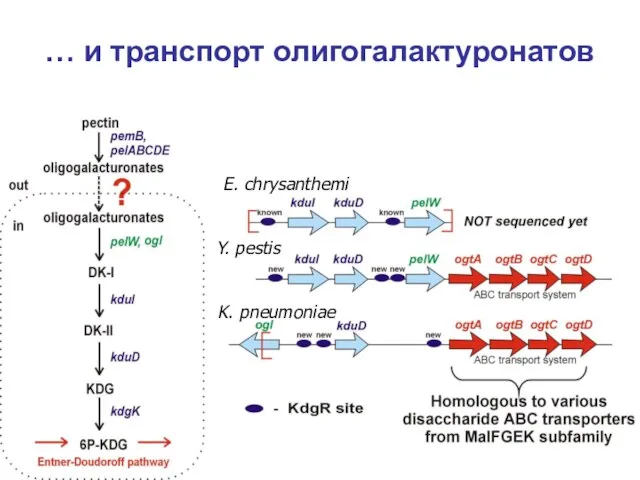
- 19. … и транспорт олигогалактуронатов E. chrysanthemi Y. pestis K. pneumoniae
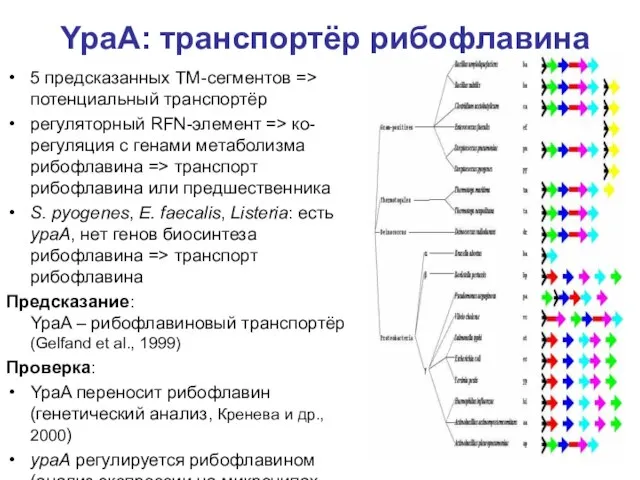
- 20. YpaA: транспортёр рибофлавина 5 предсказанных ТМ-сегментов => потенциальный транспортёр регуляторный RFN-элемент => ко-регуляция с генами метаболизма
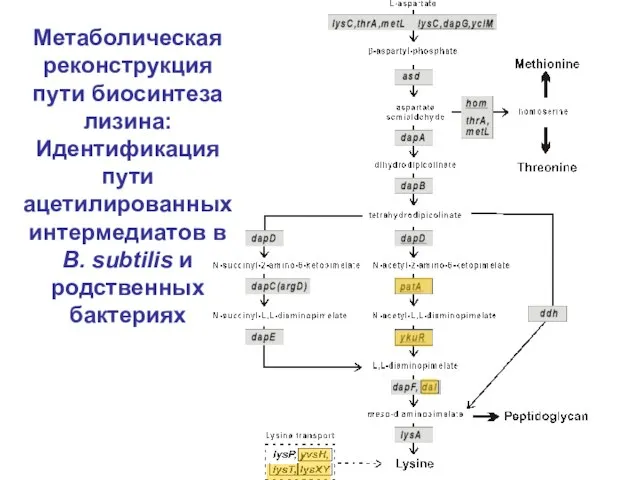
- 21. Метаболическая реконструкция пути биосинтеза лизина: Идентификация пути ацетилированных интермедиатов в B. subtilis и родственных бактериях
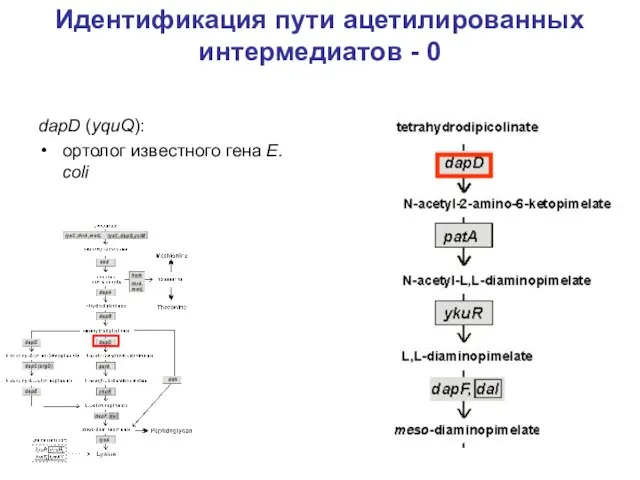
- 22. Идентификация пути ацетилированных интермедиатов - 0 dapD (yquQ): ортолог известного гена E. coli
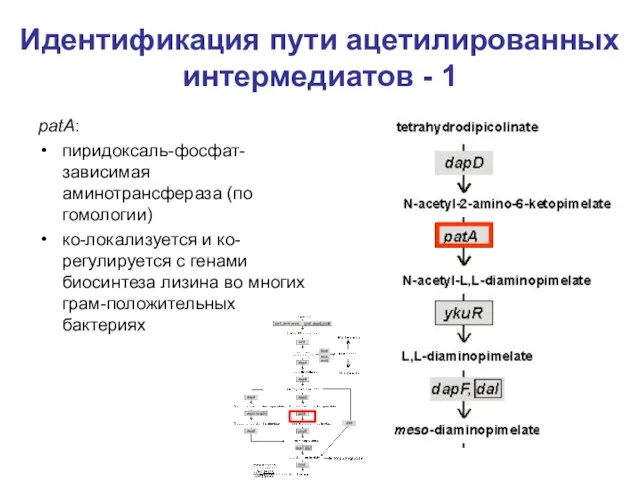
- 23. Идентификация пути ацетилированных интермедиатов - 1 patA: пиридоксаль-фосфат-зависимая аминотрансфераза (по гомологии) ко-локализуется и ко-регулируется с генами
- 24. Идентификация пути ацетилированных интермедиатов - 2 ykuR: N-ацил-L-аминокислота амидогидролаза (по гомологии) ко-локализуется и ко-регулируется с геном
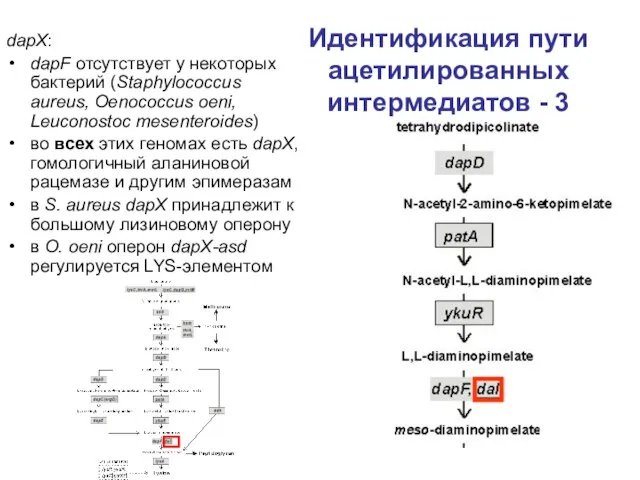
- 25. Идентификация пути ацетилированных интермедиатов - 3 dapX: dapF отсутствует у некоторых бактерий (Staphylococcus aureus, Oenococcus oeni,
- 26. Сравнительная геномика систем утилизации цинка Две роли цинка в бактериях: Структурная в ДНК-полимеразах, праймазах, рибосомных белках
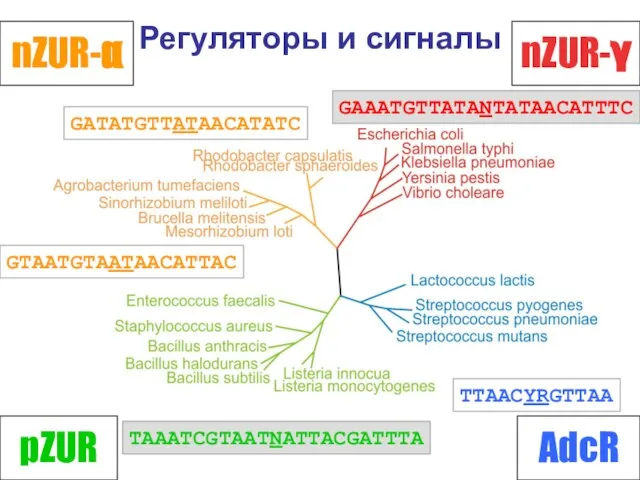
- 27. Регуляторы и сигналы nZUR-γ nZUR-α AdcR pZUR TTAACYRGTTAA GATATGTTATAACATATC GAAATGTTATANTATAACATTTC GTAATGTAATAACATTAC TAAATCGTAATNATTACGATTTA
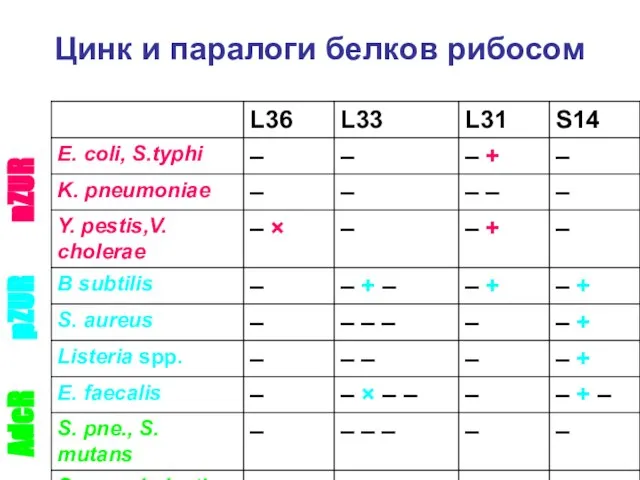
- 28. Цинк и паралоги белков рибосом nZUR pZUR AdcR
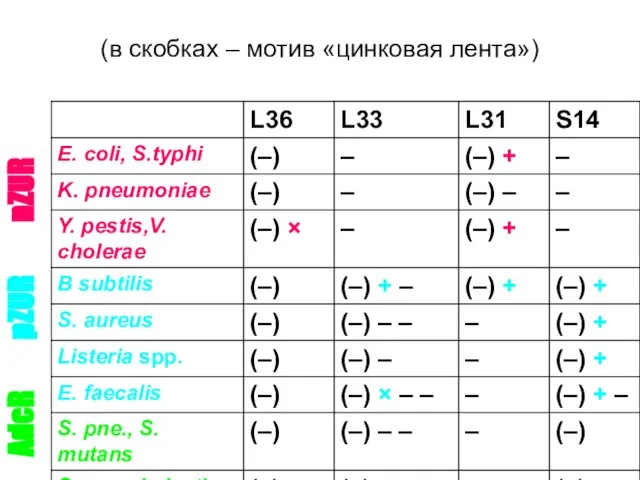
- 29. (в скобках – мотив «цинковая лента») nZUR pZUR AdcR
- 30. Сводка наблюдений: Makarova-Ponomarev-Koonin, 2001: L36, L33, L31, S14 – это единственные рибосомные белки, дуплицированные более, чем
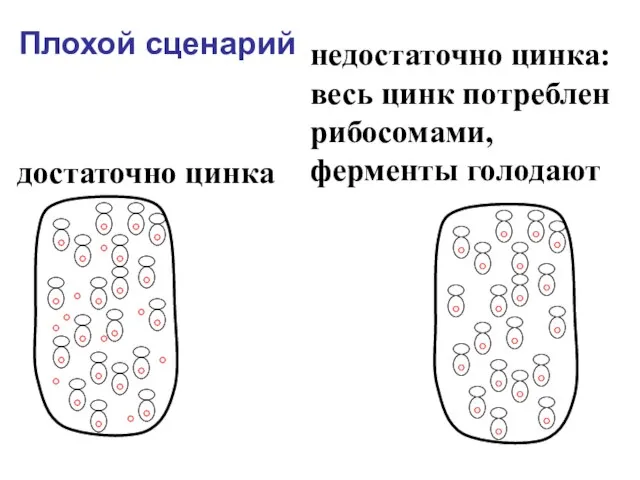
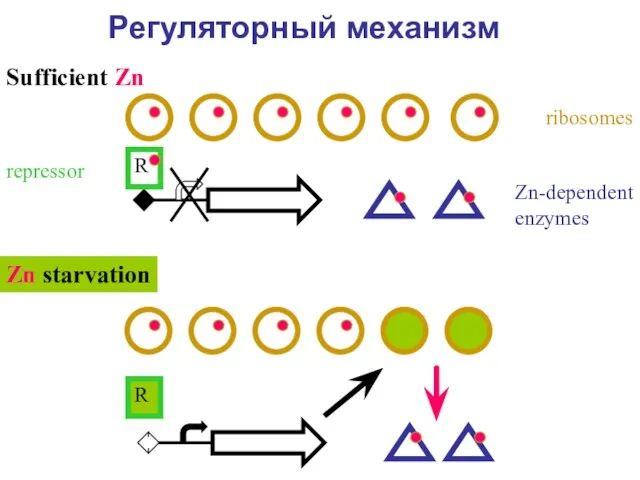
- 31. Плохой сценарий достаточно цинка недостаточно цинка: весь цинк потреблен рибосомами, ферменты голодают
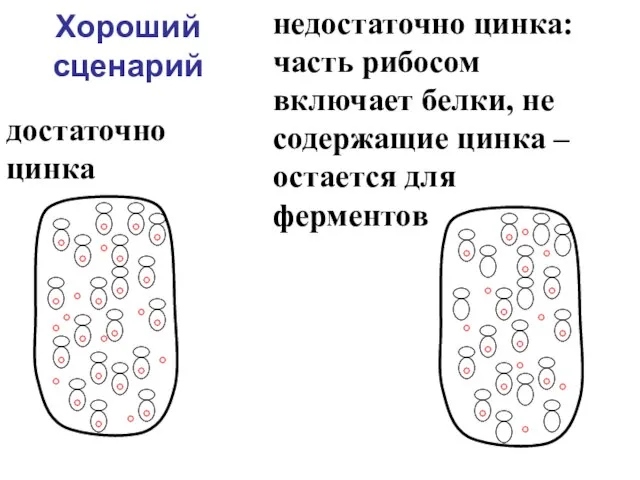
- 32. Хороший сценарий достаточно цинка недостаточно цинка: часть рибосом включает белки, не содержащие цинка – остается для
- 33. Регуляторный механизм ribosomes Zn-dependent enzymes R Sufficient Zn Zn starvation R repressor
- 34. Предсказание … (Proc Natl Acad Sci U S A. 2003 Aug 19;100(17):9912-7.) … и подтверждения (Mol
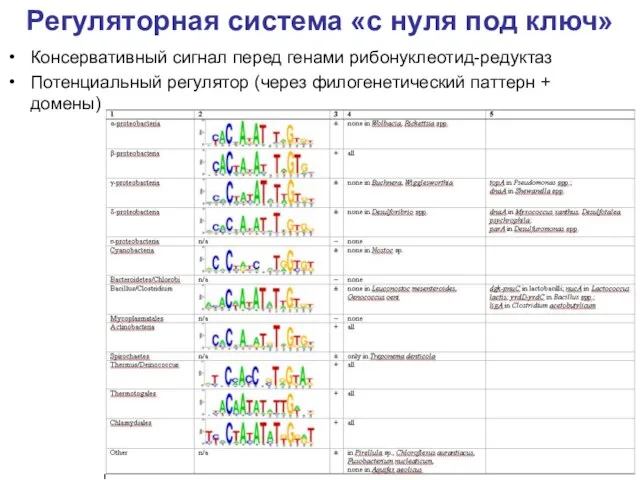
- 35. Регуляторная система «с нуля под ключ» Консервативный сигнал перед генами рибонуклеотид-редуктаз Потенциальный регулятор (через филогенетический паттерн
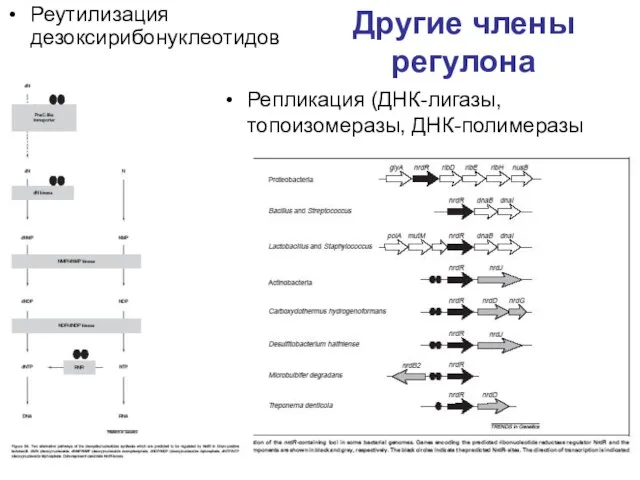
- 36. Другие члены регулона Реутилизация дезоксирибонуклеотидов Репликация (ДНК-лигазы, топоизомеразы, ДНК-полимеразы
- 37. Как регулируется: репрессия в результате кооперативного связывания
- 39. Скачать презентацию









 Организация рабочего места школьника
Организация рабочего места школьника Банда GTA - кошмар российских дорог
Банда GTA - кошмар российских дорог Электронные коллекции НТБ ТПУ
Электронные коллекции НТБ ТПУ Ты и твое здоровье
Ты и твое здоровье КОНТРОЛЛИНГ СИСТЕМЫ УПРАВЛЕНИЕ ПРЕДПРИЯТИЕМ
КОНТРОЛЛИНГ СИСТЕМЫ УПРАВЛЕНИЕ ПРЕДПРИЯТИЕМ ООО «ЛИДЕР»
ООО «ЛИДЕР» Русская культура первой половины 20 века
Русская культура первой половины 20 века Программа индивидуального развития
Программа индивидуального развития Нормативный договор как источник права в современной России
Нормативный договор как источник права в современной России Олимпиада по АФХД
Олимпиада по АФХД Внутренние воды Африки
Внутренние воды Африки Русская живопись в I половине XIX века
Русская живопись в I половине XIX века Презентация на тему Виды сил. Равнодействующая сила. Правила сложения сил (7 класс)
Презентация на тему Виды сил. Равнодействующая сила. Правила сложения сил (7 класс) Наука и астрология
Наука и астрология  Открытое ПО в информатике в школах и вузахМосквы и России
Открытое ПО в информатике в школах и вузахМосквы и России Глава 01Что такое спам
Глава 01Что такое спам Производство органоминеральных удобрений
Производство органоминеральных удобрений Тел төҙәткестәр
Тел төҙәткестәр Монетарная политика
Монетарная политика Построение матрицы ответственности IT-проекта
Построение матрицы ответственности IT-проекта Окружающий мир
Окружающий мир Планируем беременность вместе
Планируем беременность вместе Презентация на тему Будь осторожен с незнакомцем
Презентация на тему Будь осторожен с незнакомцем Stress testing 2010HSBC 18 October 2010
Stress testing 2010HSBC 18 October 2010 Электронные средства для управления производством
Электронные средства для управления производством Презентация на тему Прибавление числа 5
Презентация на тему Прибавление числа 5  Презентация на тему Весна
Презентация на тему Весна Уголовный кодекс Франции
Уголовный кодекс Франции