Содержание
- 2. BLAST – алгоритм для нахождения участков локального сходства между последовательностями. Алгоритм сравнивает входную последовательность с последовательностями
- 3. Protein BLAST: поиск аминокислотной последовательности в базе данных белков Алгоритмы blastp psi-blast phi-blast Здесь описан интерфейс,
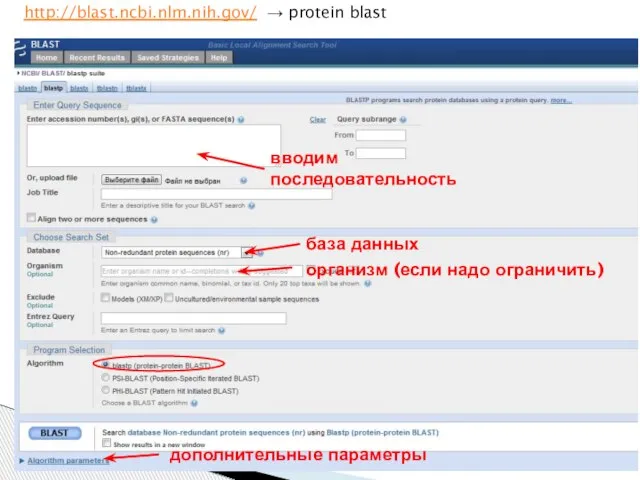
- 4. вводим последовательность база данных организм (если надо ограничить) дополнительные параметры http://blast.ncbi.nlm.nih.gov/ → protein blast
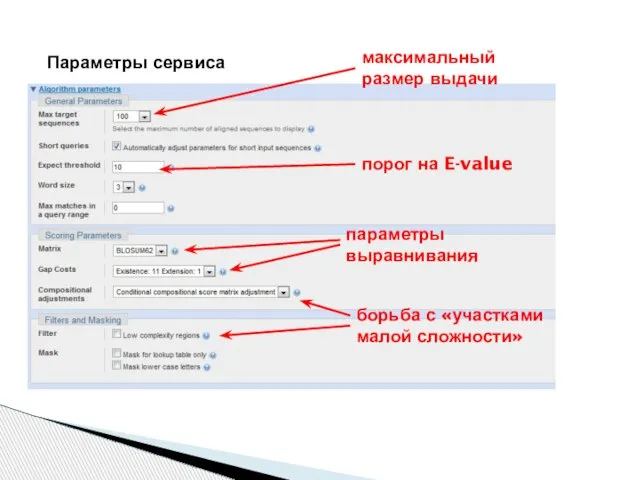
- 5. Параметры сервиса максимальный размер выдачи порог на E-value параметры выравнивания борьба с «участками малой сложности»
- 6. Участок малой сложности Ищем: белок P02929 если отключить “Compositional adjustments” и фильтр, то одной из находок
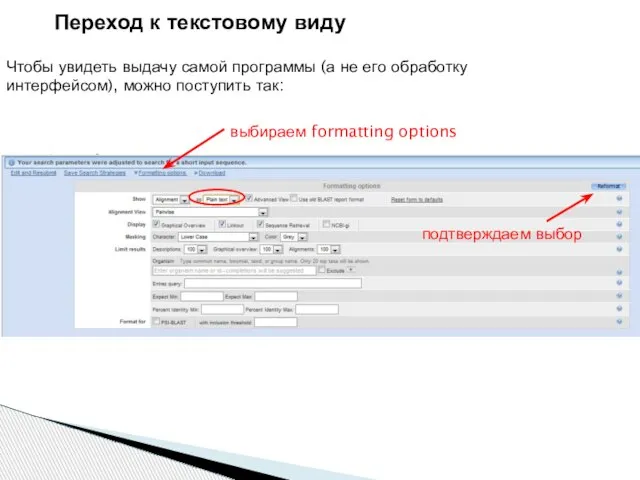
- 7. выбираем formatting options подтверждаем выбор Переход к текстовому виду Чтобы увидеть выдачу самой программы (а не
- 8. Что выдает BLAST? Набор последовательностей, сходных с входной последовательностью для каждой находки приведены E-value (“Expect”), Bit
- 9. Length=129 Score = 78.6 bits (192), Expect = 9e-15, Method: Compositional matrix adjust. Identities = 34/73
- 11. Скачать презентацию





 Технология приготовления Таджикской Чакка
Технология приготовления Таджикской Чакка Центробежные насосы, современные конструкции центробежных насосов
Центробежные насосы, современные конструкции центробежных насосов Логистика распределения и сбыта
Логистика распределения и сбыта Экономика спрос предложение и равновесная цена
Экономика спрос предложение и равновесная цена Презентация на тему Класс Паукообразные
Презентация на тему Класс Паукообразные  Регистрация на портале Gosuslugi.ru
Регистрация на портале Gosuslugi.ru «Донорство в Озинском районе»
«Донорство в Озинском районе» Автоматизация общего анализа кровиГематологические анализаторы Diatron (Австрия)
Автоматизация общего анализа кровиГематологические анализаторы Diatron (Австрия) УЛЬТРАЗВУКОВАЯ ИДЕНТИФИКАЦИЯ
УЛЬТРАЗВУКОВАЯ ИДЕНТИФИКАЦИЯ Концепция институционализма
Концепция институционализма КУЛЬТУРА РОССИИ В XVI в Выполнила Тарасова Е.В.
КУЛЬТУРА РОССИИ В XVI в Выполнила Тарасова Е.В. Старинные меры длины и веса
Старинные меры длины и веса Khimicheskoe_oruzhie_xA_Vidy_posledstvia_deystvie_na_cheloveka
Khimicheskoe_oruzhie_xA_Vidy_posledstvia_deystvie_na_cheloveka Оформление и защита проекта
Оформление и защита проекта Чистоговорки на уроках литературного чтения
Чистоговорки на уроках литературного чтения Схемы
Схемы Тема урока
Тема урока Суицид. О мерах профилактики среди детей и подростков
Суицид. О мерах профилактики среди детей и подростков Презентация "Орфей и Эвридика" - скачать презентации по МХК
Презентация "Орфей и Эвридика" - скачать презентации по МХК Девиантное поведение школьников как фактор социальной опасности и его профилактика
Девиантное поведение школьников как фактор социальной опасности и его профилактика Композиционное, стилевое и цветовое единство в изделиях народных промыслов. Искусство Гжели
Композиционное, стилевое и цветовое единство в изделиях народных промыслов. Искусство Гжели Безопасностьобразовательного учреждения
Безопасностьобразовательного учреждения Вопросы взаимодействия учреждений здравоохранения и Регионального отделения Фонда социального страхования Российской Федерац
Вопросы взаимодействия учреждений здравоохранения и Регионального отделения Фонда социального страхования Российской Федерац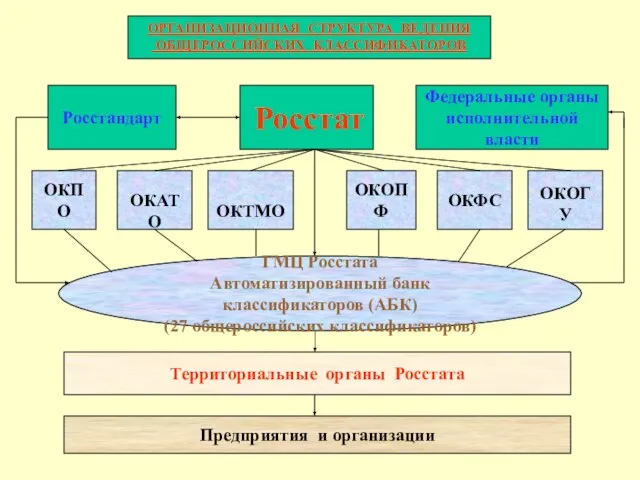 Росстат
Росстат Преемственность в подходах к реализации основных образовательных программ ДОУ и НОО. Требования к результатам освоения ООП НОО в
Преемственность в подходах к реализации основных образовательных программ ДОУ и НОО. Требования к результатам освоения ООП НОО в 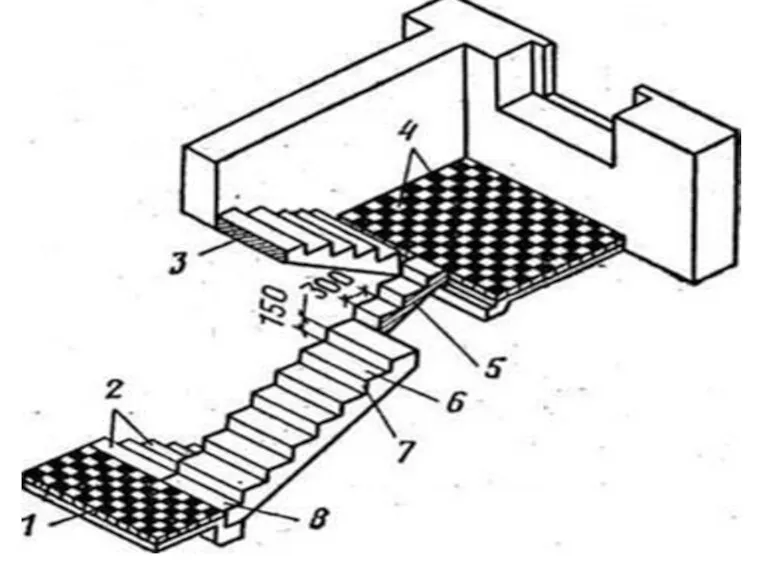 Лестница. Элементы лестницы
Лестница. Элементы лестницы МОУ ДОД «Детско-юношеский центр»
МОУ ДОД «Детско-юношеский центр» Планирование и проектирование организации
Планирование и проектирование организации