for cytocrome oxidase subunit
1.
ACCESSION 916597
VERSION
KEYWORDS .
SOURCE mitochondrion Pseudopleuronectes hersensteini (Jordan & Snyder,
1901)
ORGANISM Pseudopleuronectes hersensteini (Jordan & Snyder, 1901)
Unclassified.
REFERENCE 1 (bases 1 to 613)
AUTHORS Scharina,S., Kartavtsev,Y., Goto,T., Chichvarkhin,A., Hanzawa,N.,
Sokolovsky,A., Balanov,A., Vinnikov,K. and Ivankov,V.
TITLE BARCODING OF FAR EASTERN FLATFISH SPECIES OF RUSSIA ON CYTOCHROME
OXIDASE 1 AND CYTOCHROME B GENE SEQUENCE DATA
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 613)
AUTHORS Kartavtsev,Y., Scharina,S., Goto,T. and Chichvarkhin,A.
TITLE Direct Submission
JOURNAL Submitted (01-JUN-2007) Group of Genetic Resources, Institute of
Marine Biology, Paltchevski 17, Vladivostok, Primorye 690041,
Russia
COMMENT Bankit Comment: Peter the Great Bay, East Sea, August, 2006
[email protected].
FEATURES Location/Qualifiers
source 1..613
/organism="Pseudopleuronectes hersensteini (Jordan &
Snyder, 1901)"
/organelle="mitochondrion"
/mol_type="genomic DNA"
/PCR_primers="fwd_name: FishF1, fwd_seq:
tcaaccaaccacaaagacattggcac, rev_name: FishR1, rev_seq:
tagacttctgggtggccaaagaatca"
/genotype="7K-F1"
/note="Pisces, Pleuronectiformes, Pleuronectidae,
Pseudopleuronectes hersensteini"
gene 11..613
/gene="CO-1"
CDS 11..613
/gene="CO-1"
/codon_start=1
/transl_table=2
/protein_id="PROT_1_bankit916597"
/translation="MSLGTGLSLLIRAELSQPGALLGDDQIYNVIVTAHAFVMISL**
YQLWLEGSETDYPLMIGAPEYGLPSNKNMSFWLLPPSFLLLLASSGWSRGGN*WTVTP
H*LELAHAGAS*THHFSLHLAGISQF*GQSTLSPHMNETNSSTMYQIPYLFGPYNYRC
PSSPFPPVLAAGITCYWQTAT*TQPSLTLR*GWPILYLLG"
BASE COUNT 147 a 172 c 124 g 170 t
ORIGIN
1 tgttgtttgt gtgagcctgg ggacaggcct aagtctgctc attcgagcag agctaagcca
61 acctggggct ctcctgggag acgaccaaat ttataacgta atcgtcaccg cacacgcctt
121 tgtaataatt tctttatagt aataccaatt atgattggag ggttcggaaa ctgactatcc
181 attaataatt ggggcccccg aatatggcct tccctcgaat aaaaacatga gcttctgact
241 tctaccccca tccttcctcc ttcttttggc ctcttcaggg tgaagccggg gcgggaacag
301 gtgaaccgtt accccccact agctggaact agcacacgcc ggagcatcgt agactcacca
361 tttctctctt caccttgccg gaatttctca attctagggg caatcaactt tatcaccaca
421 tataaatgaa accaacagca gtactatgta ccaaatcccc tatttgtttg ggccgtataa
481 ttaccgctgt ccttcttctc ctttccctcc ggttctagcc gctggcatta catgctactg
541 acagaccgca acctaaacac aaccttcttt gaccctgcgg agggggtgac ccattcttta
601 cctgttgggt taa
//
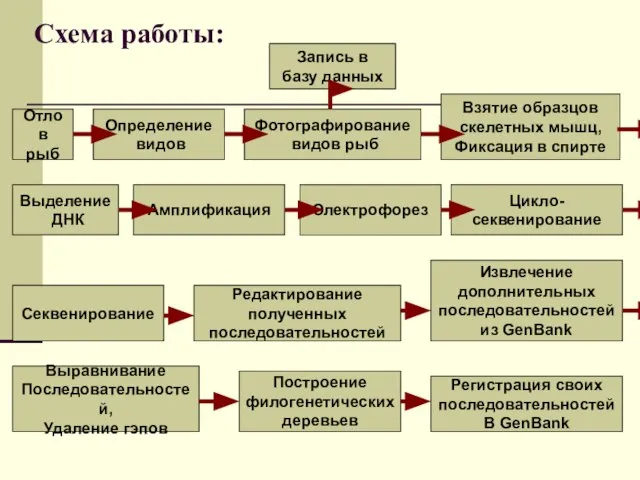
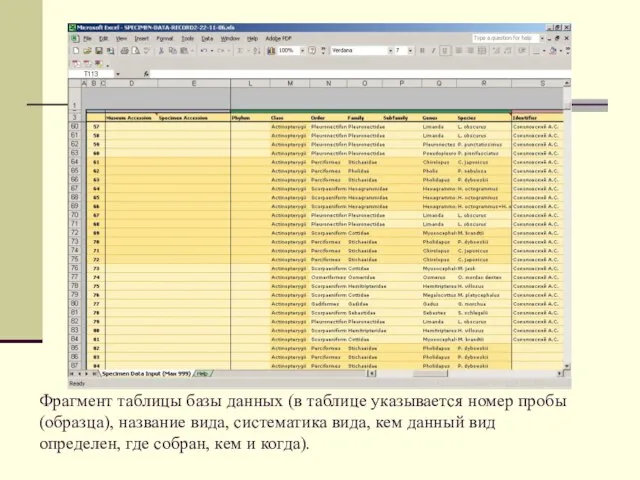
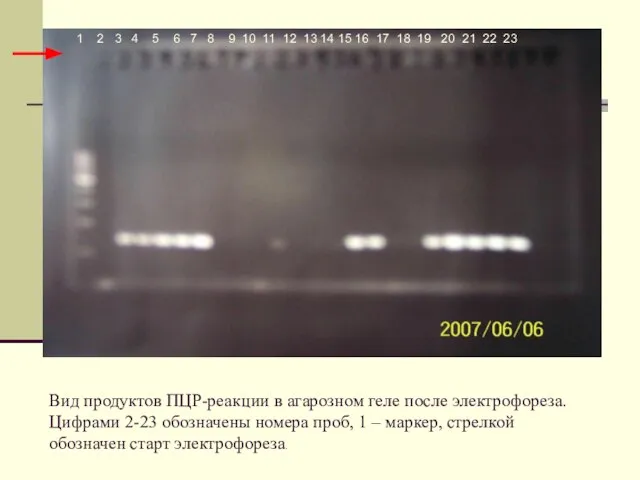
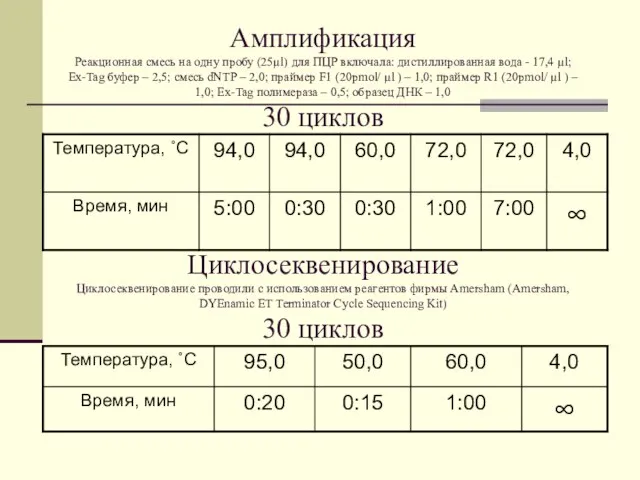
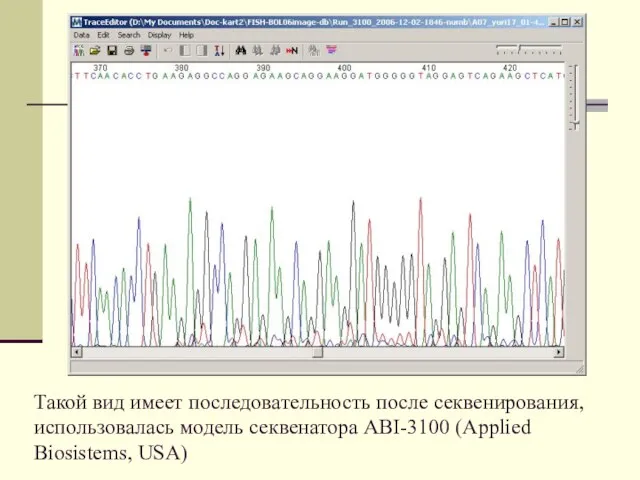
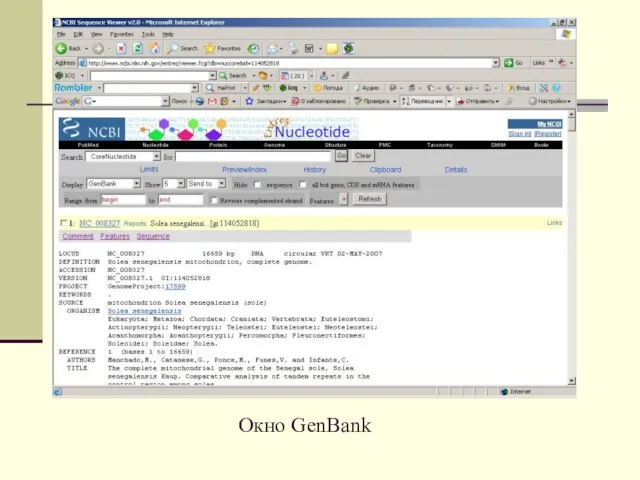
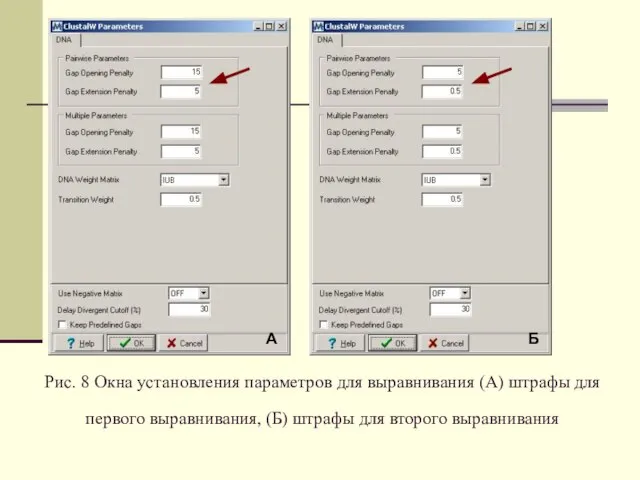
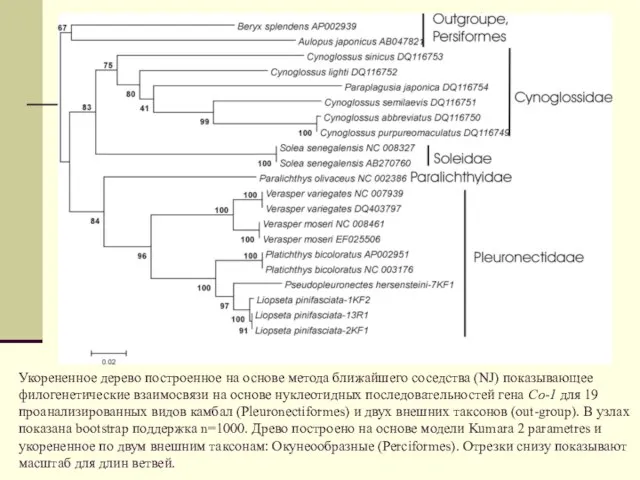
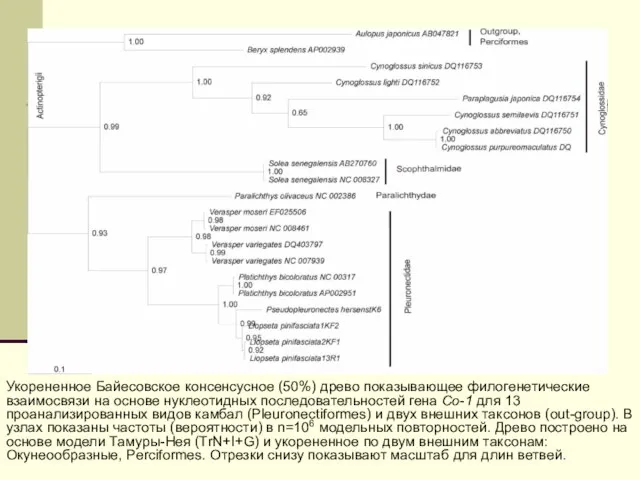
Так выглядит запись одной из последовательностей в GenBank:







 Панорамна фотозйомка
Панорамна фотозйомка СИСТЕМА ОПЛАТЫ ТРУДА ТК РФ СТАТЬЯ 135 «Установление заработной платы» Заработная плата работнику устанавливается трудовым договор
СИСТЕМА ОПЛАТЫ ТРУДА ТК РФ СТАТЬЯ 135 «Установление заработной платы» Заработная плата работнику устанавливается трудовым договор Воротники
Воротники Эссе на английском
Эссе на английском Раны, способы остановки кровотечений
Раны, способы остановки кровотечений Я умею ставить цели и достигать их
Я умею ставить цели и достигать их «Автоматизированная среда аттестации (АСА)»
«Автоматизированная среда аттестации (АСА)» Агентство по ипотечному жилищному кредитованию
Агентство по ипотечному жилищному кредитованию ФГУП машиностроительный завод им. Дзержинского
ФГУП машиностроительный завод им. Дзержинского Типы персональных компьютеров
Типы персональных компьютеров БАНК УРАЛСИБ НА РЫНКЕ Жилищно-коммунальных услуг
БАНК УРАЛСИБ НА РЫНКЕ Жилищно-коммунальных услуг РБС-Сеть — ваша партнёрская программа
РБС-Сеть — ваша партнёрская программа Правильные многогранники:
Правильные многогранники: Учимся делать салфетки
Учимся делать салфетки Зрительный диктант3
Зрительный диктант3 Петербург Достоевского
Петербург Достоевского Центр практических навыков СтГМА
Центр практических навыков СтГМА фрезерование
фрезерование Основы религиозных культур и светской этики
Основы религиозных культур и светской этики Человек и человечность. Что такое гуманизм
Человек и человечность. Что такое гуманизм Концепт для косметологической клиники
Концепт для косметологической клиники Вибродиагностика насосно-компрессорного оборудования
Вибродиагностика насосно-компрессорного оборудования Пищеварительная система у позвоночных животных
Пищеварительная система у позвоночных животных Индийско-российская программа развития телекоммуникаций и информационных технологий
Индийско-российская программа развития телекоммуникаций и информационных технологий Антонис ван Дейк и Снейдерс
Антонис ван Дейк и Снейдерс Электрошкаф
Электрошкаф Система «ОРГСТРУКТУРА»
Система «ОРГСТРУКТУРА» Терроризм угроза обществу
Терроризм угроза обществу