Слайд 2Полимеразная цепная реакция (ПЦР)
Метод, позволяющий избирательно синтезировать большие количества определённых фрагментов ДНК.
Разработан

Кэри Муллисом в 1983 году (Нобелевская премия 1993 года)
“Полимеразная” – используется фермент ДНК-полимераза
“Цепная” – состоит из повторяющихся циклов, количество ДНК с каждым циклом увеличивается в геометрической прогрессии
Слайд 3Виды ПЦР
1. В зависимости от измерения количества ПЦР-продукта:
качественная
количественная
2. Тип детекции ПЦР-продукта:
электрофоретическая
флюориметрическая
3.

Этап детекции:
в конечной точке
в реальном времени
Слайд 4Применение ПЦР
I. Получение информации
генотипирование
ДНК-диагностика
анализ уровня транскрипции
II. Получение материала (накопление)
клонирование
мутагенез
секвенирование

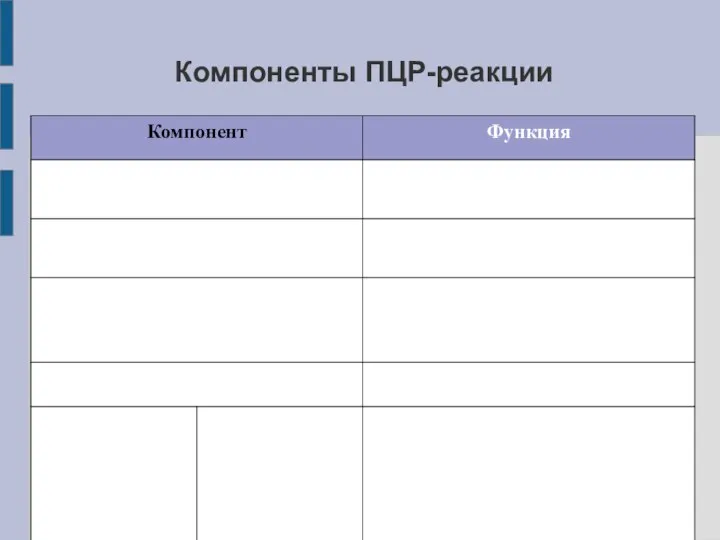
Слайд 6Состав реакционной смеси ПЦР
VРС = 10-50 мкл
ДНК 1-20 пг/мкл (плазмиды, фаги) или

2-20 нг/мкл (геном)
дНТФ 0,2 мМ
праймеры 0,2-1 мкМ
полимераза 0,01-0,05 е.а./мкл
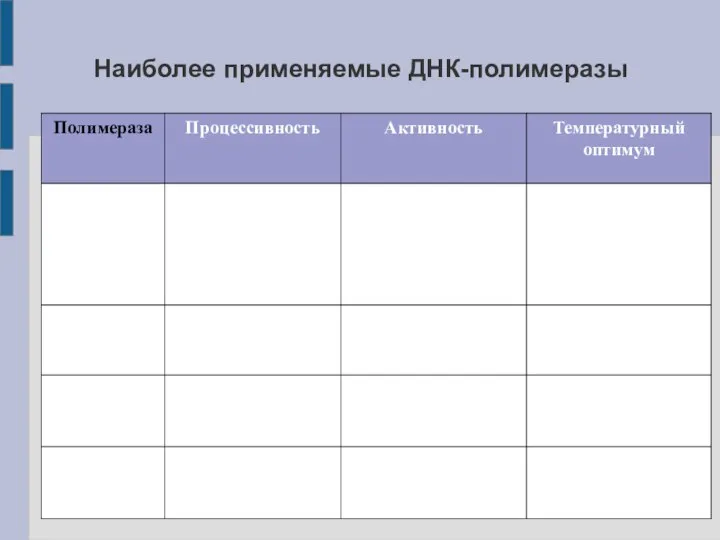
Слайд 8Наиболее применяемые ДНК-полимеразы
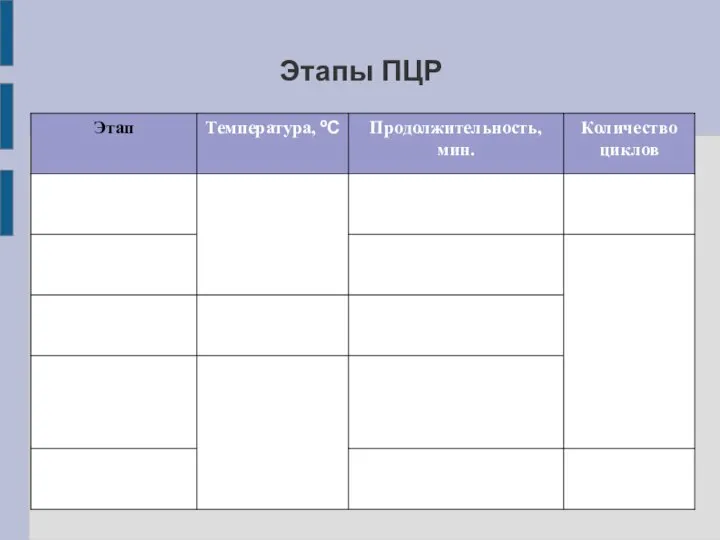
Слайд 9Денатурация (denaturation)
Необходима для разрушения водородных связей между комплементарными цепями ДНК (плавление ДНК)
1.

Температура (Td): определяется свойствами полимеразы, обычно составляет 95ºC. Для более термоустойчивых полимераз (Pwo) температура может быть увеличена.
2. Время: для крупных молекул (геномы) увеличивается, для малых (ампликоны) – уменьшается; время нужно также увеличивать в случае GC%>50%.
Слайд 10Отжиг (annealing)
На этом этапе происходит понижение температуры, и праймеры могут присоединиться к

комплементарным участкам на матрице ДНК
Температура (Ta): зависит от состава праймеров, который определяет температуру плавления (melting) комплекса праймер–матрица (Ta = Tm - 5ºC).
Слайд 11Критерии подбора праймеров
1. GC-состав должен быть в пределах 40-60%. При этом G/C

должны быть распределены равномерно (не образовывать кластеров).
2. Температура плавления у прямого и обратного праймера должна отличаться не более, чем на 5ºC.
3. Должны отсутствовать самокомплементарные участки как внутри праймера (иначе могут образовываться шпильки), так и между праймерами (иначе образуются димеры праймеров).
4. Лучше, если на 3'-конце праймеров будут G/C (связь сильнее), но не более 3х (чтобы не было отжига на неспецифические участки).
Слайд 12Температура плавления праймеров
Tm=4*[n(G)+n(C)]+2*[n(A)+n(T)]
где n(X) - количество нуклеотида X в праймере
![Температура плавления праймеров Tm=4*[n(G)+n(C)]+2*[n(A)+n(T)] где n(X) - количество нуклеотида X в праймере](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/958332/slide-11.jpg)
Слайд 13Элонгация
На этапе элонгации происходит синтез цепей ДНК с помощью ДНК-полимеразы.
1. Температура: определяется

свойствами используемой ДНК-полимеразы и лежит в области оптимума для этого фермента (для Taq – 72ºC).
2. Время: рассчитывают по одной минуте на каждую т.п.н. ампликона (для небольших молекул обычно берут 0,5 мин.).
Слайд 14Онлайн-ресурсы для подбора условий ПЦР
Хранилище нуклеотидных последовательностей генов:
National Center for Bioinformatics (ncbi.nlm.nih.gov/gene)
Подбор

праймеров:
Primer3 (bioinfo.ut.ee/primer3/)
Primer-BLAST (ncbi.nlm.nih.gov/tools/primer-blast/)
Проведение расчётов (концентрации, операции с нуклеотидными последовательностями) и мн. др.:
MOLBIOL (molbiol.ru/scripts)
Слайд 15Домашнее задание №1: подбор праймеров.
1. Найти на NCBI в базе данных Gene
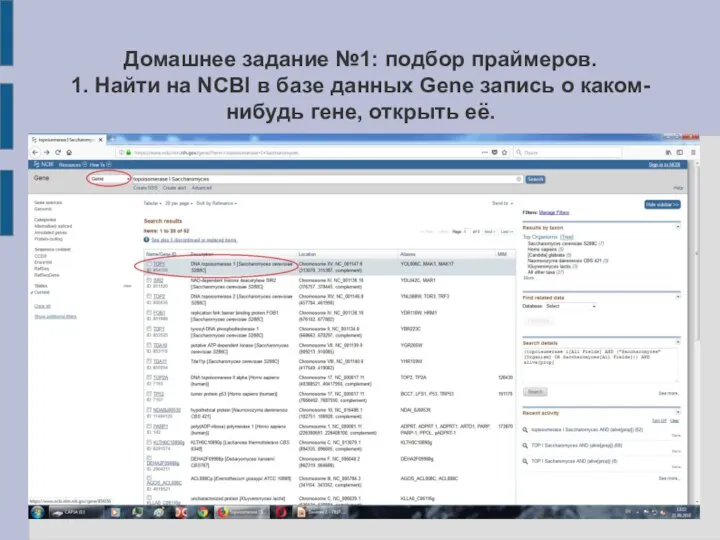
запись о каком-нибудь гене, открыть её.
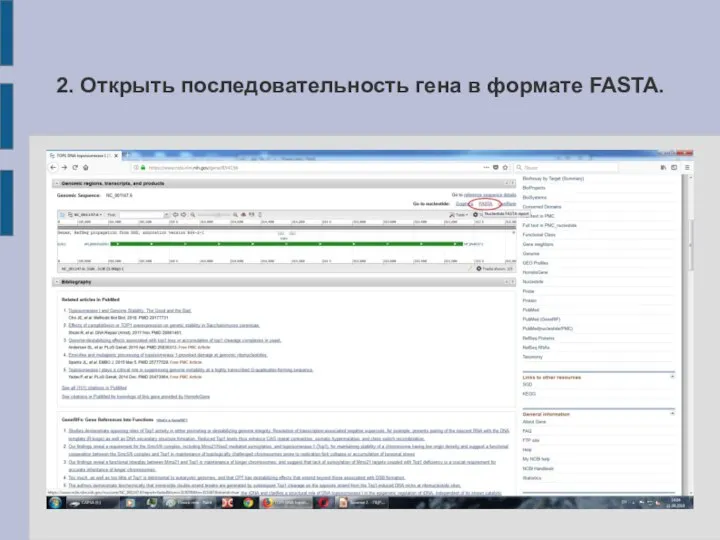
Слайд 162. Открыть последовательность гена в формате FASTA.
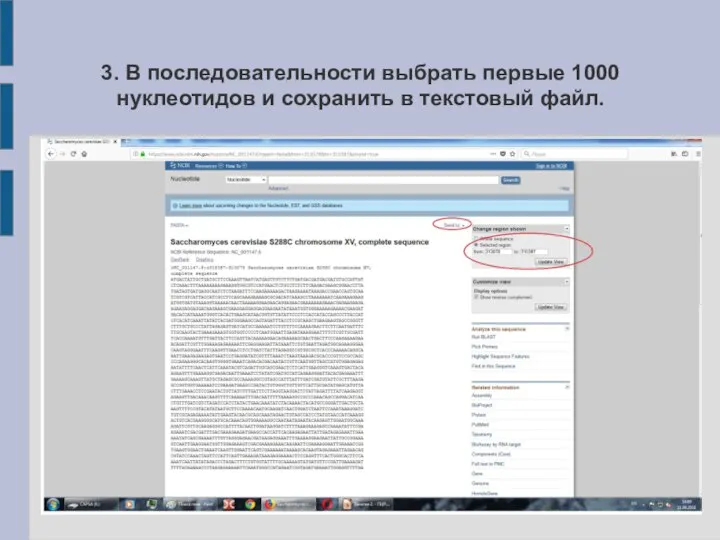
Слайд 173. В последовательности выбрать первые 1000 нуклеотидов и сохранить в текстовый файл.
Слайд 184. “Наивный” подбор праймеров: с помощью сайта Molbiol составить праймеры (по 20
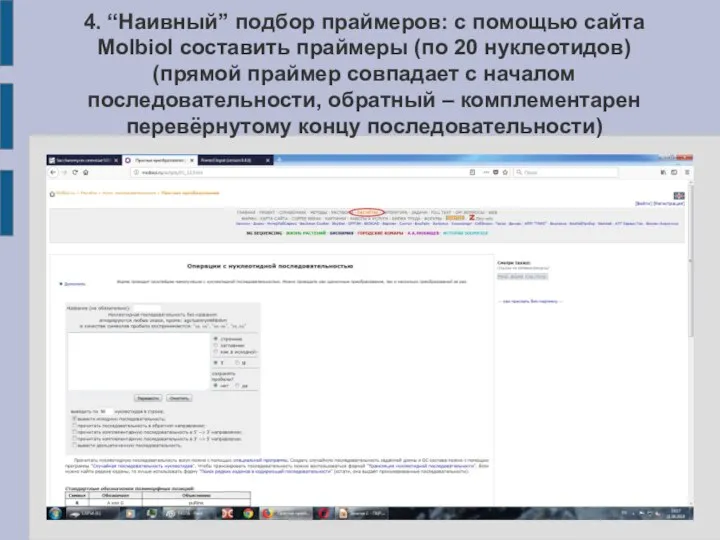
нуклеотидов) (прямой праймер совпадает с началом последовательности, обратный – комплементарен перевёрнутому концу последовательности)
Слайд 195. Подбор “оптимальных” праймеров: скопировать последовательность и вставить в окошко на сайте
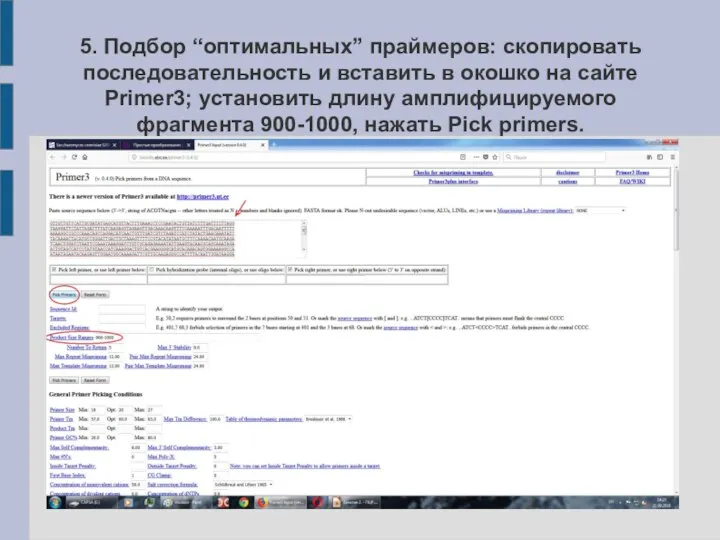
Primer3; установить длину амплифицируемого фрагмента 900-1000, нажать Pick primers.
Слайд 206. Выписать в файл с исходной последовательностью наиболее оптимальные праймеры (прямой и
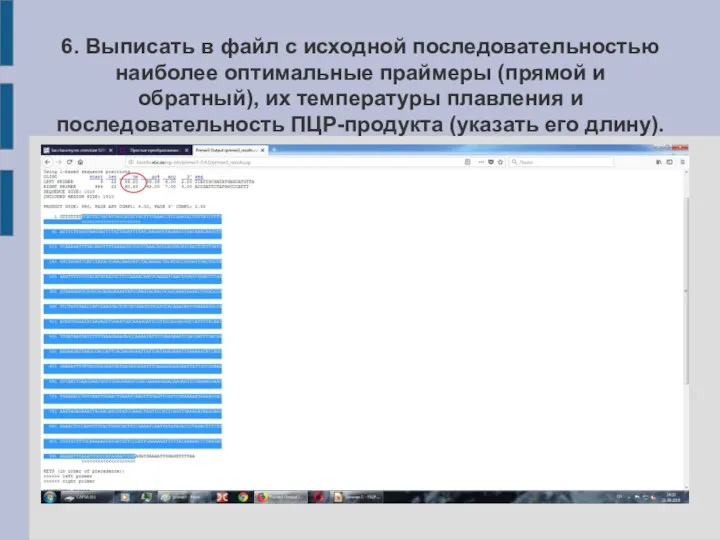
обратный), их температуры плавления и последовательность ПЦР-продукта (указать его длину).
Слайд 217. Проверка качества “наивных” праймеров: вставить последовательности “своих” праймеров и нажать Pick
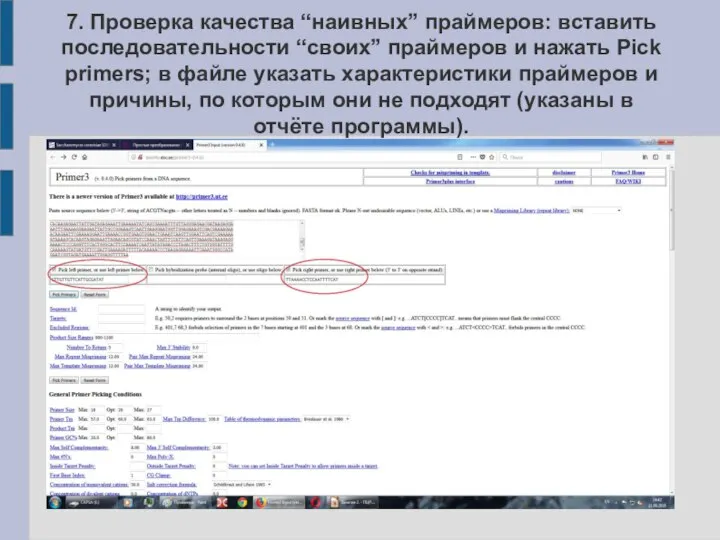
primers; в файле указать характеристики праймеров и причины, по которым они не подходят (указаны в отчёте программы).








![Температура плавления праймеров Tm=4*[n(G)+n(C)]+2*[n(A)+n(T)] где n(X) - количество нуклеотида X в праймере](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/958332/slide-11.jpg)









 Жиры в организме человека
Жиры в организме человека Разнообразие соцветий. Лабораторная работа
Разнообразие соцветий. Лабораторная работа Царство Грибы часть 3
Царство Грибы часть 3 Тема 6. Фотосинтез. Хемосинтез. Энергообмен. Клеточное дыхание
Тема 6. Фотосинтез. Хемосинтез. Энергообмен. Клеточное дыхание Биологиялық қару соның ішінде Сібір жарасы
Биологиялық қару соның ішінде Сібір жарасы Генетика
Генетика Мейоз
Мейоз Индивидуальное развитие организма
Индивидуальное развитие организма Биокатализаторы
Биокатализаторы Технология выращивания комнатных растений
Технология выращивания комнатных растений Изменчивость (11)
Изменчивость (11) Презентация на тему Юный натуралист
Презентация на тему Юный натуралист  Формы естественного отбора
Формы естественного отбора Механизмы проведения возбуждения
Механизмы проведения возбуждения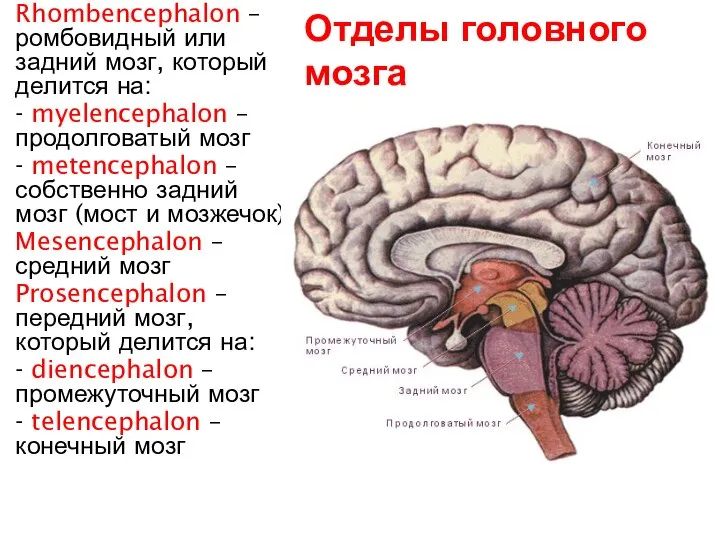 Отделы головного мозга
Отделы головного мозга Анабиоз. Исследовательская работа
Анабиоз. Исследовательская работа Теория панспермии
Теория панспермии Размножение организмов
Размножение организмов Нервные окончания
Нервные окончания Белый аист
Белый аист Игра – путешествие Удивительный мир природы. Станция Ох, уж эти растения
Игра – путешествие Удивительный мир природы. Станция Ох, уж эти растения dnk_i_rnk_-_nukleinovye_kisloty
dnk_i_rnk_-_nukleinovye_kisloty История первых великих путешествий
История первых великих путешествий Как птицы и звери готовятся к зиме
Как птицы и звери готовятся к зиме Селекция животных
Селекция животных Метод лечебной физкультуры иппотерапия
Метод лечебной физкультуры иппотерапия Соединительные ткани I
Соединительные ткани I Жүйке жүйесінің аурулары
Жүйке жүйесінің аурулары