Слайд 2Меры активности
EC50 - полумаксимальная эффективная концентрация, означает концентрацию лиганда, которая вызывает эффект,

равный половине максимального возможного для данного лиганда
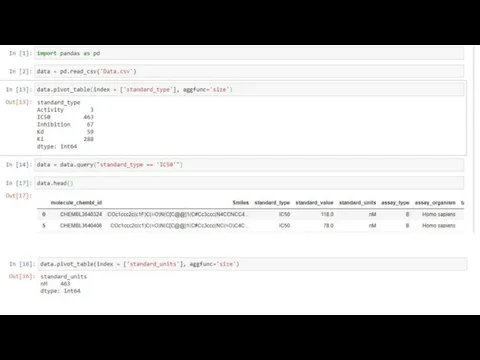
IC50 - является количественным индикатором, который показывает, сколько нужно лиганда—ингибитора для ингибирования биологического процесса на 50 %
Ki - концентрация конкурентного лиганда, при которой он связывается с половиной мест связывания, имеющихся на реакционном субстрате, при условии отсутствия агониста
Стандартные единицы измерения - nM
Слайд 4Выбираем свою меру активности
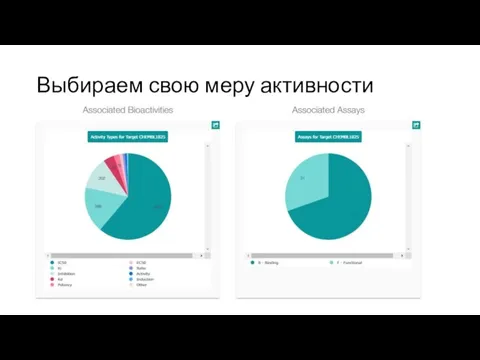
Слайд 6Is it active ? Probably…
IC50(compound 1) = 1000 nM
IC50(compound 2) = 100000

nM
Слайд 7Как узнать активно соединение или нет?
Посмотреть комментарий исследователя
Приблизительно определить активность используя «стандартные»

границы
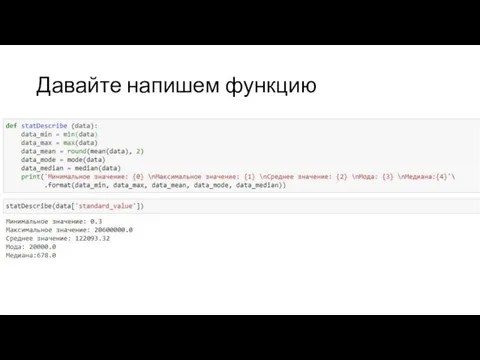
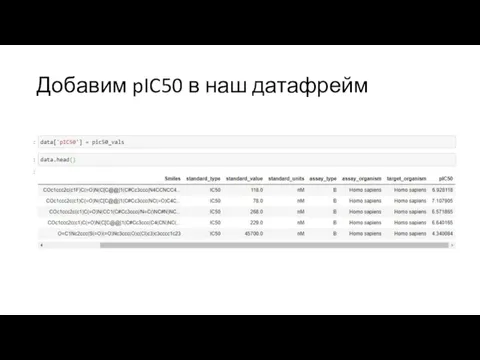
Слайд 10pIC50 (pKi, pEC50)
pIC50 = - log(IC50)
Как это работает?
Переводим в моли(М): IC50 =

100,000 nM *10^-9= 10^-4
Находим отрицательный логарифм:
–log(10^-4) = -(-4)log(10) = 4
3. Закономерность: чем выше pIC50, тем активнее средство.
Слайд 11Давайте писать еще больше функций!!

Слайд 14«Стандартные» значения для IC50
IC50 < 1000 nM – активный компонент (6.0 и
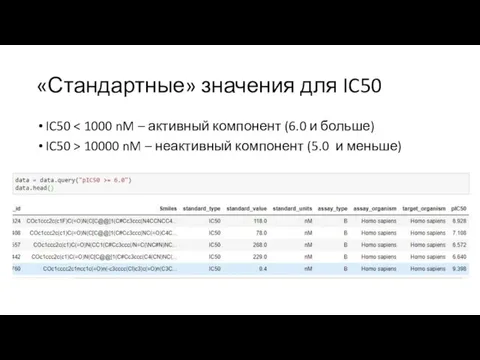
больше)
IC50 > 10000 nM – неактивный компонент (5.0 и меньше)
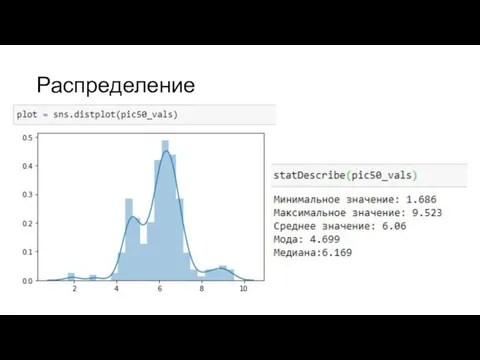
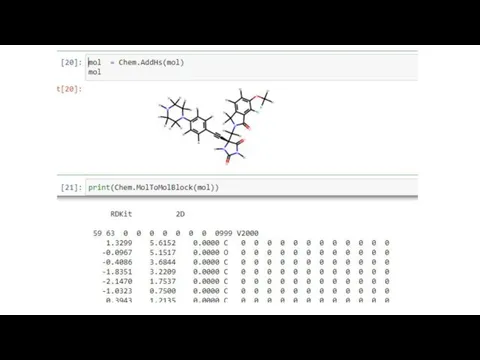
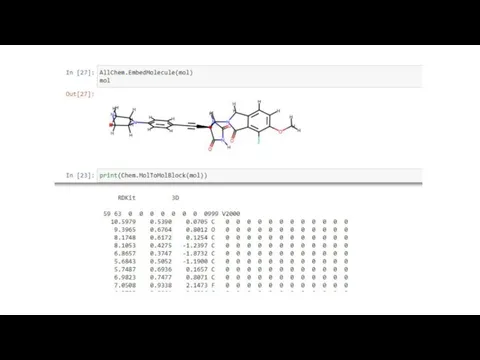
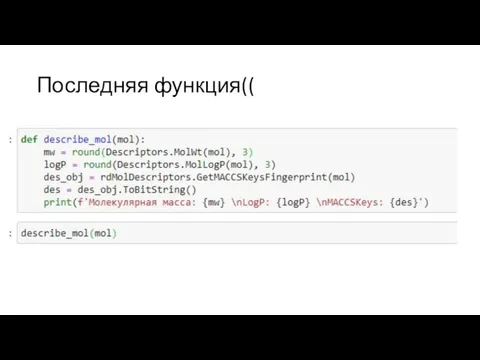
Слайд 15Введение в RDkit
Молекулярные дескрипторы

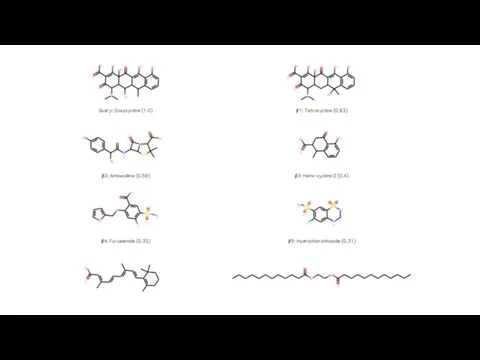
Слайд 19Идея:
1. Объединить схожие молекулы в кластеры.
2. Найти центральные молекулы каждого кластера.

3. Построить модель фармакофора
Слайд 20Молекулярные дескрипторы
Одномерные (1D): Растворимость, logP, молекулярная масса
Глобальный дескриптор: одно значение представляет все

соединение
Обычно недостаточны для применения в машинном обучении (ML)
Может быть добавлен к 2D-дескрипторам для улучшения молекулярного кодирования в ML.
Слайд 22Молекулярные дескрипторы
Двухмерные (2D): молекулярные графы, фрагменты молекул, окружения атомов.
Подробное представление отдельных частей

молекулы
Обнаружение так называемых отпечатков пальцев (фингерпринтов)
Очень часто используется в поиске сходства между молекулами и машинном обучении
Слайд 23Молекулярные дескрипторы
Основная идея – представить молекулу в виде бит-строки, где каждый бит
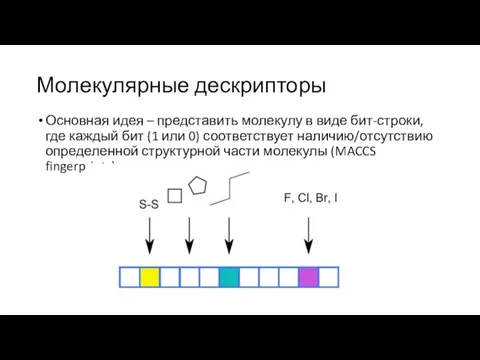
(1 или 0) соответствует наличию/отсутствию определенной структурной части молекулы (MACCS fingerprints)
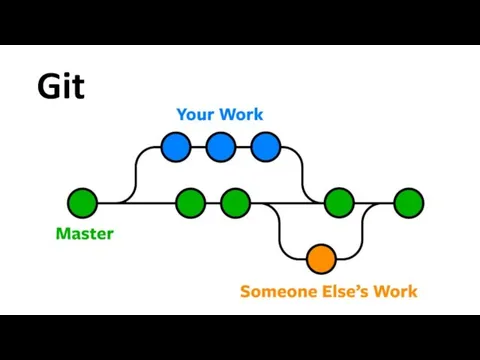
Слайд 27Git
1. Регистрируемся на GitHub
2. Создаем репозиторий
3. Скачиваем Git на РС
4. Логинимся
5. Клонируем

репозиторий (git clone)
6. Копируем файлы проекта в репозиторий на РС
7. Проверяем статус (git status)
8. Добавляем файлы (подготовка коммита) (git add .)
9. Создаем коммит (git commit –m ‘initial commit’)
10. git push














 Томография как способ бросить курить
Томография как способ бросить курить Неонатальный период (период новорожденности) у мелких животных
Неонатальный период (период новорожденности) у мелких животных Сердечно-легочная реанимация
Сердечно-легочная реанимация Общая фармакология. Фармакодинамика
Общая фармакология. Фармакодинамика Учение об иммунитете
Учение об иммунитете Дәстүрлі медицинаның концепциясы
Дәстүрлі медицинаның концепциясы Сердце. Кровеносные сосуды
Сердце. Кровеносные сосуды 10 видов полезных орехов
10 видов полезных орехов Физиология выделений. Функции почек
Физиология выделений. Функции почек Комплекс упражнений при остеохондрозе позвоночника различных возрастных групп
Комплекс упражнений при остеохондрозе позвоночника различных возрастных групп Роль акушерки в просвещении беременных и родильниц о преимуществах грудного вскармливания
Роль акушерки в просвещении беременных и родильниц о преимуществах грудного вскармливания Ольха (Alnus)
Ольха (Alnus) Метоика кормения тяжелобольного пациента
Метоика кормения тяжелобольного пациента Малярия
Малярия Выделение. Половая система
Выделение. Половая система Болезнь Паркинсона
Болезнь Паркинсона Клинический случай. Хронический дуоденит с выраженной гиперрегенераторной атрофией
Клинический случай. Хронический дуоденит с выраженной гиперрегенераторной атрофией Риск формирования перитонеальных спаек у детей с фенотипическими признаками дисплазии соединительной ткани
Риск формирования перитонеальных спаек у детей с фенотипическими признаками дисплазии соединительной ткани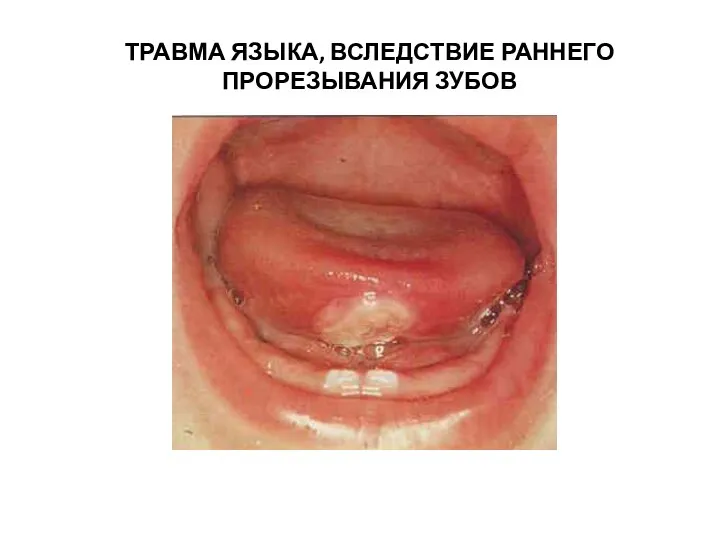 Травма языка вследствие раннего прорезывания зубов
Травма языка вследствие раннего прорезывания зубов Наш путь к протоколу лечения сепсиса
Наш путь к протоколу лечения сепсиса Хронический пиелонефрит
Хронический пиелонефрит Блокаторы лейкотриеновых рецепторов при БА и заболеваниях, сопровождаемых аллергическими реакциями
Блокаторы лейкотриеновых рецепторов при БА и заболеваниях, сопровождаемых аллергическими реакциями Ревматоидный артрит (РА)
Ревматоидный артрит (РА) Профилактика covid-19
Профилактика covid-19 Sovremennye_aspekty_vybora_i_primenenia_sredstv_dlya_narkoza
Sovremennye_aspekty_vybora_i_primenenia_sredstv_dlya_narkoza Неспецифічні запальні захворювання ЖСО
Неспецифічні запальні захворювання ЖСО Атопический дерматит. Базовый уход за кожей
Атопический дерматит. Базовый уход за кожей Переломы голени
Переломы голени