Содержание
- 2. Цель: поиск новых ингибиторов гепараназы на основе построенной модели фармакофора Задачи: 1) Провести скрининг соединений, обладающих
- 3. Материалы и методы.
- 4. Результаты и их обсуждение. Для построения модели фармакофора использовалось 504 ингибитора гепараназы, найденные в базе данных
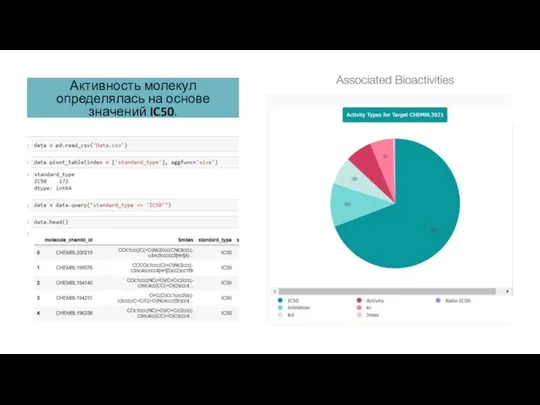
- 5. Активность молекул определялась на основе значений IC50.
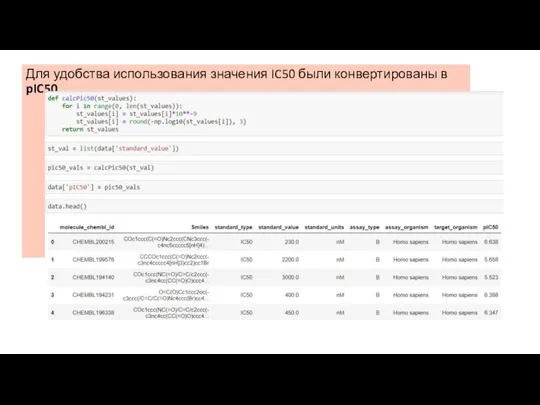
- 6. Для удобства использования значения IC50 были конвертированы в pIC50.
- 7. Активными считались соединения со значением pIC50 ≥ 6.0. Их количество составило 65.
- 8. Далее с помощью алгоритма кластеризации Butina наиболее структурно схожие соединения были объединены в 6 кластеров, из
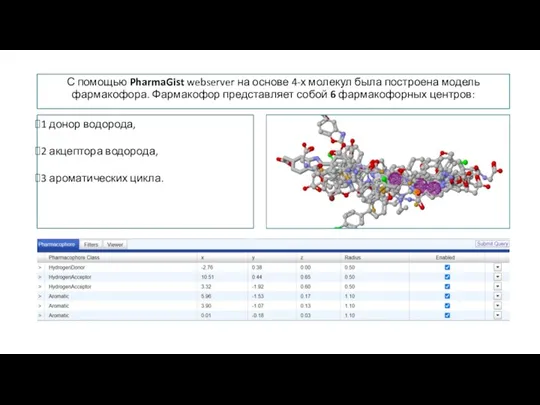
- 9. С помощью PharmaGist webserver на основе 4-х молекул была построена модель фармакофора. Фармакофор представляет собой 6
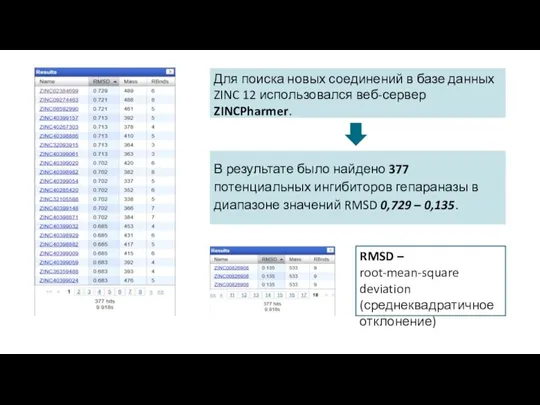
- 10. В результате было найдено 377 потенциальных ингибиторов гепараназы в диапазоне значений RMSD 0,729 – 0,135. Для
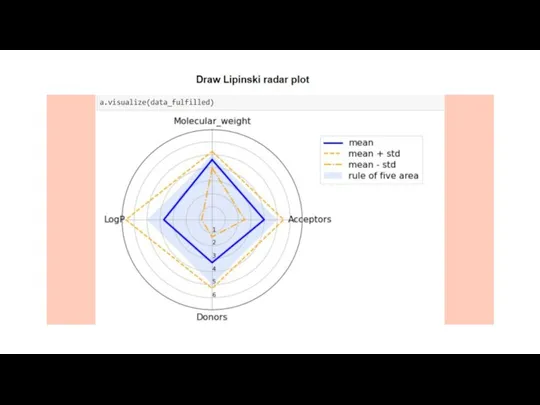
- 11. Соединения были отобраны в соответствии с правилом Липинского.
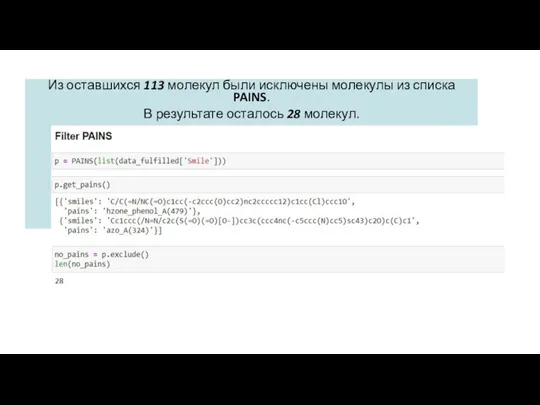
- 13. Из оставшихся 113 молекул были исключены молекулы из списка PAINS. В результате осталось 28 молекул.
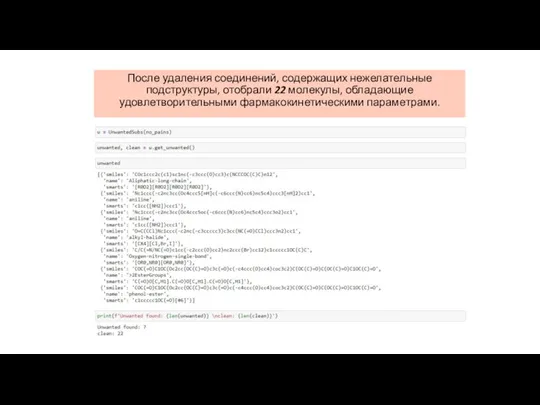
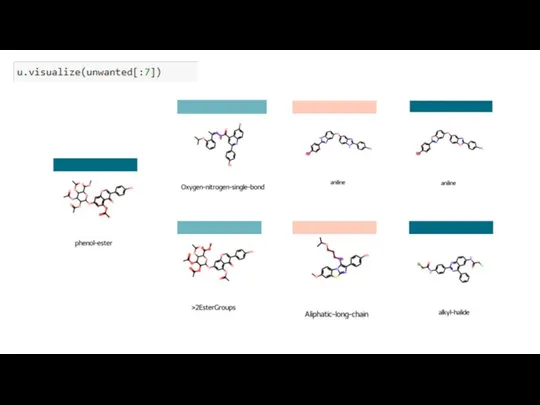
- 14. После удаления соединений, содержащих нежелательные подструктуры, отобрали 22 молекулы, обладающие удовлетворительными фармакокинетическими параметрами.
- 16. Выводы.
- 18. Скачать презентацию






 Хранение и транспортировка мфтв
Хранение и транспортировка мфтв Частная психопатология
Частная психопатология Гимнастика для глаз Облако
Гимнастика для глаз Облако Жіті тыныс жетіспеушілігі
Жіті тыныс жетіспеушілігі Удивительная жидкость - кровь
Удивительная жидкость - кровь Будь здоров без докторов!
Будь здоров без докторов! Кишечные инфекции
Кишечные инфекции Методики приготовления гистологических препаратов
Методики приготовления гистологических препаратов Миология. Мышца как орган. Строение, форма и классификация мышц
Миология. Мышца как орган. Строение, форма и классификация мышц Закаливание младшего школьника: ходьба босиком
Закаливание младшего школьника: ходьба босиком KhPN
KhPN Генитоуринарный менопаузальный синдром
Генитоуринарный менопаузальный синдром Типовые операции на прямой кишке
Типовые операции на прямой кишке Диффузная нейроэндокринная система энтероэндокринные клетки
Диффузная нейроэндокринная система энтероэндокринные клетки Методы определения функционального состояния зубочелюстной системы
Методы определения функционального состояния зубочелюстной системы Genetically modified organisms - salvation for the poor and hungry or something poisonous and harmful for people?
Genetically modified organisms - salvation for the poor and hungry or something poisonous and harmful for people? Табак. Общий вред табакокурения
Табак. Общий вред табакокурения Регенерация желудка
Регенерация желудка НОРМАТИВНО-ПРАВОВОЕ ОБЕСПЕЧЕНИЕ ДЕЯТЕЛЬНОСТИ УЧИТЕЛЯ-ЛОГОПЕДА ОБЩЕОБРАЗОВАТЕЛЬНОЙ ШКОЛЫ
НОРМАТИВНО-ПРАВОВОЕ ОБЕСПЕЧЕНИЕ ДЕЯТЕЛЬНОСТИ УЧИТЕЛЯ-ЛОГОПЕДА ОБЩЕОБРАЗОВАТЕЛЬНОЙ ШКОЛЫ Жүрек ісіктері
Жүрек ісіктері Роль полноценного сна в жизни ребенка
Роль полноценного сна в жизни ребенка Отслойка сетчатки
Отслойка сетчатки Гигиеническое значение температуры, влажности, скорости и направления движения воздуха. Их влияние на организм человека
Гигиеническое значение температуры, влажности, скорости и направления движения воздуха. Их влияние на организм человека Плод, как объект родов
Плод, как объект родов Раны. Первая помощь
Раны. Первая помощь Токсикологическая служба и токсикологическая ситуация в Республике Беларусь
Токсикологическая служба и токсикологическая ситуация в Республике Беларусь Боль. Наркотические анальгетики
Боль. Наркотические анальгетики Диагностический подход к пациентам с солидными новообразованиями
Диагностический подход к пациентам с солидными новообразованиями