Содержание
- 2. Журнальный клуб 18 марта: Jimenez-Sanchez G, Childs B, Valle D. Human disease genes. Nature. 2001 Feb
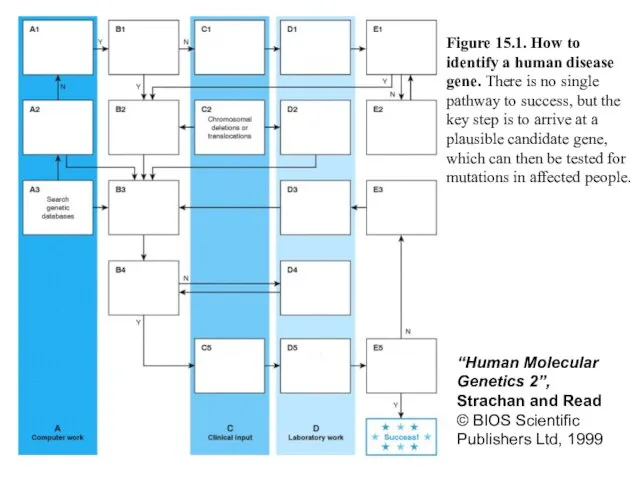
- 3. Figure 15.1. How to identify a human disease gene. There is no single pathway to success,
- 4. Стратегии поиска генов заболеваний Functional/Candidate Gene Cloning: “Either a known protein that is responsible for an
- 5. Сложные заболевания: примеры Аутизм Астма Диабет Ожирение Алкоголизм Гипертония Шизофрения
- 6. Особенности сложных заболеваний Неполная пенетрантность (Не всякая генетическая предрасположенность проявляется как болезнь) Различный возраст развития заболевания
- 8. Evolution revisited: упрощенный взгляд на болезнетворные аллели Моногенные заболевания Сильный эффект аллелей Мутационно-отборное равновесие Сложные заболевания
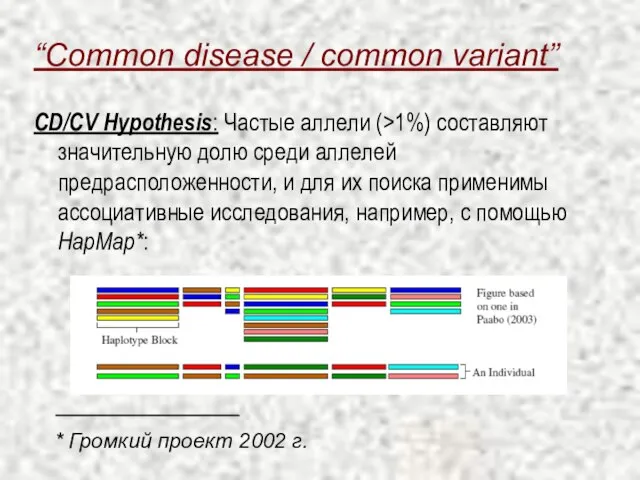
- 9. “Common disease / common variant” CD/CV Hypothesis: Частые аллели (>1%) составляют значительную долю среди аллелей предрасположенности,
- 10. “Common disease / rare variant” CD/RV Hypothesis: Редкие аллели ( * Громкий проект 2008 г.
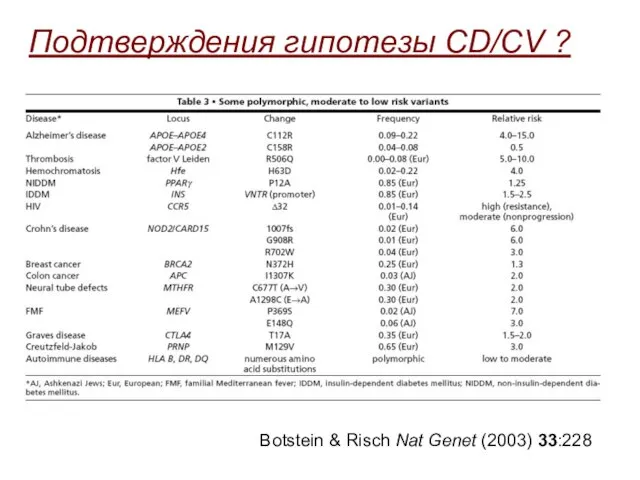
- 11. Botstein & Risch Nat Genet (2003) 33:228 Подтверждения гипотезы CD/CV ?
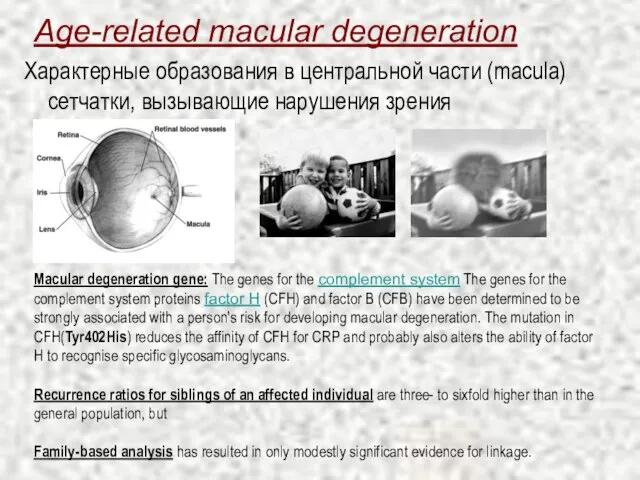
- 12. Age-related macular degeneration Характерные образования в центральной части (macula) сетчатки, вызывающие нарушения зрения Macular degeneration gene:
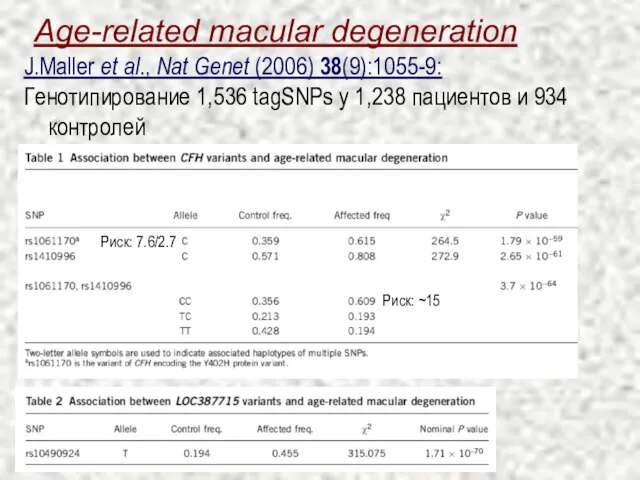
- 13. Age-related macular degeneration J.Maller et al., Nat Genet (2006) 38(9):1055-9: Генотипирование 1,536 tagSNPs у 1,238 пациентов
- 14. Age-related macular degeneration J.Maller et al., Nat Genet (2006) 38(9):1055-9: Полиморфизм в локусе, содержащем гены системы
- 15. Age-related macular degeneration
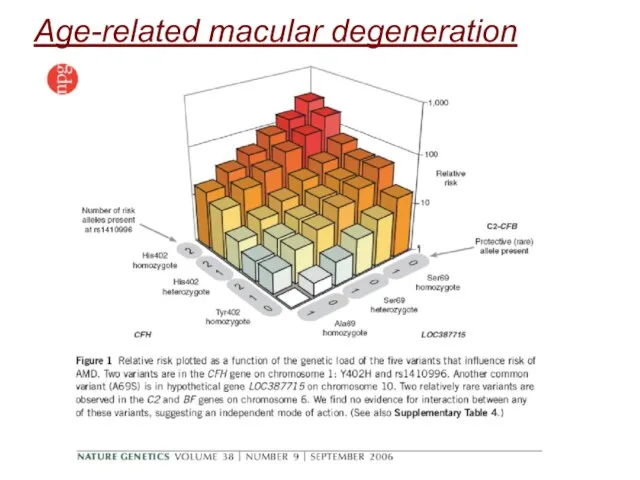
- 16. Age-related macular degeneration (i) there can exist common alleles of substantial effect on common disease (in
- 17. Влияние редких аллелей на уровень холестерина высоко й плотности Низкой уровень холестерина высокой плотности (HDL-C, «хорошего»
- 18. Влияние редких аллелей на уровень холестерина высоко й плотности J.Cohen et al., Science (2004) 305:869-72 The
- 20. Скачать презентацию










 КОММЕРЧЕСКОЕ ПРЕДЛОЖЕНИЕ
КОММЕРЧЕСКОЕ ПРЕДЛОЖЕНИЕ Презентация на тему Решение задач по нахождению величин по сумме и разности
Презентация на тему Решение задач по нахождению величин по сумме и разности Представленные ниже ребусы можно использовать на уроках ОБЖ или внеклассных мероприятиях, при изучении правил дорожного движения
Представленные ниже ребусы можно использовать на уроках ОБЖ или внеклассных мероприятиях, при изучении правил дорожного движения Окорка фанерного сырья
Окорка фанерного сырья ggg
ggg «Дорога добра»
«Дорога добра» Cтекло лаб (1)
Cтекло лаб (1) Технологии поддержки принятия решений.
Технологии поддержки принятия решений. Презентация на тему Устойчивые выражения с глаголом to be
Презентация на тему Устойчивые выражения с глаголом to be Рисунок цилиндра и куба
Рисунок цилиндра и куба Презентация на тему Основные характеристики персонального компьютера
Презентация на тему Основные характеристики персонального компьютера  Содержание тренажера Рукодельница
Содержание тренажера Рукодельница Способности человека
Способности человека Организация деятельности управляющего проектом строительства и взаимодействие с ключевыми участниками проекта.Опыт работы с кр
Организация деятельности управляющего проектом строительства и взаимодействие с ключевыми участниками проекта.Опыт работы с кр Правосознание и его деформации
Правосознание и его деформации Делопроизводство. Организационные документы
Делопроизводство. Организационные документы Шаблон презентации для инициативной разработки
Шаблон презентации для инициативной разработки Последствия парникового эффекта для Земли
Последствия парникового эффекта для Земли Святые Земли Ростовской
Святые Земли Ростовской Биоклиматическая архитектура
Биоклиматическая архитектура Роль системы комплаенс в управлении рисками организации
Роль системы комплаенс в управлении рисками организации Cущинская Светлана
Cущинская Светлана ВИП Консульт
ВИП Консульт Проблема формирования и использования финансового потенциала фирмы
Проблема формирования и использования финансового потенциала фирмы «Купить или не купить, вот в чем вопрос…»или проблема приобретения программного обеспечения для домашнего компьютера.
«Купить или не купить, вот в чем вопрос…»или проблема приобретения программного обеспечения для домашнего компьютера. Funktsionalnaya_Asimmetria_golovnogo_mozga1_Zhuchkov
Funktsionalnaya_Asimmetria_golovnogo_mozga1_Zhuchkov Автоматизированнай пульсодиагностический комплекс (АПДК)
Автоматизированнай пульсодиагностический комплекс (АПДК) Молодежный совет как образ мышления
Молодежный совет как образ мышления