Содержание
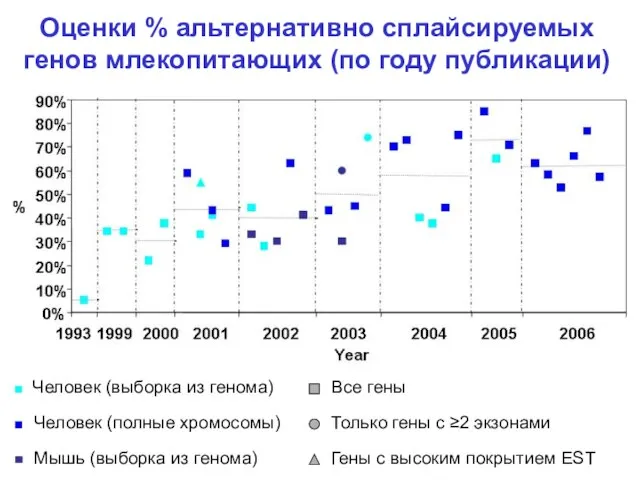
- 2. Оценки % альтернативно сплайсируемых генов млекопитающих (по году публикации) Человек (выборка из генома) Человек (полные хромосомы)
- 3. Значение альтернативного сплайсинга Функциональное: поддержание белкового разнообразия Человек: ~30.000 генов, >100.000 белков поддержание белкового единообразия Например,
- 4. Эволюция альтернативной экзон-интронной структуры млекопитающие: человек, мышь, собака двукрылые насекомые: Drosophila melanogaster, D. pseudoobscura, Anopheles gambiae
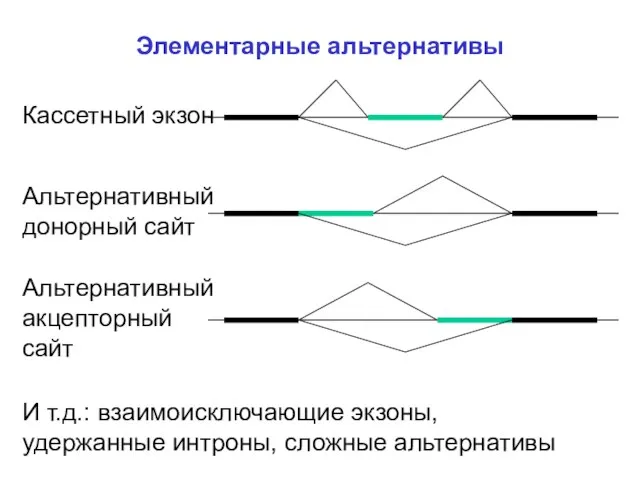
- 5. Элементарные альтернативы Кассетный экзон Альтернативный донорный сайт Альтернативный акцепторный сайт И т.д.: взаимоисключающие экзоны, удержанные интроны,
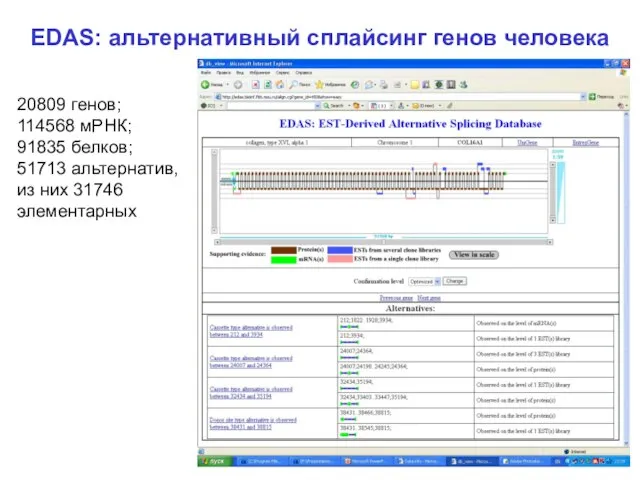
- 6. EDAS: альтернативный сплайсинг генов человека 20809 генов; 114568 мРНК; 91835 белков; 51713 альтернатив, из них 31746
- 7. Альтернативная экзон-интроная структура генов млекопитающих Тройки ортологичных генов: человек-мышь-собака Следим за судьбой (консервативностью) альтернатив человека в
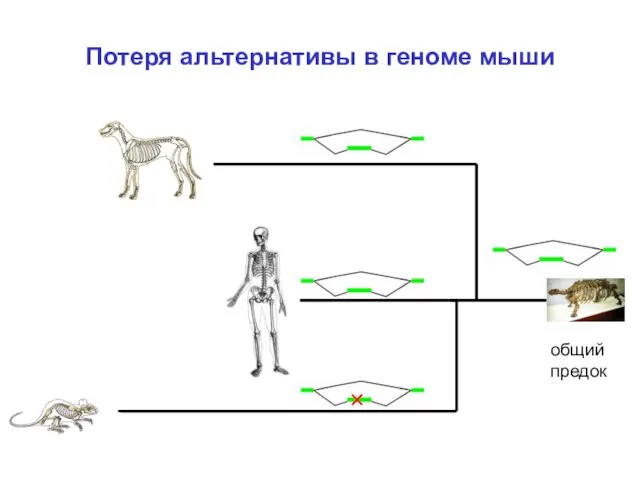
- 8. Потеря альтернативы в геноме мыши общий предок
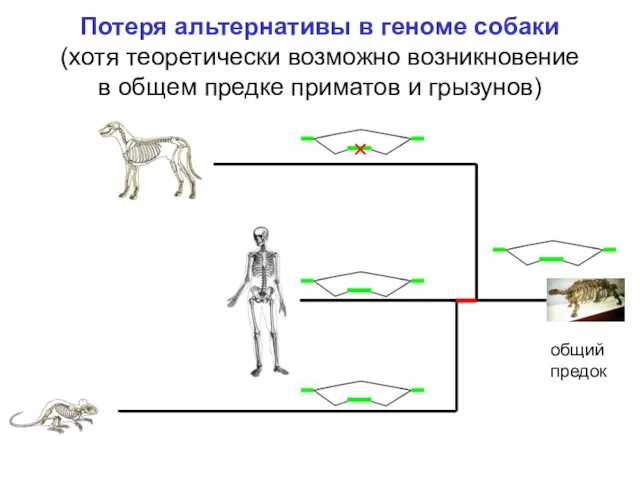
- 9. Потеря альтернативы в геноме собаки (хотя теоретически возможно возникновение в общем предке приматов и грызунов) общий
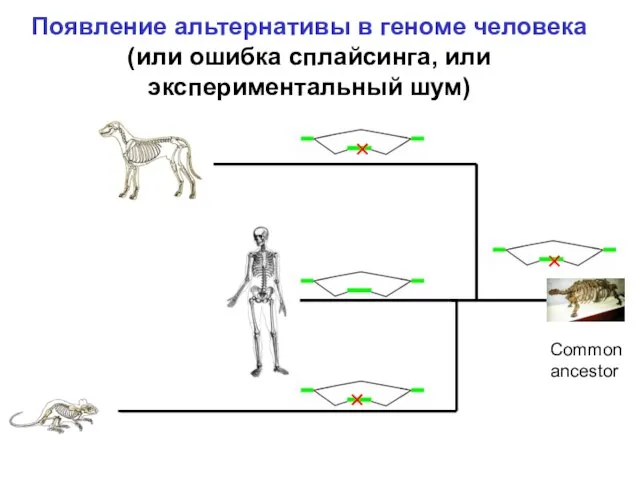
- 10. Появление альтернативы в геноме человека (или ошибка сплайсинга, или экспериментальный шум) Common ancestor
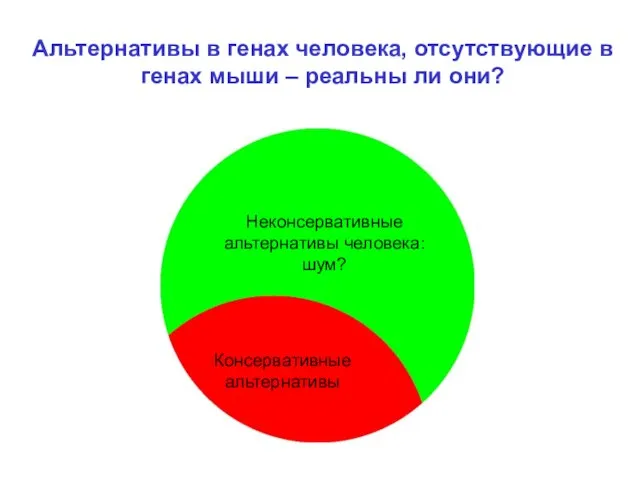
- 11. Неконсервативные альтернативы человека: шум? Консервативные альтернативы Альтернативы в генах человека, отсутствующие в генах мыши – реальны
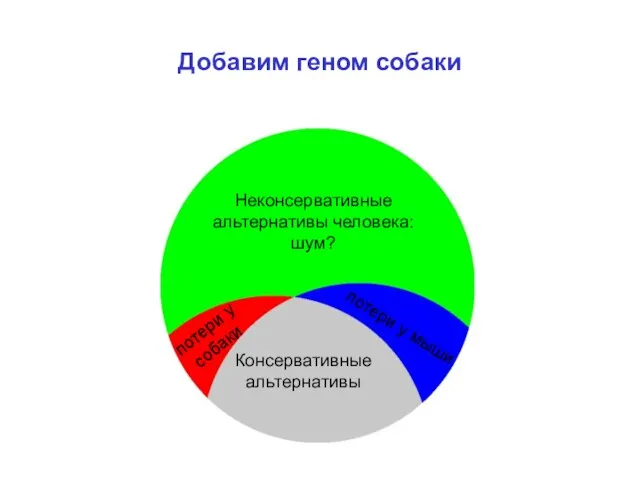
- 12. Human-specific alternatives: noise? Conserved alternatives Добавим геном собаки Консервативные альтернативы потери у собаки потери у мыши
- 13. Наблюдения Часто вставляемые экзоны консервативны независимо от того, сбивают ли они рамку Редко вставляемые экзоны менее
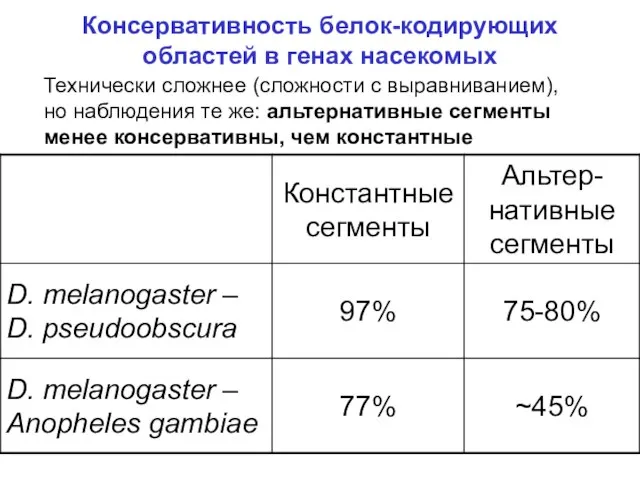
- 14. Консервативность белок-кодирующих областей в генах насекомых Технически сложнее (сложности с выравниванием), но наблюдения те же: альтернативные
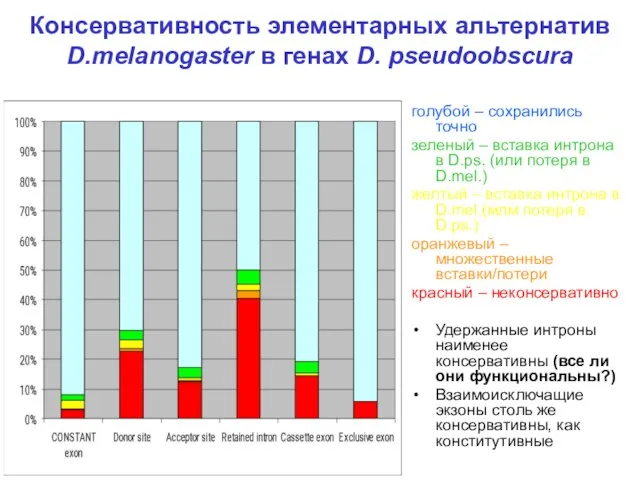
- 15. Консервативность элементарных альтернатив D.melanogaster в генах D. pseudoobscura голубой – сохранились точно зеленый – вставка интрона
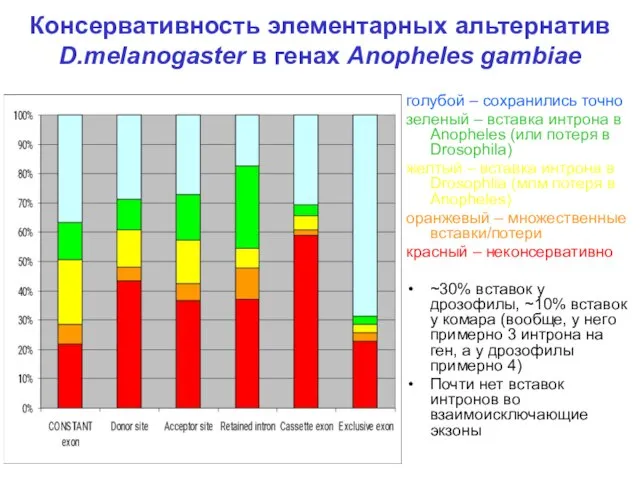
- 16. Консервативность элементарных альтернатив D.melanogaster в генах Anopheles gambiae голубой – сохранились точно зеленый – вставка интрона
- 17. Скорость эволюции и тип отбора в альтернативных и константных областях Пары ортологичных генов человек и мышь
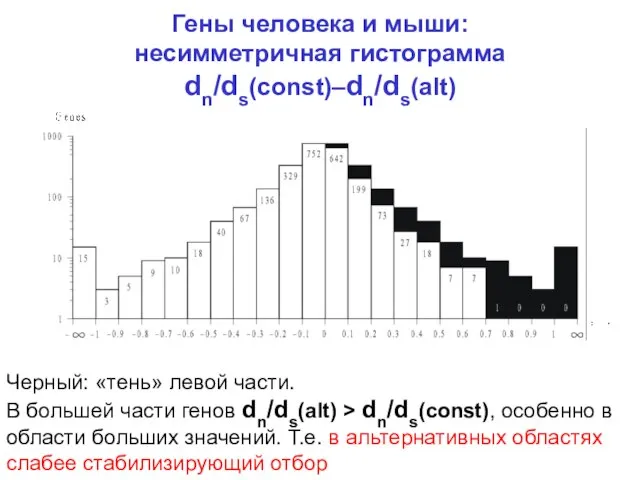
- 18. Гены человека и мыши: несимметричная гистограмма dn/ds(const)–dn/ds(alt) Черный: «тень» левой части. В большей части генов dn/ds(alt)
- 19. Гены Drosophila: слабее отбор в альтернативных областях? Больше замен в альт. обл. Равный уровень замен Больше
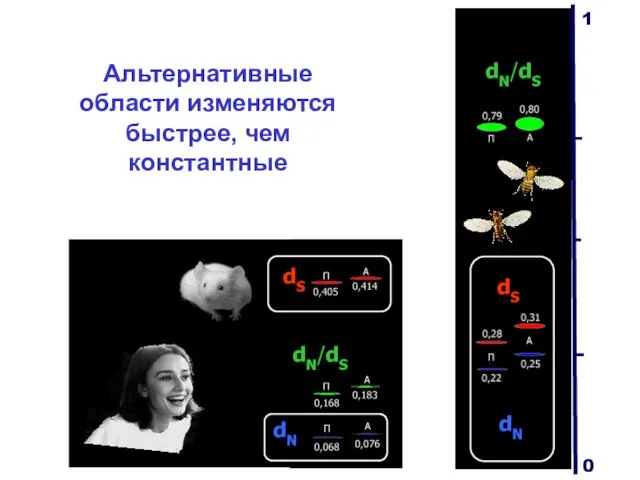
- 20. Альтернативные области изменяются быстрее, чем константные dN dN/dS dS dN/dS dS dN 1 0
- 21. Ослабление стабилизирующего отбора в альтернативных областях dN/dS dN dS dN/dS dS dN 1 0
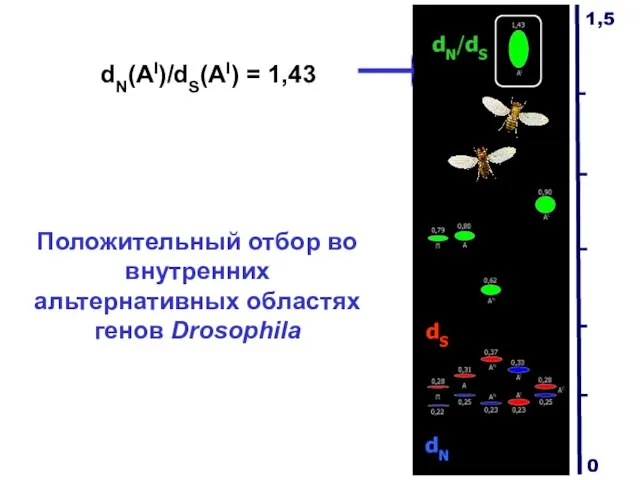
- 22. Положительный отбор во внутренних альтернативных областях генов Drosophila dN/dS dS dN dN(AI)/dS(AI) = 1,43 1,5 0
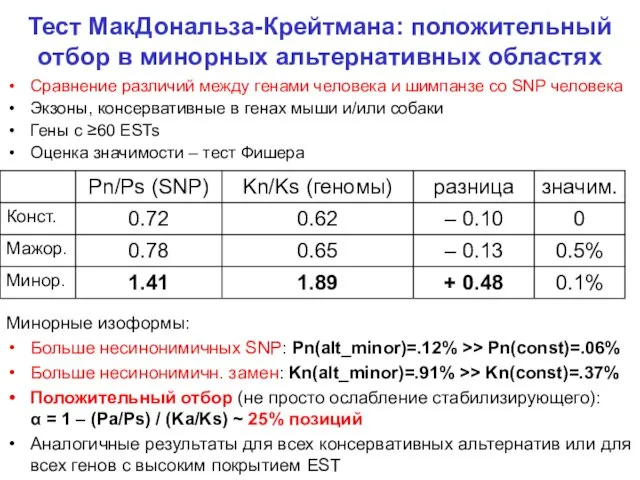
- 23. Тест МакДональза-Крейтмана: положительный отбор в минорных альтернативных областях Сравнение различий между генами человека и шимпанзе со
- 24. Попытка синтеза Альтернативный сплайсинг часто видоспецифичен «молодые» альтернативные изоформы часто тканеспецифичны … но все же функционален
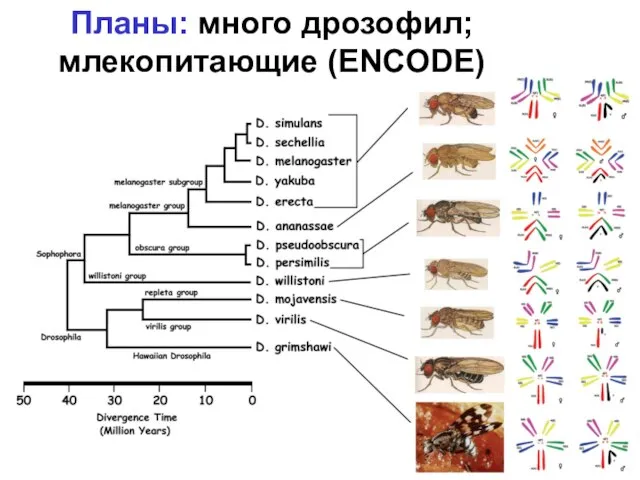
- 25. Планы: много дрозофил; млекопитающие (ENCODE)
- 26. Что делать? Оценить не только скорость потерь альтернатив, но и скорость приобретений (отличая молодые изоформы от
- 27. Благодарности Обсуждения Евгений Кунин (NCBI) Игорь Рогозин (NCBI) Всеволод Макеев (ГосНИИГенетика) Дмитрий Петров (Stanford) Дмитрий Фришман
- 28. Авторы Андрей Миронов (МГУ, ИППИ) Рамиль Нуртдинов (МГУ) – человек/мышь/собака Дмитрий Малько (ГосНИИГенетика) – дрозофилы/комар Екатерина
- 29. Основные публикации 10,1 D.B.Malko, V.J.Makeev, A.A.Mironov, M.S.Gelfand (2006) Evolution of the exon-intron structure and alternative splicing
- 31. Скачать презентацию












 Презентация
Презентация  Декоративные швы
Декоративные швы Пешеходный туризм
Пешеходный туризм Нормы и нормативы расхода материальных ресурсов в разработке нефтяных месторождений
Нормы и нормативы расхода материальных ресурсов в разработке нефтяных месторождений Спортивные игры. Волейбол
Спортивные игры. Волейбол Олимпийские игры – как фактор развития международного бизнеса
Олимпийские игры – как фактор развития международного бизнеса Потоки платежей
Потоки платежей "Предварительные установки, или Организационные рамки семинара"
"Предварительные установки, или Организационные рамки семинара" Аграрная реформа П.А.Столыпина. Крах? Небывалый успех? Незавершенный характер?
Аграрная реформа П.А.Столыпина. Крах? Небывалый успех? Незавершенный характер? Как изменить цветовой оттенок фонового рисунка
Как изменить цветовой оттенок фонового рисунка Цель урока:
Цель урока: Алкалоиды
Алкалоиды История джинсов
История джинсов Понятие информационной системы (ИС). Классификация ИС
Понятие информационной системы (ИС). Классификация ИС ООО НТЦ Безопасность. Несчастные случаи
ООО НТЦ Безопасность. Несчастные случаи ГИА-2012
ГИА-2012 Раздел Введение в вычислительные системы и сетевую обработку данных
Раздел Введение в вычислительные системы и сетевую обработку данных Front, side, console
Front, side, console Стили семейного воспитания.
Стили семейного воспитания. Бухгалтерский учет, анализ и аудит
Бухгалтерский учет, анализ и аудит Семья Столыпина П.А.
Семья Столыпина П.А. Типы парламентов и их влияние на законотворческий процесс. Общая структура и назначение
Типы парламентов и их влияние на законотворческий процесс. Общая структура и назначение Строительство жилого загородного дома проект № лси-155-кпнф
Строительство жилого загородного дома проект № лси-155-кпнф Материальная помощь. Основные положения. Правила оформления
Материальная помощь. Основные положения. Правила оформления ВАШ ИДЕАЛЬНЫЙ ЗАВТРАК!
ВАШ ИДЕАЛЬНЫЙ ЗАВТРАК! М.Ю.Лермонтов. Страницы биографии
М.Ю.Лермонтов. Страницы биографии Как вести себя, если вы стали заложником террористов
Как вести себя, если вы стали заложником террористов Основные закономерности развития науки
Основные закономерности развития науки