Содержание
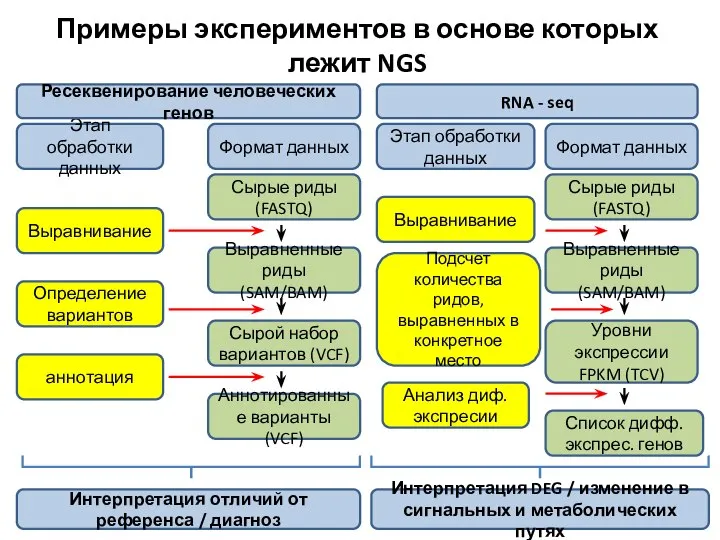
- 2. Примеры экспериментов в основе которых лежит NGS Этап обработки данных Формат данных Ресеквенирование человеческих генов RNA
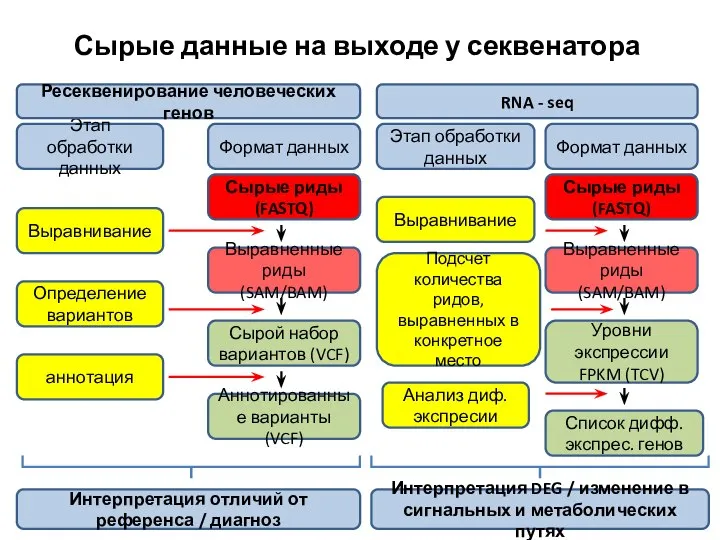
- 3. Сырые данные на выходе у секвенатора Этап обработки данных Формат данных Ресеквенирование человеческих генов RNA -
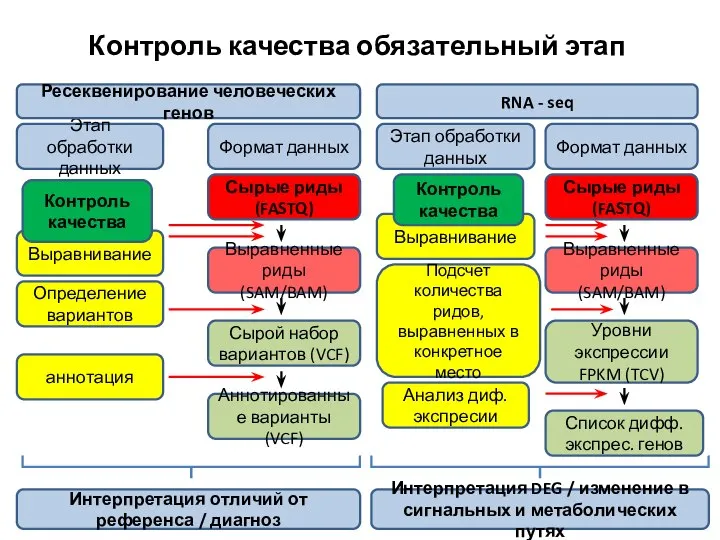
- 4. Контроль качества обязательный этап Этап обработки данных Формат данных Ресеквенирование человеческих генов RNA - seq Формат
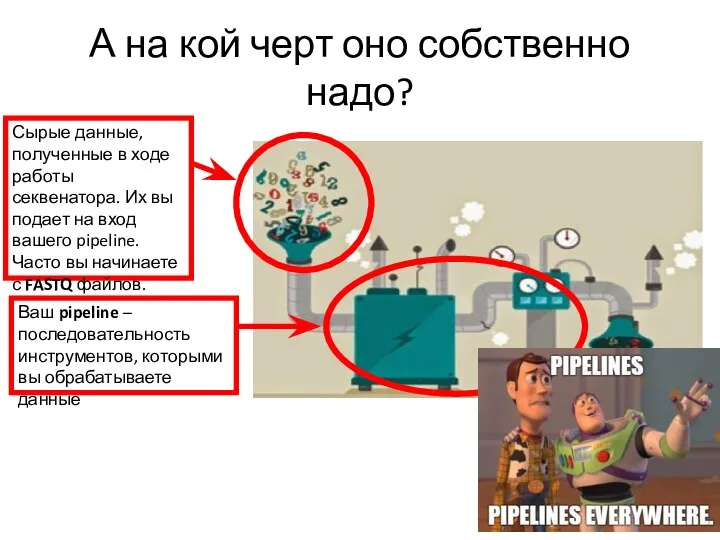
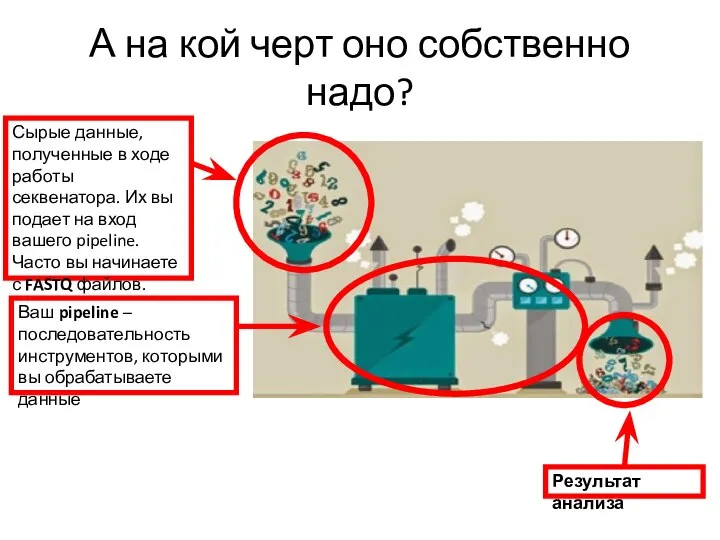
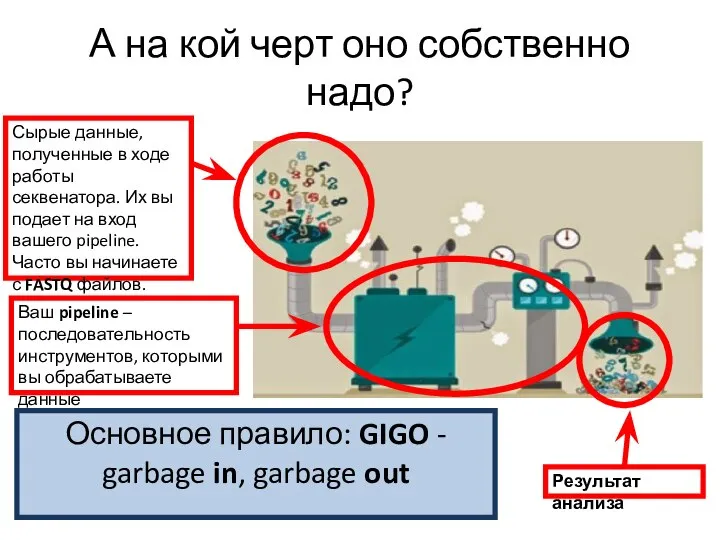
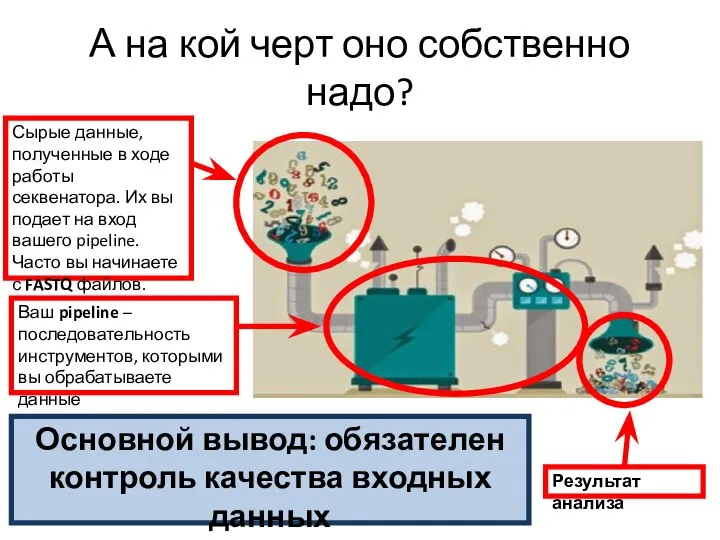
- 5. А на кой черт оно собственно надо?
- 6. А на кой черт оно собственно надо? Сырые данные, полученные в ходе работы секвенатора. Их вы
- 7. А на кой черт оно собственно надо? Сырые данные, полученные в ходе работы секвенатора. Их вы
- 8. А на кой черт оно собственно надо? Сырые данные, полученные в ходе работы секвенатора. Их вы
- 9. А на кой черт оно собственно надо? Сырые данные, полученные в ходе работы секвенатора. Их вы
- 10. А на кой черт оно собственно надо? Сырые данные, полученные в ходе работы секвенатора. Их вы
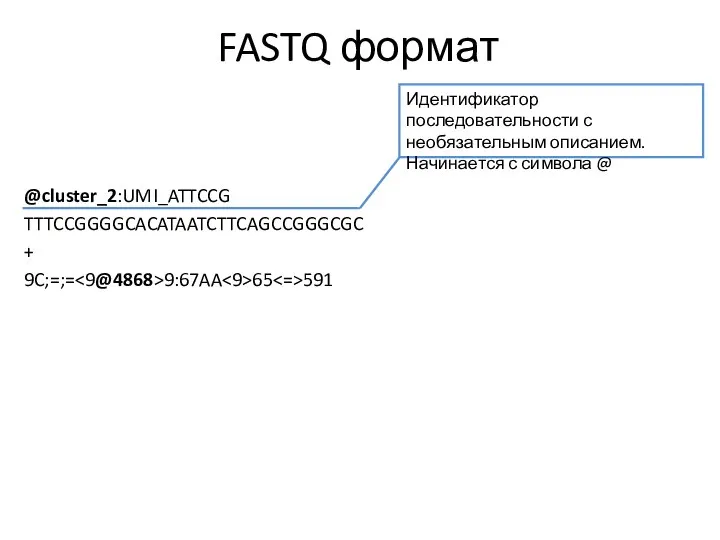
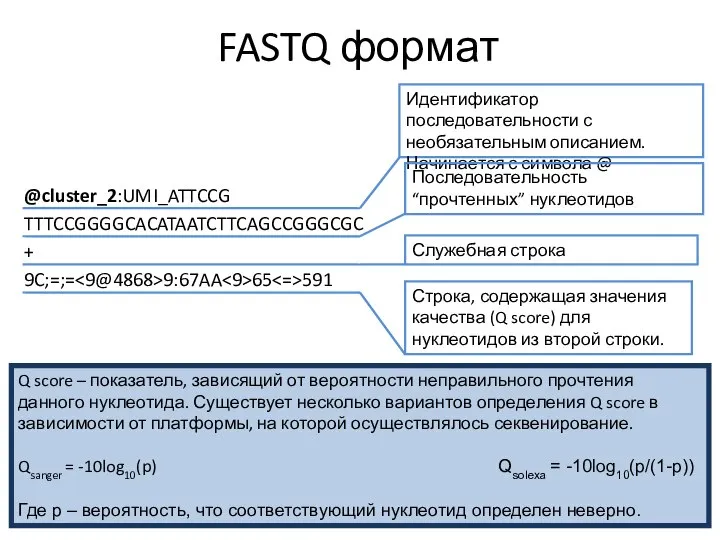
- 11. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591
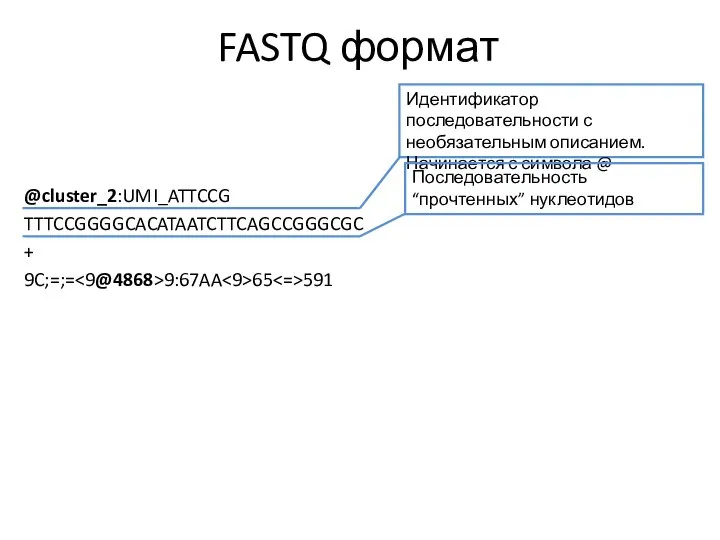
- 12. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591 Идентификатор последовательности с необязательным описанием. Начинается с
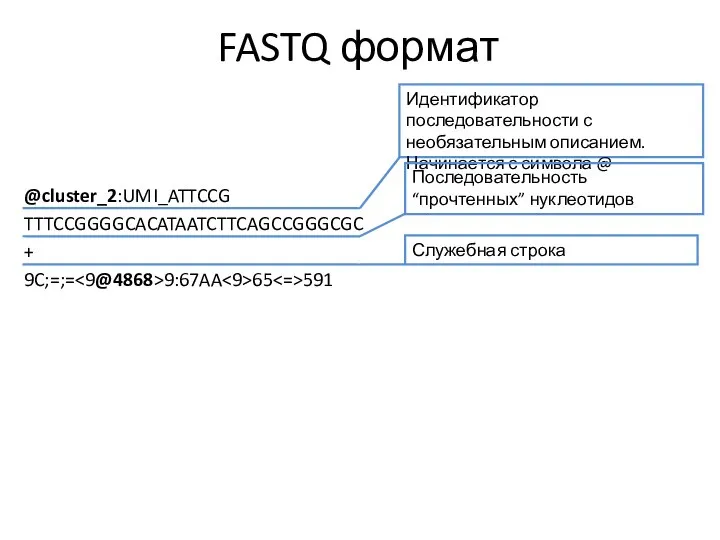
- 13. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591 Идентификатор последовательности с необязательным описанием. Начинается с
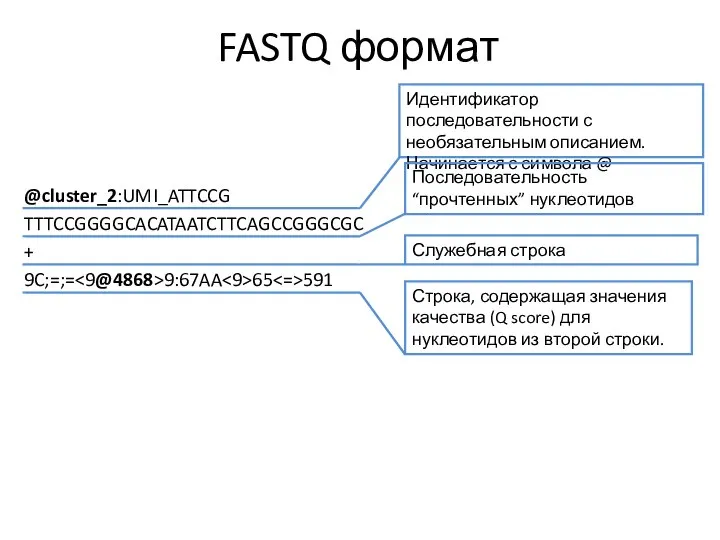
- 14. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591 Идентификатор последовательности с необязательным описанием. Начинается с
- 15. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591 Идентификатор последовательности с необязательным описанием. Начинается с
- 16. FASTQ формат @cluster_2:UMI_ATTCCG TTTCCGGGGCACATAATCTTCAGCCGGGCGC + 9C;=;= 9:67AA 65 591 Идентификатор последовательности с необязательным описанием. Начинается с
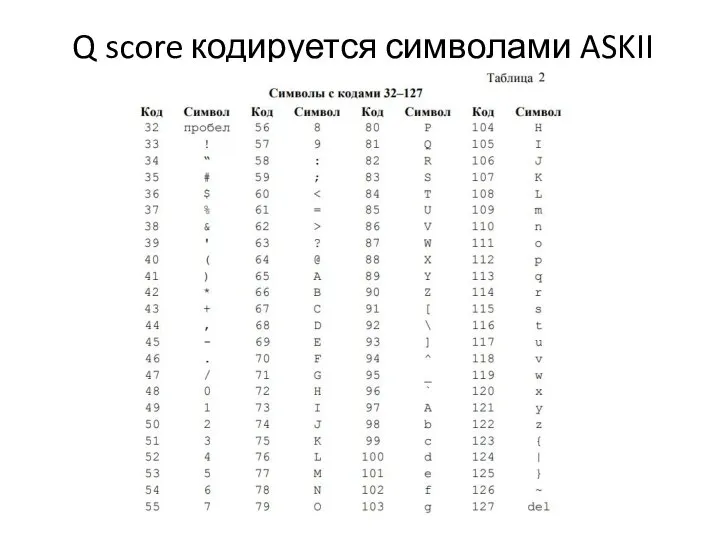
- 17. Q score кодируется символами ASKII
- 18. FastQC FastQC – инструмент, позволяющий проводить контроль качества сырых ридов. В настоящее время по сути стал
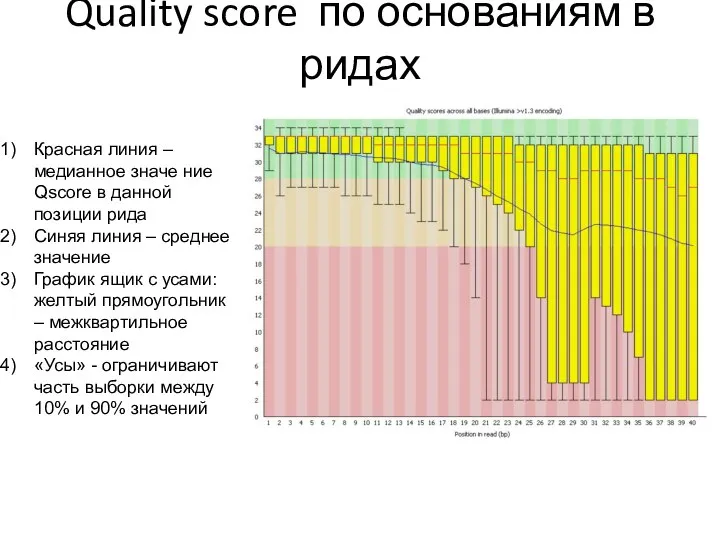
- 19. Quality score по основаниям в ридах Красная линия – медианное значе ние Qscore в данной позиции
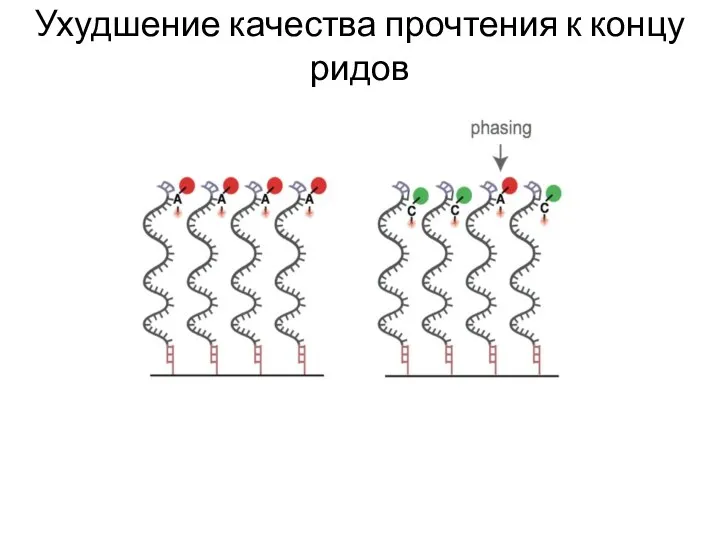
- 20. Ухудшение качества прочтения к концу ридов
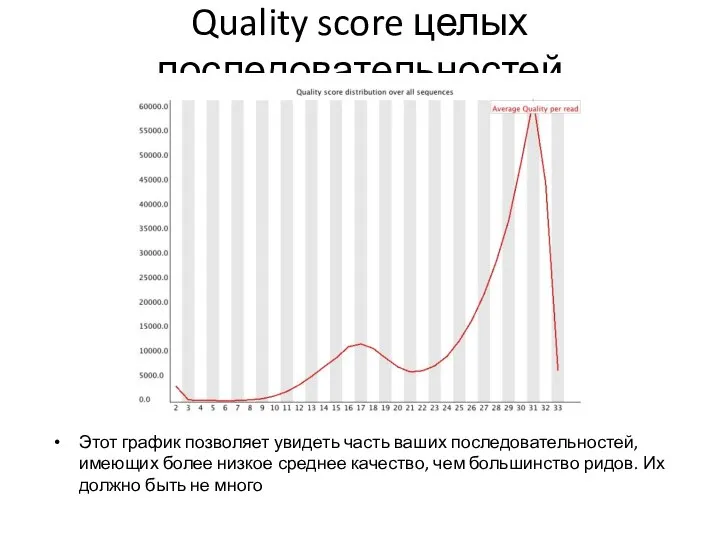
- 21. Quality score целых последовательностей Этот график позволяет увидеть часть ваших последовательностей, имеющих более низкое среднее качество,
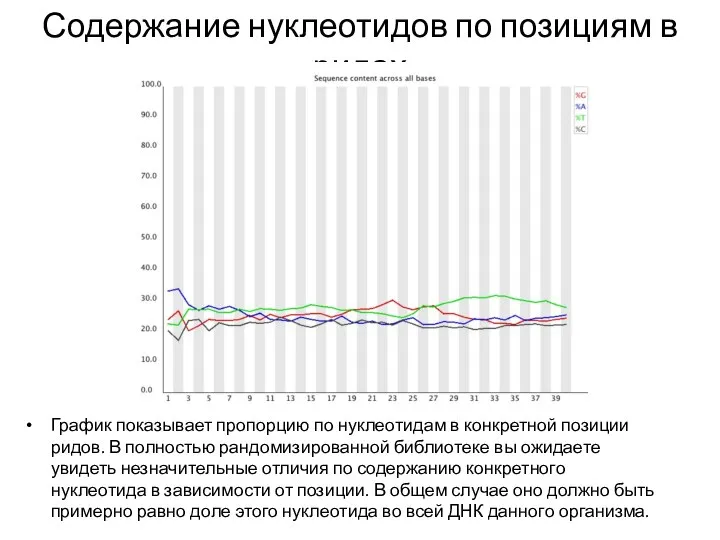
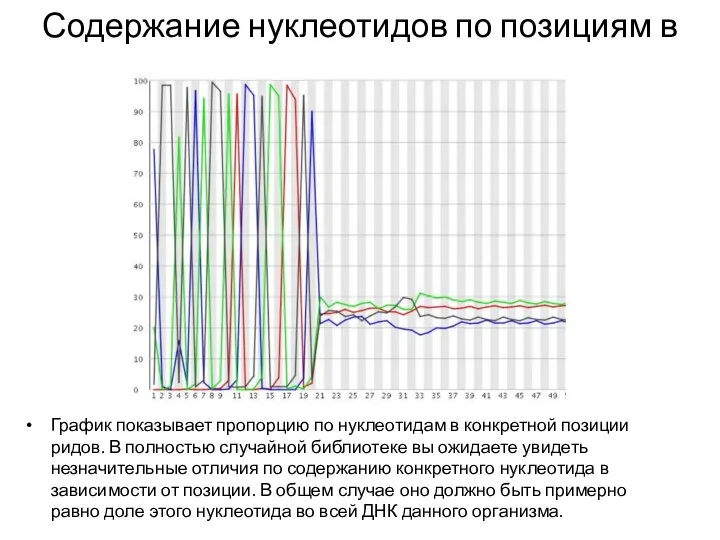
- 22. Содержание нуклеотидов по позициям в ридах График показывает пропорцию по нуклеотидам в конкретной позиции ридов. В
- 23. Содержание нуклеотидов по позициям в ридах График показывает пропорцию по нуклеотидам в конкретной позиции ридов. В
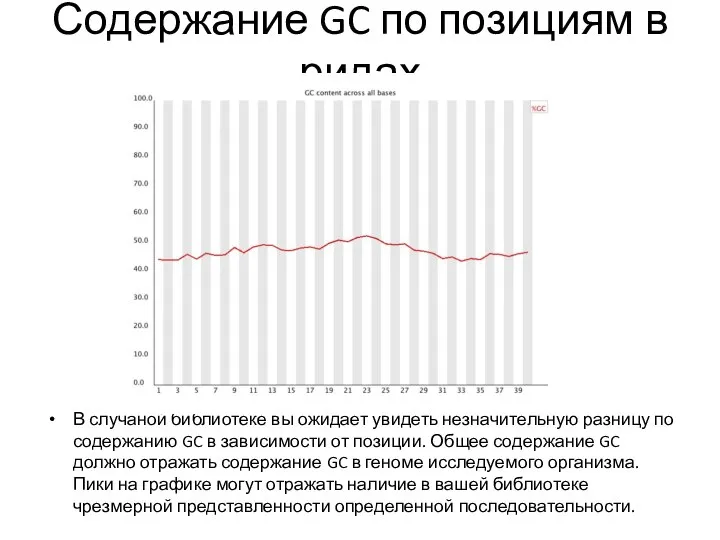
- 24. Содержание GC по позициям в ридах В случаной библиотеке вы ожидает увидеть незначительную разницу по содержанию
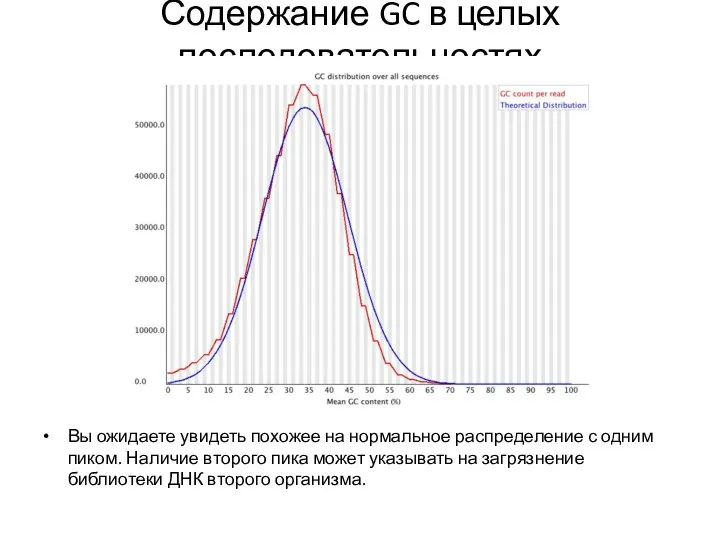
- 25. Содержание GC в целых последовательностях Вы ожидаете увидеть похожее на нормальное распределение с одним пиком. Наличие
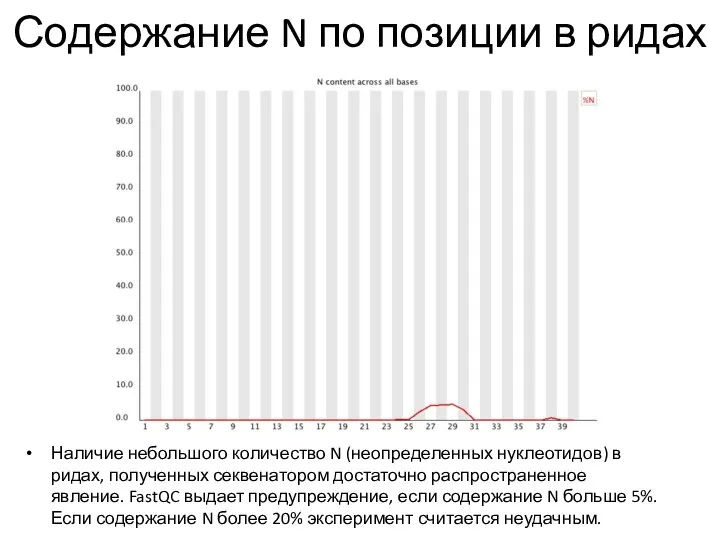
- 26. Содержание N по позиции в ридах Наличие небольшого количество N (неопределенных нуклеотидов) в ридах, полученных секвенатором
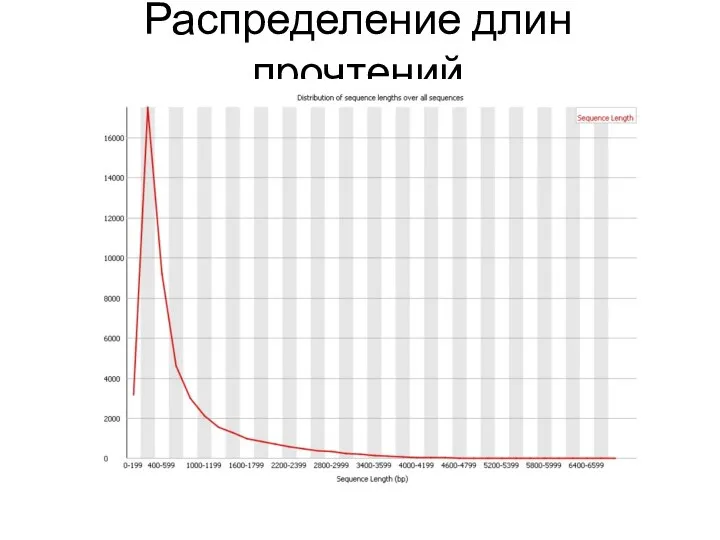
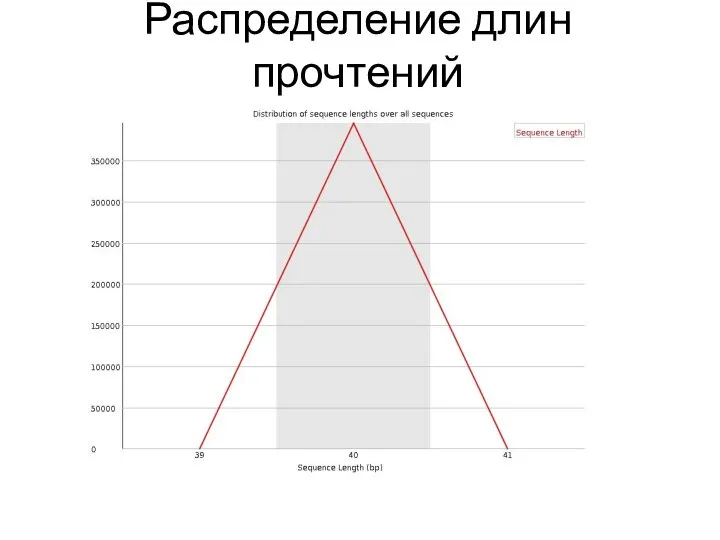
- 27. Распределение длин прочтений
- 28. Распределение длин прочтений
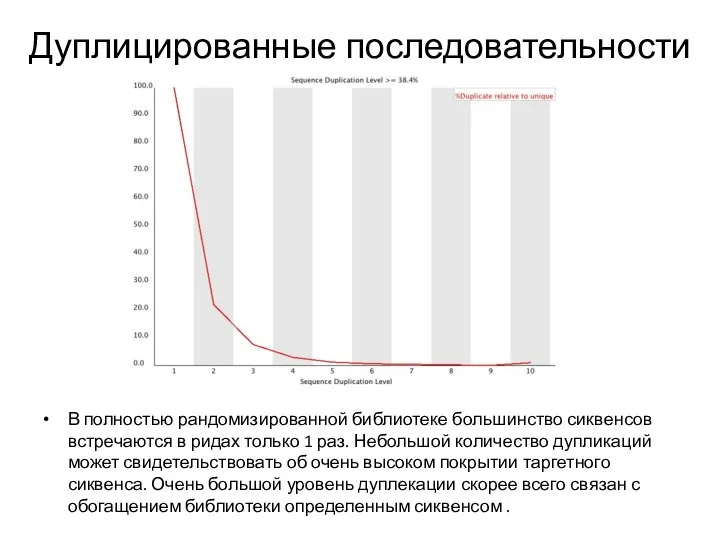
- 29. Дуплицированные последовательности В полностью рандомизированной библиотеке большинство сиквенсов встречаются в ридах только 1 раз. Небольшой количество
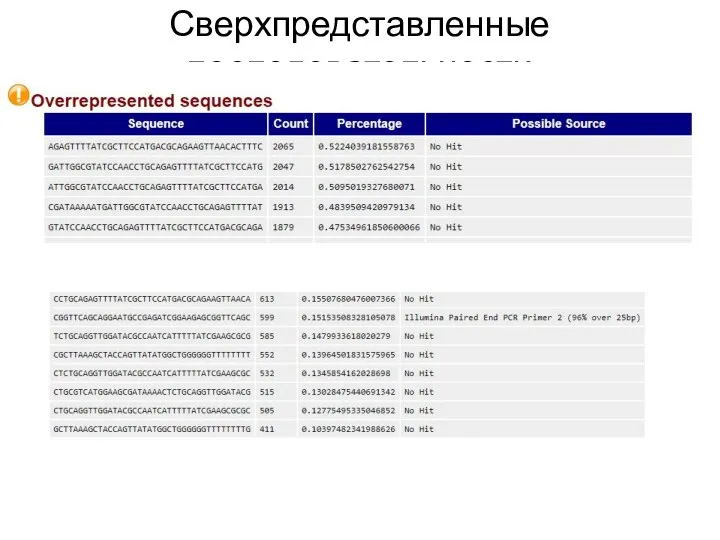
- 30. Сверхпредставленные последовательности Обычно библиотека для NGS содержит разнообразный набор последовательностей, без единственной последовательности, составляющая существенную часть
- 31. Сверхпредставленные последовательности
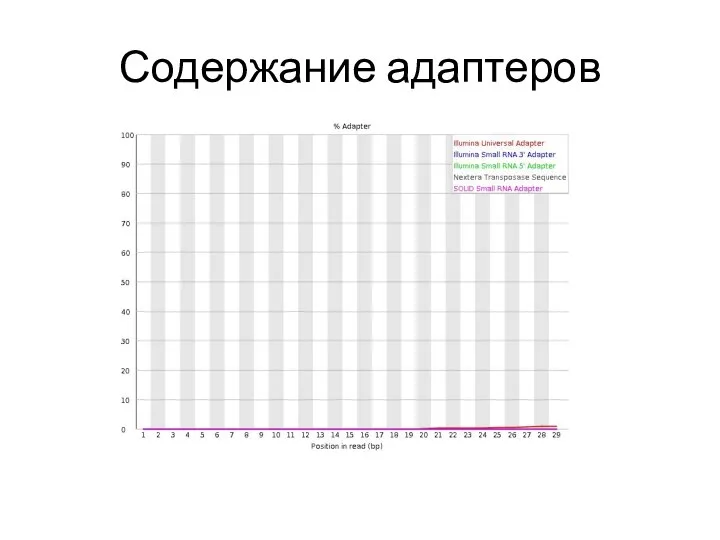
- 32. Содержание адаптеров
- 34. Скачать презентацию




 Prezentatsia_Microsoft_Office_PowerPoint_2007 (1)
Prezentatsia_Microsoft_Office_PowerPoint_2007 (1) Zberthyesit. Zinxhiri ushqimor
Zberthyesit. Zinxhiri ushqimor Ценность биоразнообразия для отдыха и туризма. Возможности экономической оценки
Ценность биоразнообразия для отдыха и туризма. Возможности экономической оценки Абиотический фактор – солнечная радиация
Абиотический фактор – солнечная радиация Гепатит А Это острое вирусное заболевание с преимущественным поражением печени, протекающее с интоксикацией, в ряде случаев сопр
Гепатит А Это острое вирусное заболевание с преимущественным поражением печени, протекающее с интоксикацией, в ряде случаев сопр Марал
Марал Превращения внутри нас
Превращения внутри нас Стебель, его строение и значение
Стебель, его строение и значение Нейро-гуморальная регуляция
Нейро-гуморальная регуляция Африканский страус
Африканский страус Мой любимый хомячок
Мой любимый хомячок Почему глаза не замерзают зимой. Познавательно-исследовательский проект дошкольника
Почему глаза не замерзают зимой. Познавательно-исследовательский проект дошкольника Онтогенез, морфология и физиология слухового анализатора
Онтогенез, морфология и физиология слухового анализатора Форма кустов при укрывной культуре
Форма кустов при укрывной культуре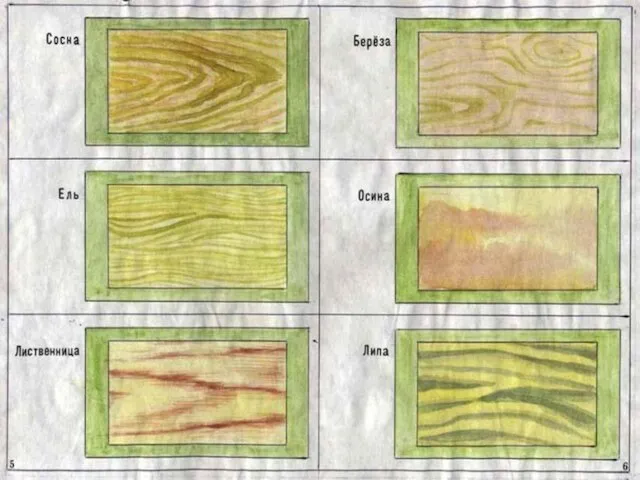 Разрез деревьев
Разрез деревьев Spring Book
Spring Book Барботирование семян
Барботирование семян Женская половая система
Женская половая система Патология органов слуха, зрения и речи. Дальнозоркость
Патология органов слуха, зрения и речи. Дальнозоркость Изучение искусственного отбора
Изучение искусственного отбора Тарас Григорович Шевченко
Тарас Григорович Шевченко Птица, занесённая в Красную книгу
Птица, занесённая в Красную книгу Жизнь дерева
Жизнь дерева Черты сходства приматов и человека
Черты сходства приматов и человека Внешнее строение насекомого. Развитие майского жука
Внешнее строение насекомого. Развитие майского жука Онтогенез индивидуальное развитие организма
Онтогенез индивидуальное развитие организма пищеварение
пищеварение Порядок Агариковые
Порядок Агариковые