Содержание
- 2. Зачем это всё? Для использования NGS в клинической практике нужны точные и воспроизводимые результаты Новые или
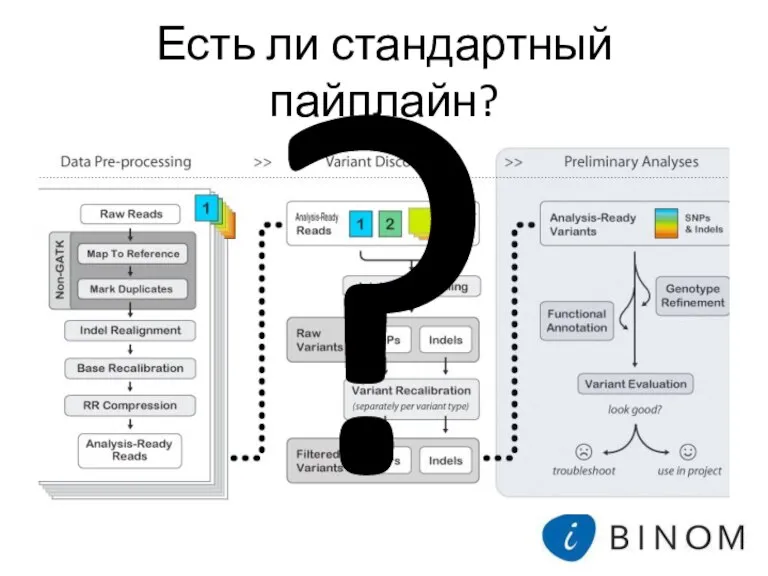
- 3. Есть ли стандартный пайплайн? ?
- 4. Нельзя просто так взять и получить vcf файл!
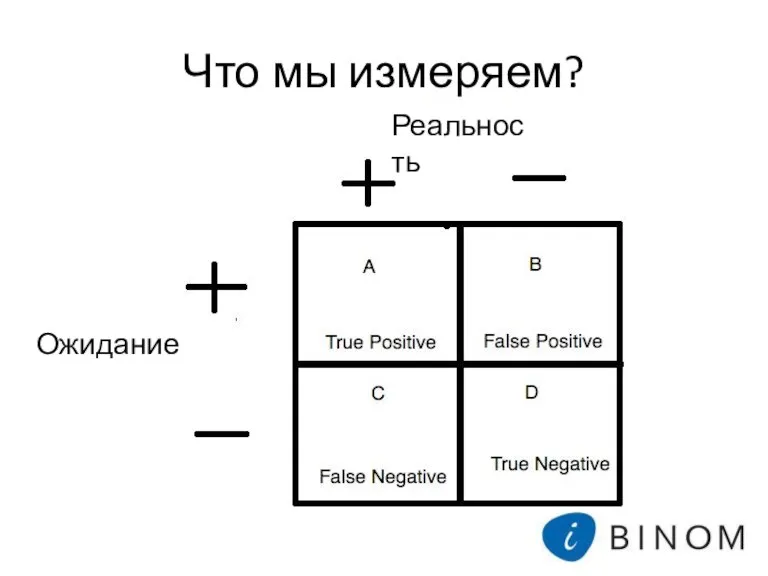
- 5. Что мы измеряем? Ожидание Реальность
- 6. Что мы измеряем?
- 7. Что мы измеряем? Точность (Precision) = TP / (TP + FP) – как много найденных вариантов
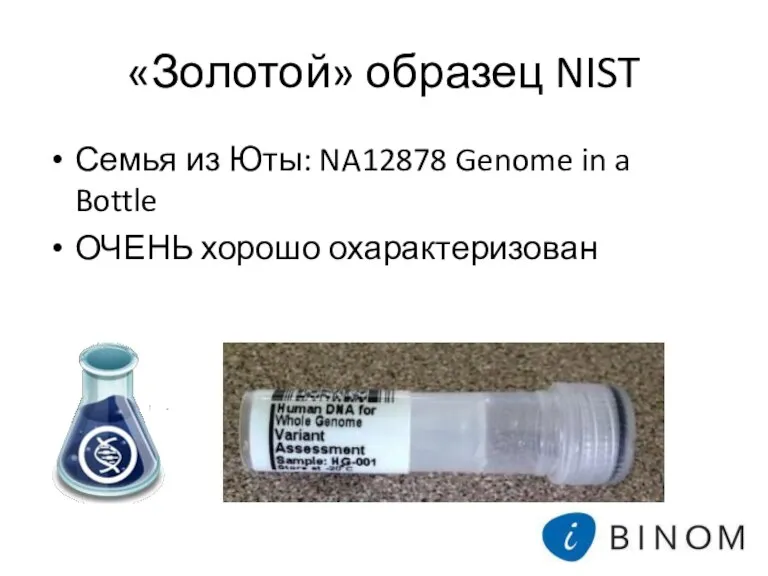
- 8. «Золотой» образец NIST Семья из Юты: NA12878 Genome in a Bottle ОЧЕНЬ хорошо охарактеризован
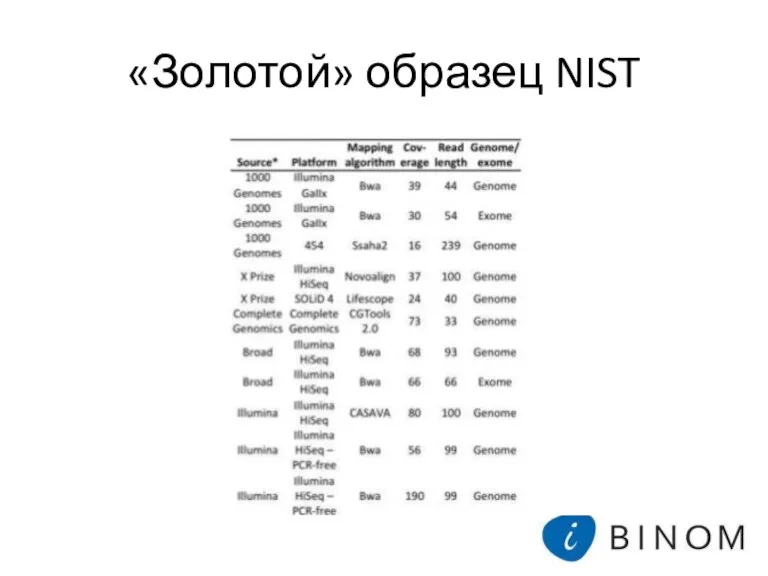
- 9. «Золотой» образец NIST
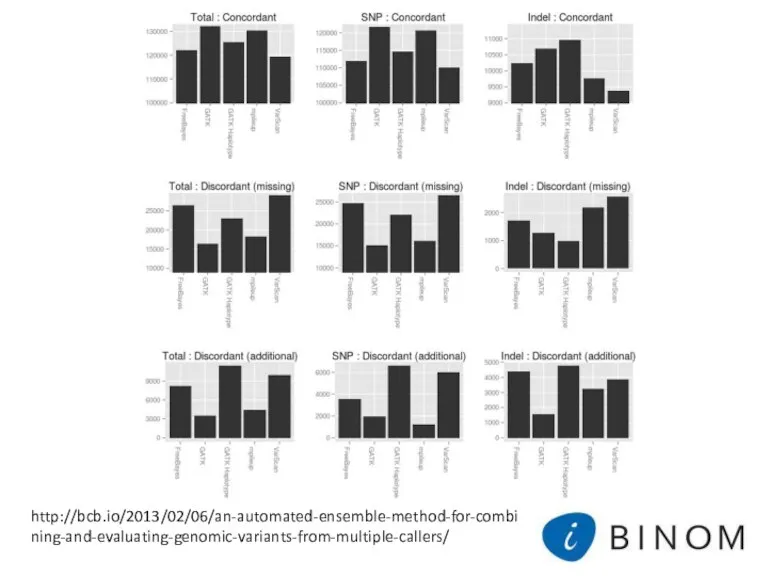
- 10. http://bcb.io/2013/02/06/an-automated-ensemble-method-for-combining-and-evaluating-genomic-variants-from-multiple-callers/
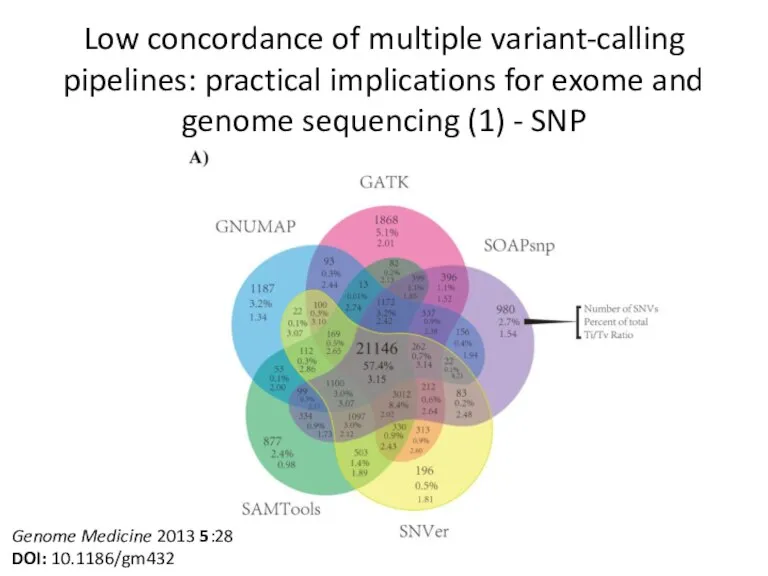
- 11. Low concordance of multiple variant-calling pipelines: practical implications for exome and genome sequencing (1) - SNP
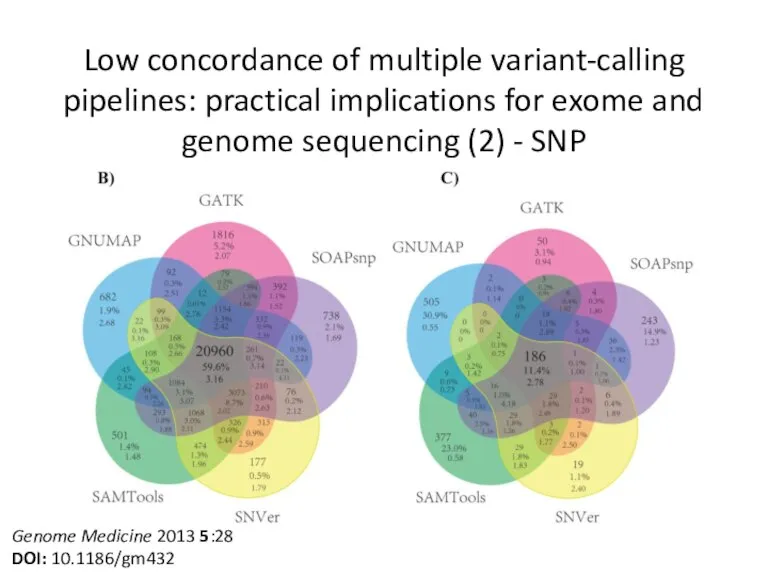
- 12. Low concordance of multiple variant-calling pipelines: practical implications for exome and genome sequencing (2) - SNP
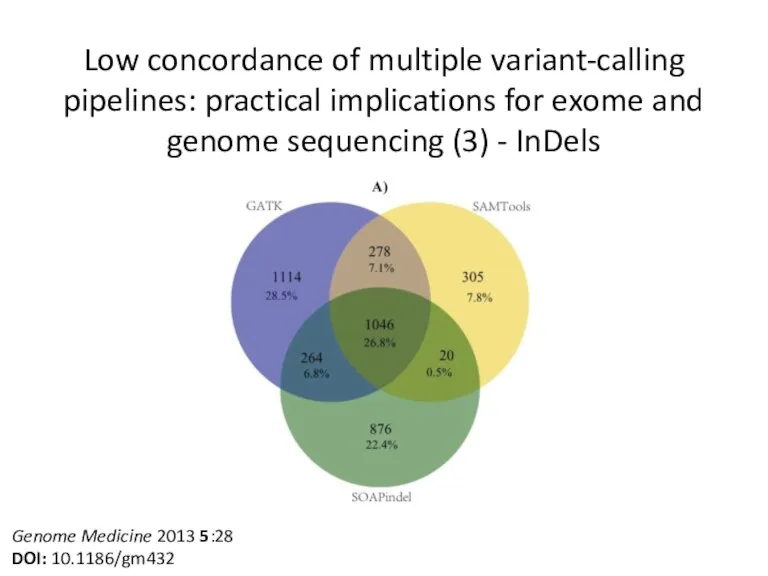
- 13. Low concordance of multiple variant-calling pipelines: practical implications for exome and genome sequencing (3) - InDels
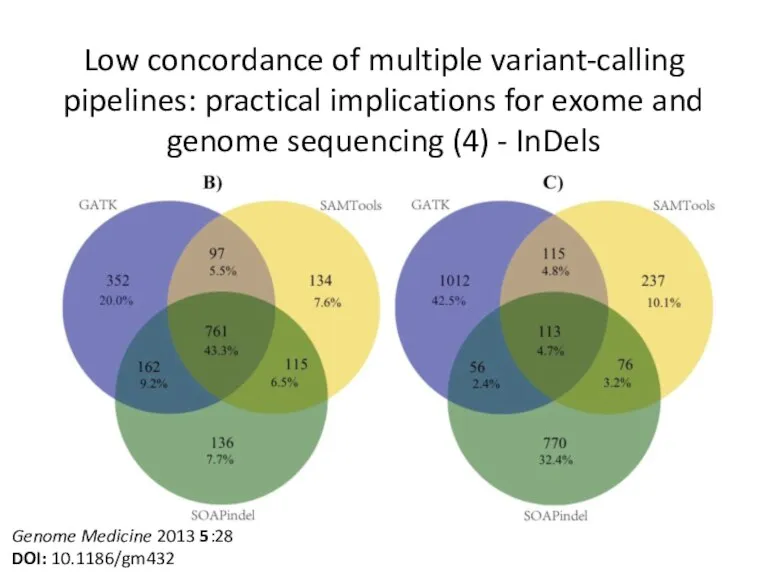
- 14. Low concordance of multiple variant-calling pipelines: practical implications for exome and genome sequencing (4) - InDels
- 15. Что мы решили проверить? Bowtie 2 (version 2.1.0, http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) BWA-MEM (version 0.7.8, http://bio-bwa.sourceforge.net/) Novoalign (version 3,
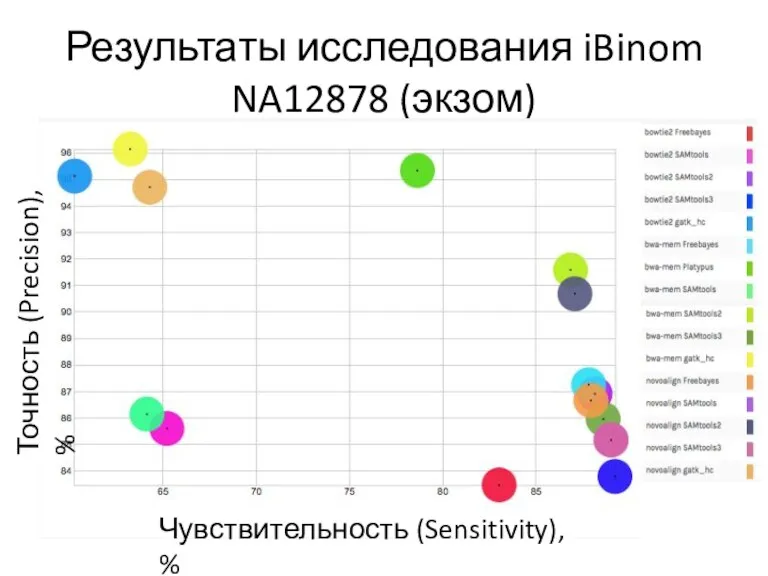
- 16. Результаты исследования iBinom NA12878 (экзом) Чувствительность (Sensitivity), % Точность (Precision), %
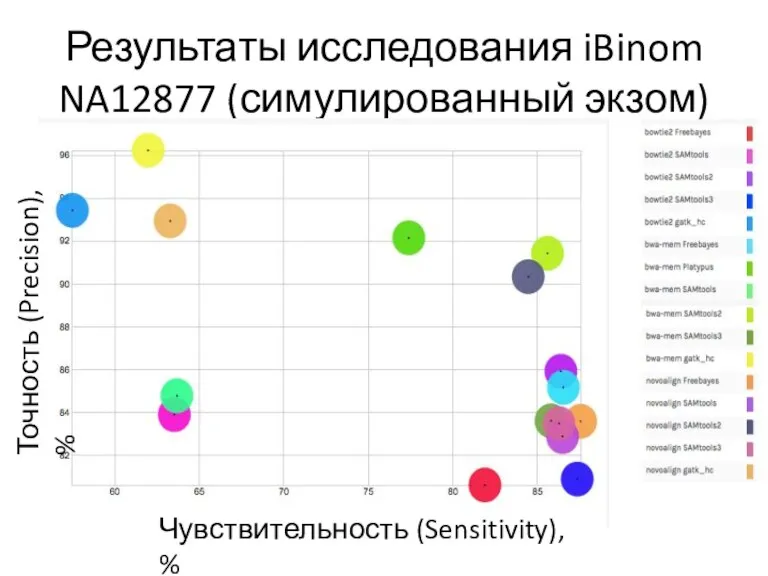
- 17. Результаты исследования iBinom NA12877 (симулированный экзом) Чувствительность (Sensitivity), % Точность (Precision), %
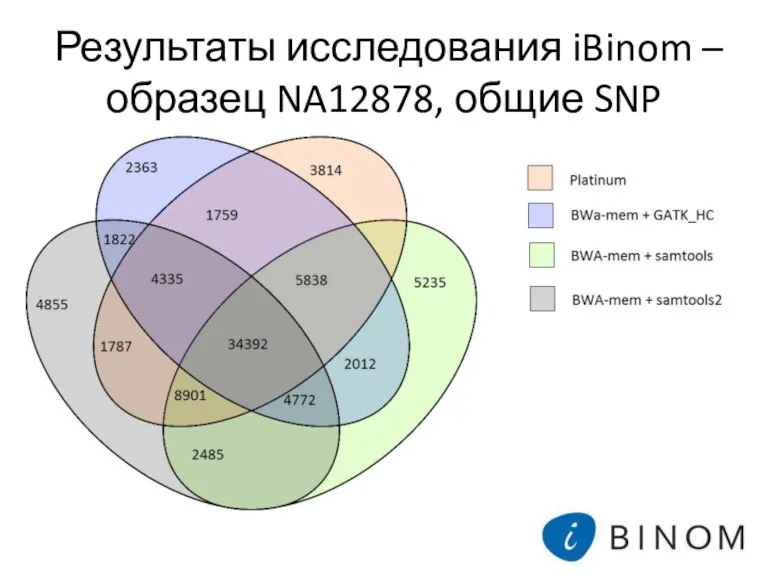
- 18. Результаты исследования iBinom – образец NA12878, общие SNP
- 19. Пара слов о важности настроек bwa_samtools_new2 -- "call -c" (без специальных опций) bwa_samtools_new3 -- "call -p
- 20. Выводы исследования iBinom Не всегда 2 хороших тула хорошо работают вместе (пример: BWA-MEM + GATK HC)
- 21. Почему разные пайплайны дают столь отличающиеся результаты? Потому что входящие в пайплайны блоки варьируются, меняя условия
- 22. Как проверить свои результаты http://www.bioplanet.com/gcat
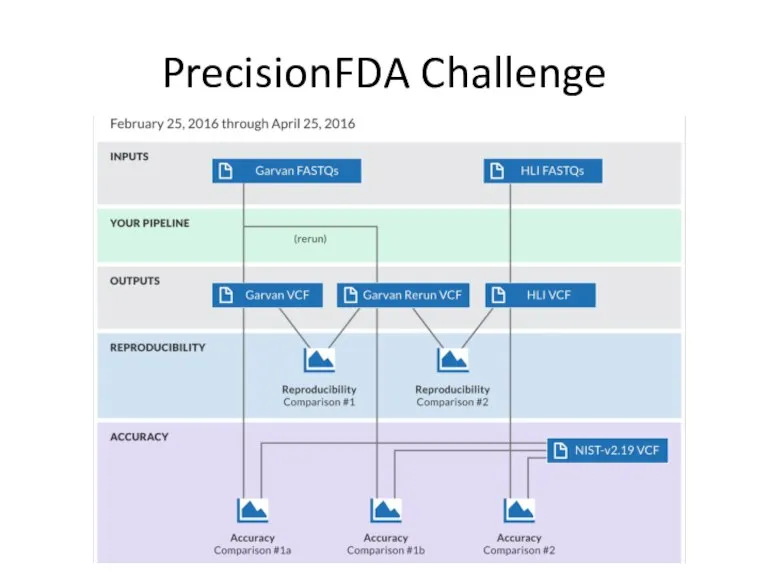
- 23. PrecisionFDA Challenge
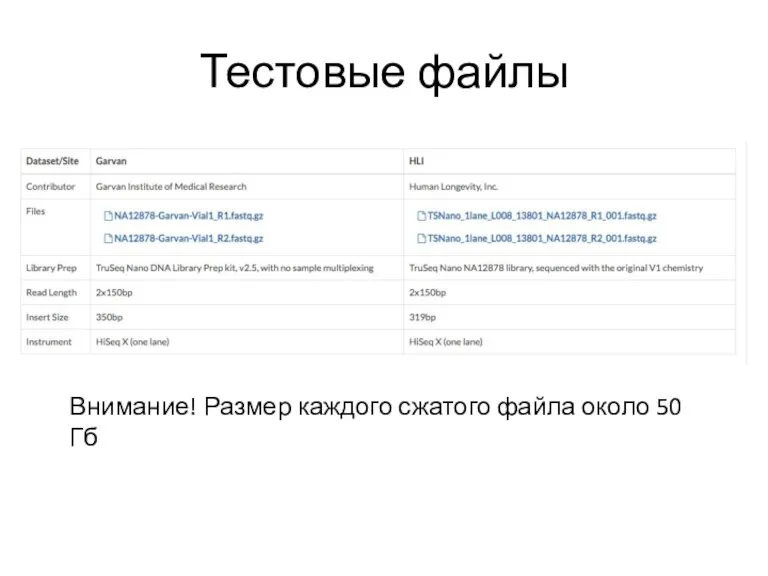
- 24. Тестовые файлы Внимание! Размер каждого сжатого файла около 50 Гб
- 25. Проблемы Невоспроизводимость результатов одного и того же пайплайна! Многие коллеры используют вероятностные модели В силу вероятностной
- 27. Скачать презентацию










 Медиаполигон: Екатеринбург. Написание заметки
Медиаполигон: Екатеринбург. Написание заметки Кодирование информации
Кодирование информации Методы борьбы с дезинформацией
Методы борьбы с дезинформацией Компьютерное зрение
Компьютерное зрение Планерка. Семейство IG
Планерка. Семейство IG Знакомим родителей с программой ПиктоМир
Знакомим родителей с программой ПиктоМир Создание альтернативного сайта КЖРЭУП Железнодорожное
Создание альтернативного сайта КЖРЭУП Железнодорожное Диаграмма компонентов
Диаграмма компонентов Работа с lego mindstorms ev3. Задание № 4: создание собственных изображений и звуков
Работа с lego mindstorms ev3. Задание № 4: создание собственных изображений и звуков Научный поиск и принципы формирования научных работ
Научный поиск и принципы формирования научных работ Тип данных. Структура и коллекции. Visual Studio c#
Тип данных. Структура и коллекции. Visual Studio c# Презентация на тему омпьютерные вирусы и антивирусные программы
Презентация на тему омпьютерные вирусы и антивирусные программы  Циклы в С/С++
Циклы в С/С++ Введение в алгебру логики / логические операции. Таблицы истинности
Введение в алгебру логики / логические операции. Таблицы истинности Как защититься от кибермошенничества
Как защититься от кибермошенничества Просмотр каталогов и файлов без привилегий администратора
Просмотр каталогов и файлов без привилегий администратора Создание мультимедийного электронного ресурса Знаменитые люди Белгородского района
Создание мультимедийного электронного ресурса Знаменитые люди Белгородского района Презентация к лекции_4_10.05.05
Презентация к лекции_4_10.05.05 Программирование на Python. Изучение возможностей и синтаксиса Python: Классы и ООП. Часть 2. 15 занятие
Программирование на Python. Изучение возможностей и синтаксиса Python: Классы и ООП. Часть 2. 15 занятие English Time. Час Англійської. Запускаємо Appinvenor. Lesson 25
English Time. Час Англійської. Запускаємо Appinvenor. Lesson 25 ООП на Delphi
ООП на Delphi Создание программы NC Browser для начинающих пользователей
Создание программы NC Browser для начинающих пользователей Суперкомпьютер IBM Roadrunner
Суперкомпьютер IBM Roadrunner Правовое просвещение пожилых людей. Лекция-семинар: Осторожно - мошенники!
Правовое просвещение пожилых людей. Лекция-семинар: Осторожно - мошенники! Информационная безопасность: mr. Robot в реалиях российского университета
Информационная безопасность: mr. Robot в реалиях российского университета Обучение госслужащих в связи с переводом компьютерного оснащения учреждения на отечественную операционную систему
Обучение госслужащих в связи с переводом компьютерного оснащения учреждения на отечественную операционную систему Передача информации. Локальные компьютерные сети
Передача информации. Локальные компьютерные сети Научно-производственная фирма. Распределенная обработка информации
Научно-производственная фирма. Распределенная обработка информации