Содержание
- 2. Будущее медицины Митио Каку (род. 1947)
- 3. Понятие биоинформатики. Задачи и методы биоинформатики. Молекула ДНК. Основы анализа нуклеиновых последовательностей. Выравнивание нуклеиновых последовательностей. Анализ
- 4. Молекулярная эволюция – это наука, изучающая изменения генетических макромолекул (ДНК, РНК, белков) в процессе эволюции, закономерности
- 5. Биоинформатика — совокупность методов и подходов, включающих в себя: математические методы компьютерного анализа в сравнительной геномике
- 6. 1868 г. - Фридрих Мишер открыл ДНК. 1953 г. – структура ДНК расшифрована Francis Crick, James
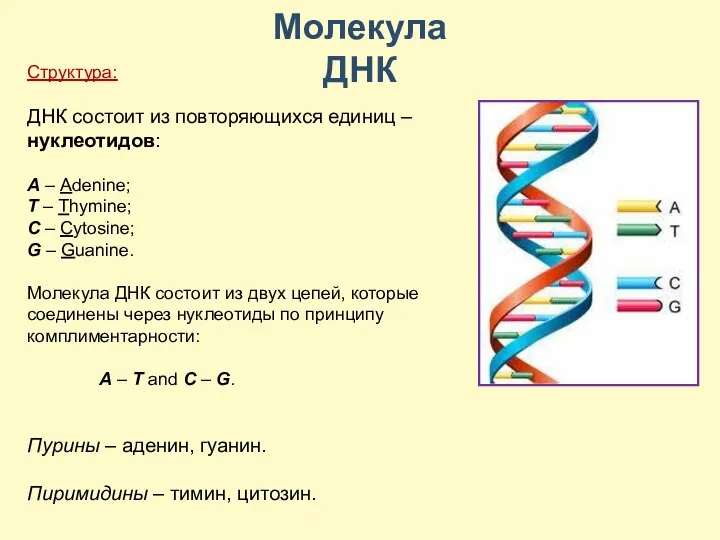
- 7. Структура: ДНК состоит из повторяющихся единиц – нуклеотидов: A – Adenine; T – Thymine; C –
- 8. При воспроизведении генетической информации возникают ошибки.
- 9. Преимущество белого медведя перед бурым при охоте «в снегу».
- 10. Один медведь не эволюционирует, эволюционирует ПОПУЛЯЦИЯ!!!
- 11. Кто родственник человека??? (участок ДНК, отвечающий за усвоение сахаров)
- 12. Все живые объекты – результат молекулярной эволюции
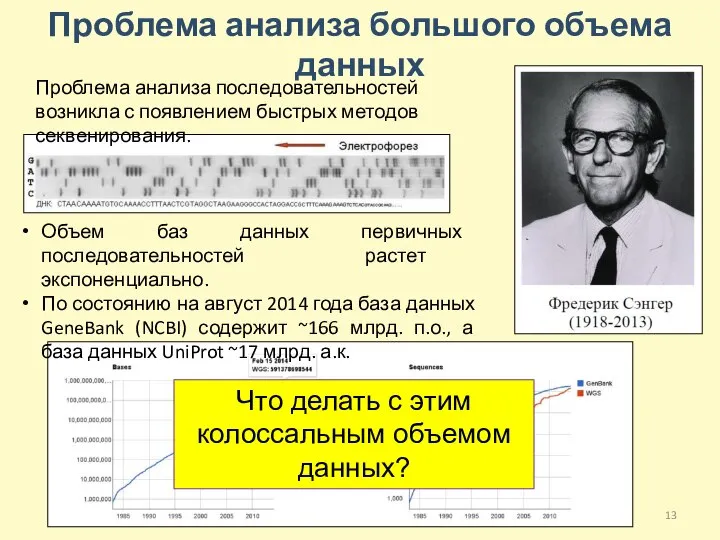
- 13. Проблема анализа большого объема данных Проблема анализа последовательностей возникла с появлением быстрых методов секвенирования. Объем баз
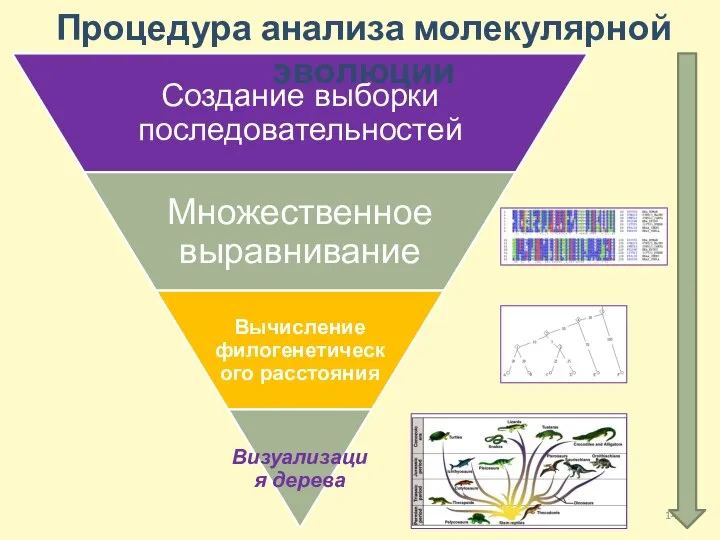
- 14. Процедура анализа молекулярной эволюции
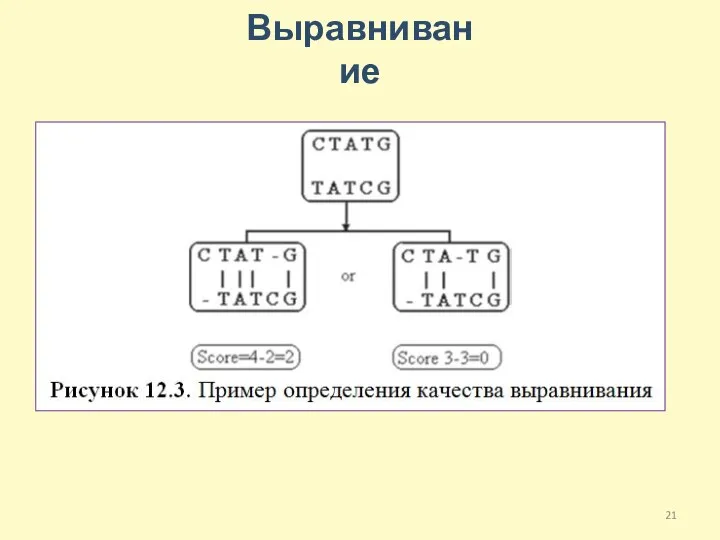
- 15. Выравнивание - процедура попарного сопоставления остатков в последовательностях, направленное на поиск соответствия между ними. Выравнивание позволяет
- 16. Задание последовательности. Статистический анализ. Выравнивание последовательностей. Анализ последовательностей с использованием компьютера
- 17. Задание последовательностей randseq Seq=randseq(SeqLength) Seq=randseq(SeqLength, ...'Alphabet', AlphabetValue,...) randseq(20) ans = TAGCTGGCCAAGCGAGCTTG
- 18. Задание последовательностей aa2nt Преобразование аминокислотной последовательности в нуклеотидную. nucleo_seq= aa2nt(amino_seq, 'GeneticCode', code_number, …'alphabet', 'alphabet_name'), Пример: >>
- 19. Статистический анализ aacount amino_s=aacount(amino_seq, 'chart', chart_value), >> seq=randseq(25,'alphabet','amino') >> amino_acid=aacount(seq) seq = RCYDTLVRHNVASTWRGQTHYDQNN amino_acid = A:
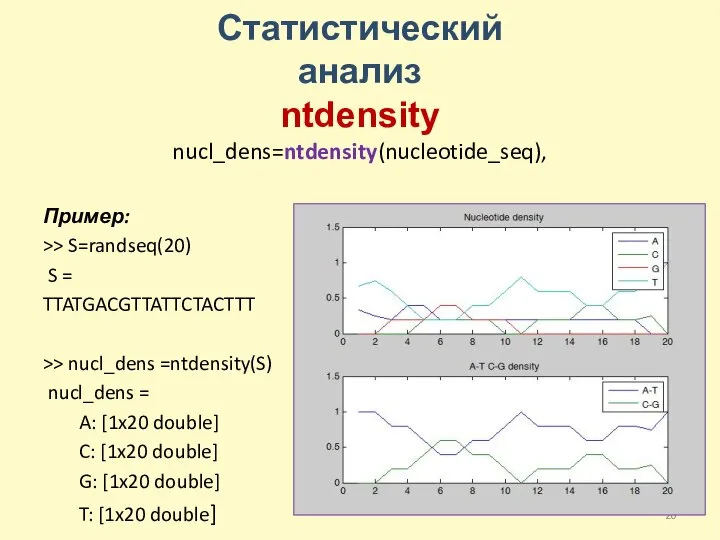
- 20. Статистический анализ ntdensity nucl_dens=ntdensity(nucleotide_seq), Пример: >> S=randseq(20) S = TTATGACGTTATTCTACTTT >> nucl_dens =ntdensity(S) nucl_dens = A:
- 21. Выравнивание
- 22. Способы выравнивания Глобальное выравнивание (последовательности одинаковой длины) Локальное выравнивание
- 24. Выравнивание Глобальное выравнивание Алгоритм Нидлмана-Вунша [score, alignment]=nwalign(seq_1, seq_2, 'showscore', true) s_1='lifescience'; s_2='sefesici'; [score, alignment]=nwalign(s_1, s_2, 'showscore',true)
- 25. Выравнивание Локальное выравнивание Алгоритм Смита-Ватермана [score, alignment]=swalign(seq_1, seq_2, 'showscore', true). s_1='lifescience'; s_2='sefesici'; [score, alignment]=swalign (s_1, s_2,
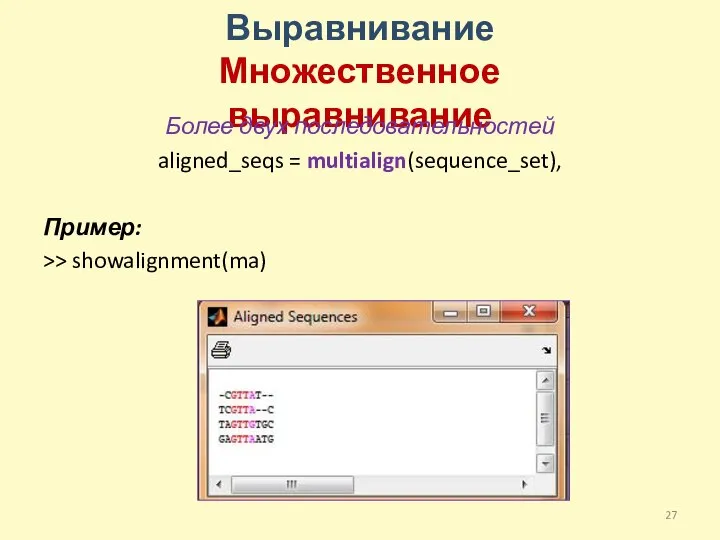
- 26. Выравнивание Множественное выравнивание Более двух последовательностей aligned_seqs = multialign(sequence_set), Пример: >> seqs = strvcat ('CGTTAT', 'TCGTTAC',
- 27. Выравнивание Множественное выравнивание Более двух последовательностей aligned_seqs = multialign(sequence_set), Пример: >> showalignment(ma)
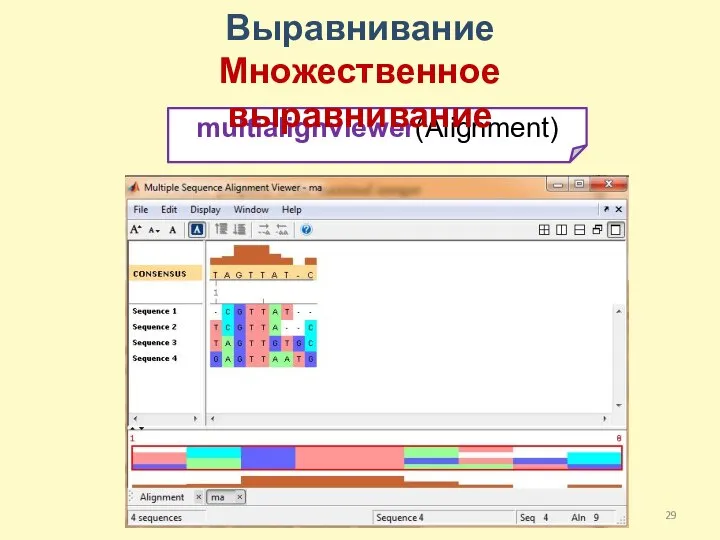
- 28. Выравнивание Множественное выравнивание Для длинных последовательностей (длиннее 64 символов) нужно использоваться другую форму команды showalignment: showalignment(Alignment,
- 29. multialignviewer(Alignment) Выравнивание Множественное выравнивание
- 30. Литература Обязательная Бутиловский, А.В. Основные методы молекулярной эволюции: Монография / А.В. Бутиловский, Е.В. Барковский, В.Э. Бутиловский,
- 31. Глаз – продукт слепой эволюции???
- 32. «Слепой мир» МУТАЦИЯ
- 33. Так видит светочувствительная бактерия
- 34. Бактерии научились избегать солнечного света
- 35. Появление светочувствительного пятна «Отыскивали» солнечный свет
- 36. Плоские черви (чешуйчатое углубление) Видны очертания
- 37. Появилось углубление
- 38. Сужение отверстие до размеров точки Покрылось пленкой
- 39. Сужение отверстие до размеров точки Узкое отверстие пропускает меньше света, но изображение более четкое
- 41. Скачать презентацию

















![Выравнивание Глобальное выравнивание Алгоритм Нидлмана-Вунша [score, alignment]=nwalign(seq_1, seq_2, 'showscore', true) s_1='lifescience'; s_2='sefesici';](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1026263/slide-23.jpg)
![Выравнивание Локальное выравнивание Алгоритм Смита-Ватермана [score, alignment]=swalign(seq_1, seq_2, 'showscore', true). s_1='lifescience'; s_2='sefesici';](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1026263/slide-24.jpg)











 Школьное профессиональное объединение учителей математики в условиях перехода на ФГОС ООО Широкова О.Г., заведующая кафедрой ма
Школьное профессиональное объединение учителей математики в условиях перехода на ФГОС ООО Широкова О.Г., заведующая кафедрой ма Презентация на тему Площадь трапеции (8 класс)
Презентация на тему Площадь трапеции (8 класс)  Объемы тел вращения
Объемы тел вращения Решение задач на проценты
Решение задач на проценты Сложение и вычитание дробей с разными знаменателями
Сложение и вычитание дробей с разными знаменателями Деление на равные части
Деление на равные части Решение неравенств методом интервалов
Решение неравенств методом интервалов Метод Гаусса
Метод Гаусса Вычисление неопределенного интеграла
Вычисление неопределенного интеграла Презентация по математике "Старинные меры длинны" -
Презентация по математике "Старинные меры длинны" -  Решение СЛАУ методом Крамера
Решение СЛАУ методом Крамера Линейная зависимость и линейная независимость системы векторов. Семинар 4
Линейная зависимость и линейная независимость системы векторов. Семинар 4 Приемы вычислений для случаев вида 26+7. 2 класс
Приемы вычислений для случаев вида 26+7. 2 класс Теоретический зачет по теме Начальные геометрические сведения
Теоретический зачет по теме Начальные геометрические сведения Цилиндр
Цилиндр Число и цифра 7
Число и цифра 7 Сколько? Как? Почему? Математическая игра
Сколько? Как? Почему? Математическая игра Основы эконометрического моделирования
Основы эконометрического моделирования Тела вращения. Цилиндр и его элементы
Тела вращения. Цилиндр и его элементы Равенство, неравенство. Устный счет (1 класс)
Равенство, неравенство. Устный счет (1 класс) Первые цифры
Первые цифры Презентация на тему Решение задач на построение сечений многогранников
Презентация на тему Решение задач на построение сечений многогранников  Великолепный часослов герцога Беррийского. Математика в каждом
Великолепный часослов герцога Беррийского. Математика в каждом Решите неравенство
Решите неравенство Численные методы. ВСР 3
Численные методы. ВСР 3 Полет на планету чисел. Открытый урок
Полет на планету чисел. Открытый урок Иррациональные уравнения
Иррациональные уравнения Порядок выполнения действий в числовых выражениях
Порядок выполнения действий в числовых выражениях