Содержание
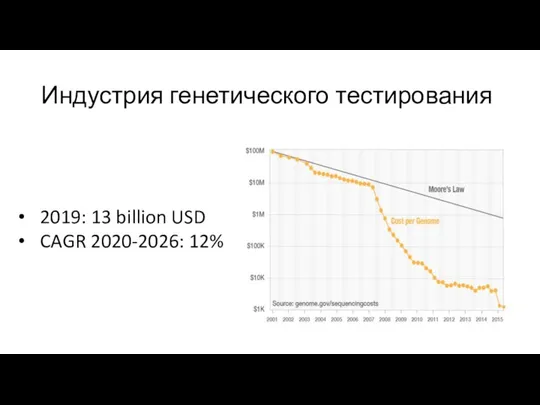
- 2. Индустрия генетического тестирования 2019: 13 billion USD CAGR 2020-2026: 12%
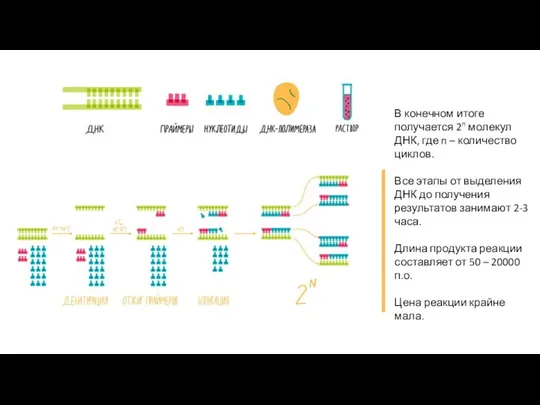
- 5. В конечном итоге получается 2n молекул ДНК, где n – количество циклов. Все этапы от выделения
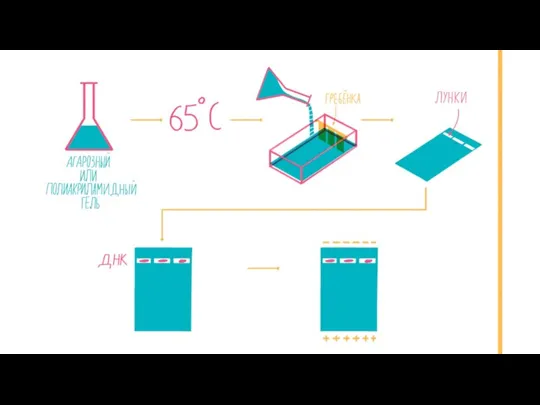
- 7. Полимеразная цепная реакция Маркер длин ДНК ПК Образец – Норма ОК Образец – Делеция
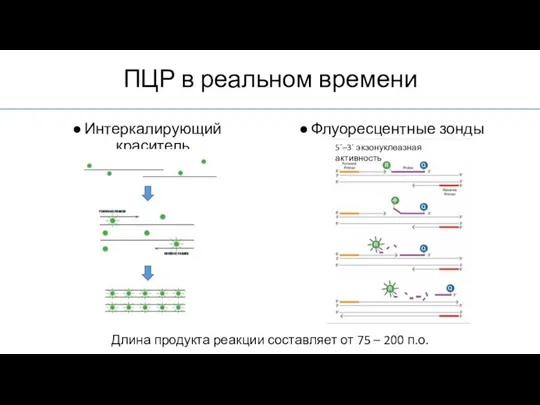
- 8. ПЦР в реальном времени Интеркалирующий краситель Флуоресцентные зонды Длина продукта реакции составляет от 75 – 200
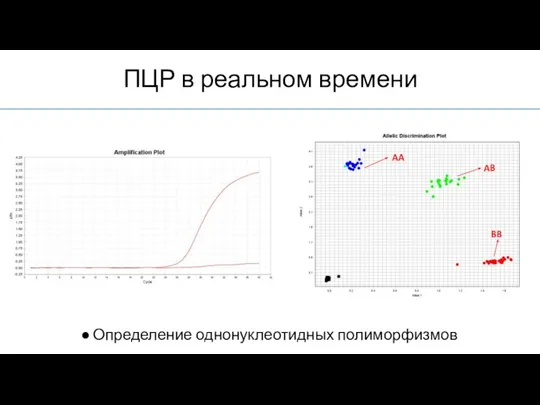
- 9. ПЦР в реальном времени Определение однонуклеотидных полиморфизмов AA AB BB
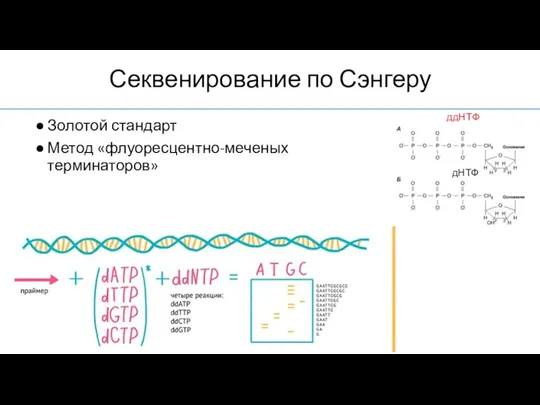
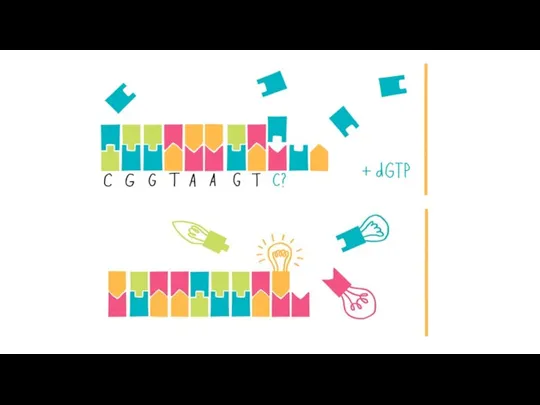
- 10. Секвенирование по Сэнгеру Золотой стандарт Метод «флуоресцентно-меченых терминаторов» ддНТФ дНТФ
- 11. 3130/3130xL 4/16 капилляров SeqStudio 4 капилляра 3500/3500xL 8/16 капилляров Секвенирование по Сэнгеру
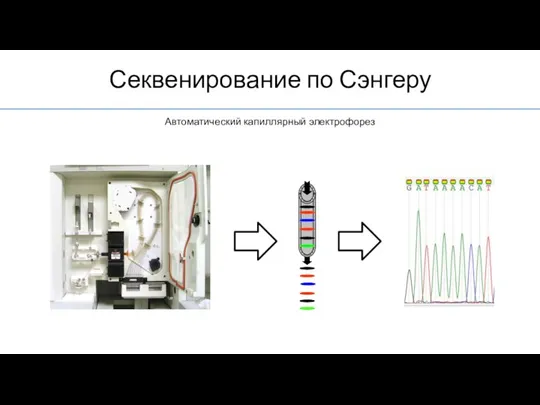
- 12. Секвенирование по Сэнгеру Автоматический капиллярный электрофорез
- 13. Секвенирование по Сэнгеру Вариант chr7:103159789 C>T, RELN выявлена в гомозиготной форме
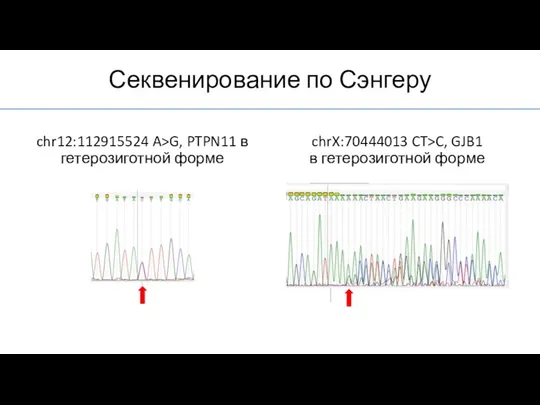
- 14. Секвенирование по Сэнгеру chrX:70444013 CT>C, GJB1 в гетерозиготной форме chr12:112915524 A>G, PTPN11 в гетерозиготной форме
- 15. Секвенирование по Сэнгеру Этапы подготовки образцов: Выделение ДНК – 1 час Подбор праймеров – 1 час
- 16. Секвенирование по Сэнгеру Проблемы: Сложность при прохождение гомополимерных последовательностей
- 17. Секвенирование по Сэнгеру Аллель специфичные праймеры - при попадании однонуклеотидного полиморфизма в 3’ область праймера
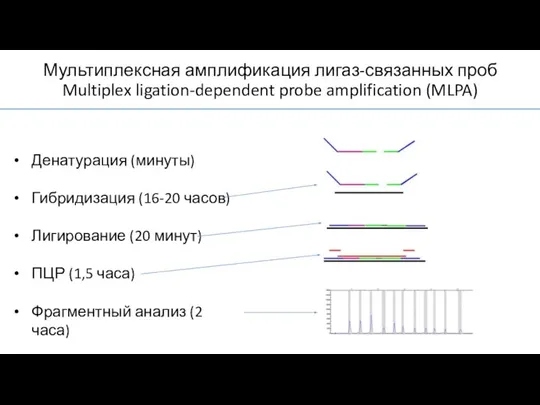
- 18. Мультиплексная амплификация лигаз-связанных проб Multiplex ligation-dependent probe amplification (MLPA) Денатурация (минуты) Гибридизация (16-20 часов) Лигирование (20
- 19. Мультиплексная амплификация лигаз-связанных проб Multiplex ligation-dependent probe amplification (MLPA)
- 20. Плюсы: Позволяет одновременно детектировать большое количество мутаций или наличие/отсутствие определенных участков ДНК (до 50) (SNP, делеции,
- 21. Методы анализа длины тринуклеотидных повторов Основным методом является ПЦР-анализ полиморфных повторов с последующей визуализацией продуктов амплификации.
- 22. Методы анализа длины тринуклеотидных повторов Фрагментный анализ образцов пациента с Синдромом Мартина — Белла
- 23. Полногеномные методы исследования
- 24. Выделение геномной ДНК Фрагментация ДНК Лигирование адаптеров ПЦР Полногеномное секвенирование Обогащение Таргетное секвенирование Фрагментация Окрашивание -
- 25. Лизис клеток Разделение фаз ц/ф Экстракция Очень важный этап! От 30 минут до 2 суток Очень
- 26. Фрагментация ДНК Лигирование адаптеров ПЦР Очень важный этап! Необходимо получить смесь фрагментов ДНК в определенном диапазоне
- 27. Высокопроизводительное секвенирование Секвенируем то, что хотим проанализировать. Обогащаем смесь!
- 28. Высокопроизводительное секвенирование
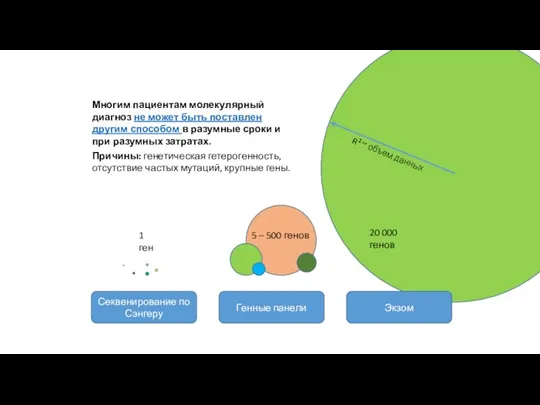
- 29. 20 000 генов Экзом Генные панели Секвенирование по Сэнгеру 5 – 500 генов 1 ген Многим
- 30. Высокопроизводительное секвенирование
- 32. Illumina: Точность сопоставима с секвенированием по Сэнгеру, 0.1% Очень высокая производительность Низкая стоимость на нуклеотид Парные
- 33. Повторы Покрытие Сбалансированные транслокации, инверсии То, что не покрыто панелью CNV Высокопроизводительное секвенирование
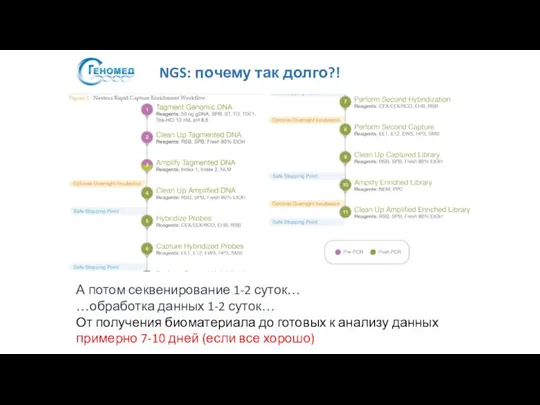
- 34. NGS: почему так долго?! А потом секвенирование 1-2 суток… …обработка данных 1-2 суток… От получения биоматериала
- 35. Секвенировать 1 образец – глупо Собирается «пул» образцов, каждый из которых баркодируется. Все это смешивается в
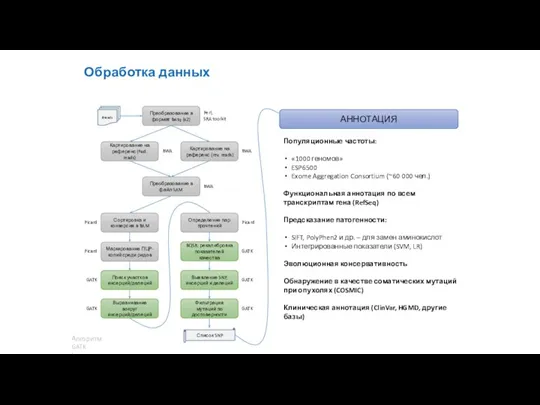
- 36. Обработка данных Преобразование в формат fastq (x2) Картирование на референс (fwd. reads) Картирование на референс (rev.
- 37. Система Genexus – новое решение от Thermo Fisher Scientific для клиник
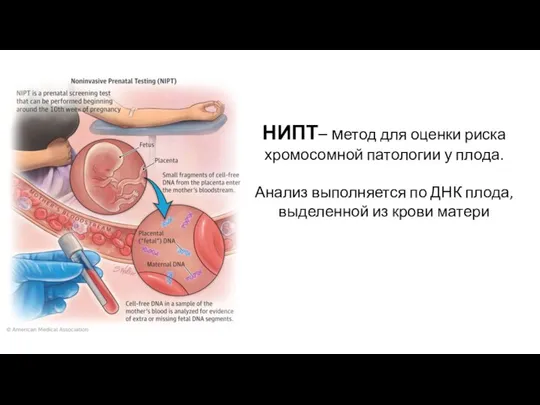
- 38. НИПТ– метод для оценки риска хромосомной патологии у плода. Анализ выполняется по ДНК плода, выделенной из
- 39. Методика анализа по cffDNA НИПС Panorama
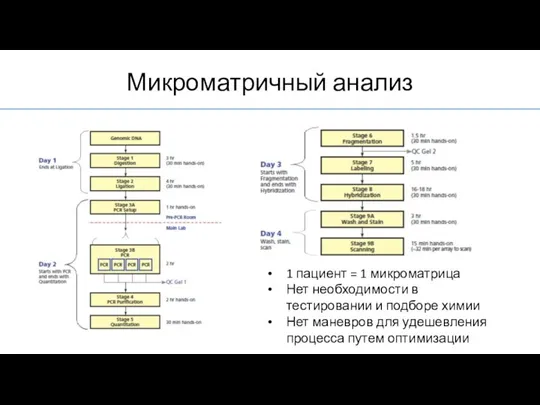
- 41. Микроматричный анализ
- 42. 1 пациент = 1 микроматрица Нет необходимости в тестировании и подборе химии Нет маневров для удешевления
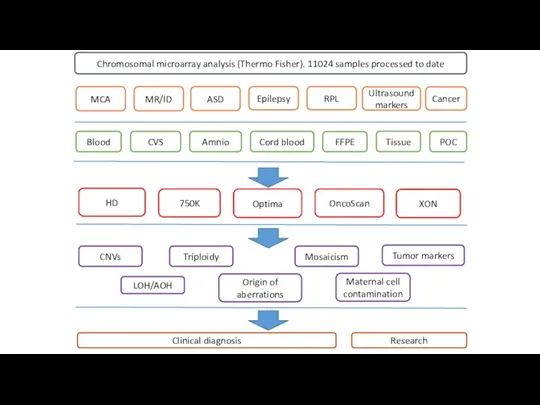
- 43. Clinical diagnosis Research Blood CVS FFPE Amnio Cord blood Mosaicism LOH/AOH CNVs HD Tissue 750K Optima
- 44. Coverage of clinically important regions in different microarrays HD 750K Optima
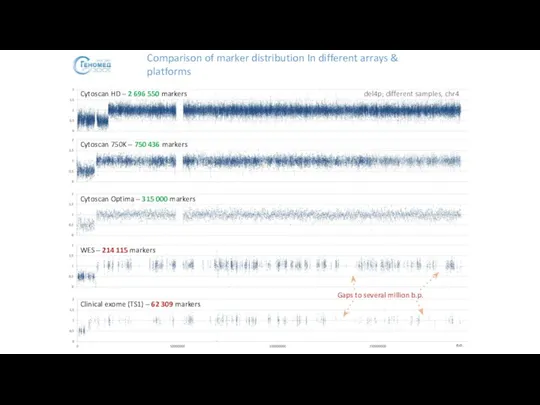
- 45. Comparison of marker distribution In different arrays & platforms Clinical exome (TS1) – 62 309 markers
- 46. Сбалансированные хромосомные перестройки (транслокации, инверсии) Точковые мутации Болезни экспансии тринуклеотидных повторов Микроделеции/микродупликации, размер которых меньше разрешающей
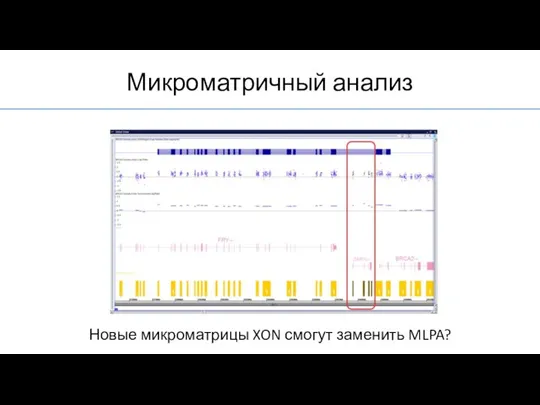
- 47. Микроматричный анализ Новые микроматрицы XON смогут заменить MLPA?
- 48. Будущее молекулярного кариотипирования
- 49. Технология молекулярной инверсионной пробы (MIP)
- 50. Онкоскан (хромосомный микроматричный анализ) 220 000 snp маркеров Полногеномный анализ числа копий генов и хромосомных сегментов,
- 51. Научная работа Научный сервис для различных государственных и коммерческих учреждений Разработка проекта эксперимента, его выполнение, анализ
- 52. СЕСАНА – официальный поставщик оборудования и реагентов для исследований в области молекулярной биологии и генетики [email protected]
- 53. Геномед – первый OEM-партнер Thermo Fisher Scientific в России
- 55. Скачать презентацию

































 Эндокринная регуляция функций организма человека
Эндокринная регуляция функций организма человека Водоросли. Среда обитания
Водоросли. Среда обитания Собака – друг человека. 1 класс
Собака – друг человека. 1 класс Подцарство Одноклеточные
Подцарство Одноклеточные Органы чувств
Органы чувств Бактерии
Бактерии Bottleneck Effect In Human Population
Bottleneck Effect In Human Population будова клітіини (1)
будова клітіини (1) Различие в содержании вдыхаемого и выдыхаемого воздуха. Кислород. Окисление сложных органических веществ
Различие в содержании вдыхаемого и выдыхаемого воздуха. Кислород. Окисление сложных органических веществ Презентация на тему Живі організми як особливе середовище існування
Презентация на тему Живі організми як особливе середовище існування  Жизненный цикл клетки. Митотический цикл. Регуляция митотического цикла. Понятие об апоптозе
Жизненный цикл клетки. Митотический цикл. Регуляция митотического цикла. Понятие об апоптозе Рыбы. Общая характеристика
Рыбы. Общая характеристика 0b77-00096dce-87c54f21 (1)
0b77-00096dce-87c54f21 (1) Сибирская язва
Сибирская язва Ткани
Ткани Взаимоотношения в природных сообществах
Взаимоотношения в природных сообществах Окрашивание препаратов для микроскопии
Окрашивание препаратов для микроскопии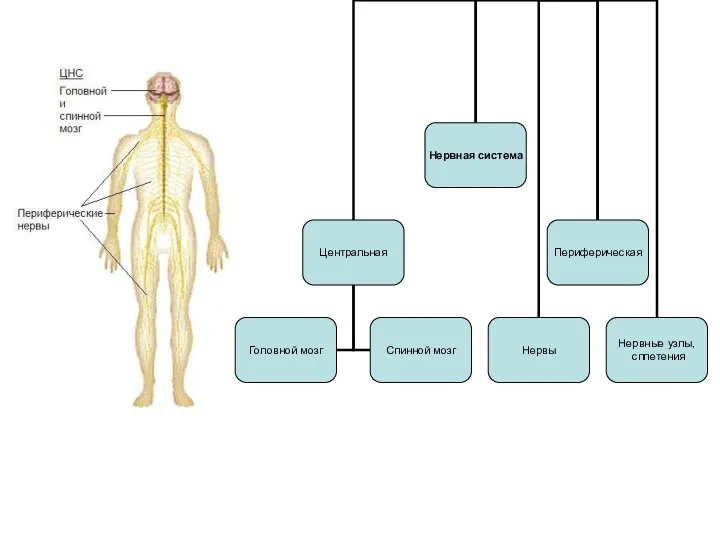 Нервная система. Микроструктура
Нервная система. Микроструктура Биология. Наука о жизни
Биология. Наука о жизни Африканский страус
Африканский страус Оплодотворение. Двойное оплодотворение
Оплодотворение. Двойное оплодотворение Ягодная викторина
Ягодная викторина Лягушки
Лягушки Лучевая болезнь. Патофизиология воздействия ионизирующего излучения
Лучевая болезнь. Патофизиология воздействия ионизирующего излучения Презентация на тему Отряд Таракановые
Презентация на тему Отряд Таракановые  Классификация цветковых растений Цель: познакомиться с классификацией растений Научиться давать классификацию растения
Классификация цветковых растений Цель: познакомиться с классификацией растений Научиться давать классификацию растения Состав плазмы крови
Состав плазмы крови Меры профилактики кожных заболеваний
Меры профилактики кожных заболеваний