Содержание
- 2. Механизмы репарации ДНК
- 3. Объекты для изучения репарации Escherichia coli Saccharomyces cerevisiae Caenorhabditis elegans Arabidopsis Thaliana Мыши, крысы Клеточные культуры
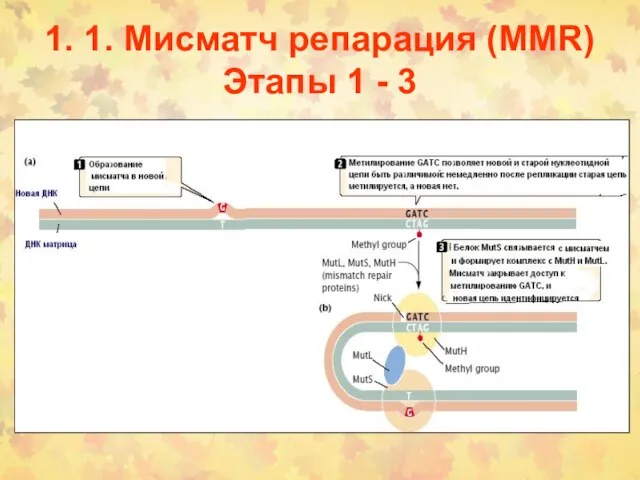
- 4. 1. 1. Мисматч репарация (MMR) Этапы 1 - 3
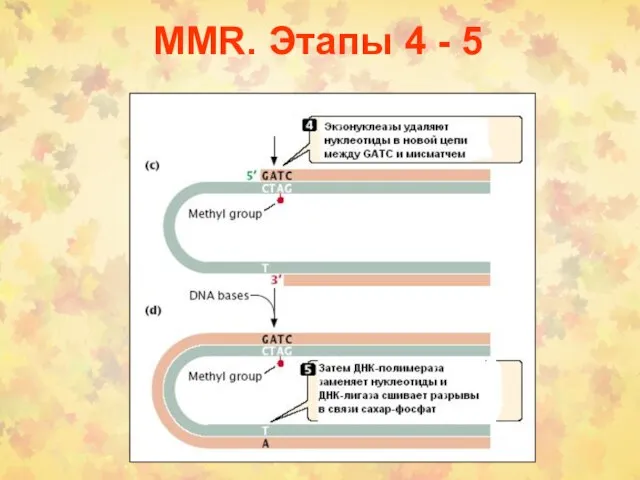
- 5. MMR. Этапы 4 - 5
- 6. А. Репарация мисматчей у бактерий VSP- very short patch repair Short patch repair Long patch repair
- 7. VSP- very short patch repair – 1 В основном удаляется Т из мисматчей G/T и C/T.
- 8. VSP- very short patch repair – 2 MutY заменяет А из мисматчей C/A и G/A. Это
- 9. MMR млекопитающих 9 генов: MLH1, MLH3, PMS1-2, MSH2-6 MSH – гомолог MutS MLH – гомолог MutL
- 10. MMR человека На примере болезни HNPCC (heredity non-polyposis colorectal cancer) в 1993-1994 гг. У человека найдено
- 11. MMR человека На примере болезни HNPCC (heredity non-polyposis colorectal cancer) в 1993-1994 гг. Пациенты с HNPCC
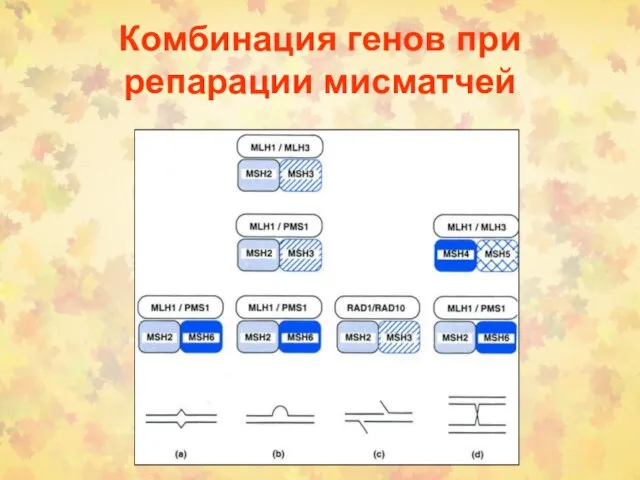
- 12. Комбинация генов при репарации мисматчей
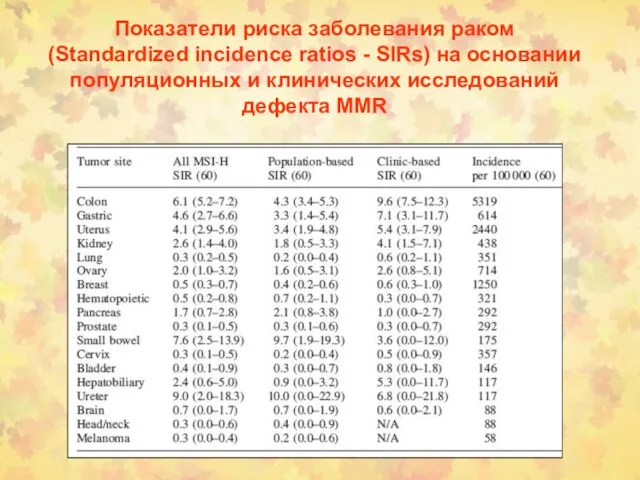
- 13. Показатели риска заболевания раком (Standardized incidence ratios - SIRs) на основании популяционных и клинических исследований дефекта
- 14. Механизмы, осуществляющие вклад в специфичность клеточных типов, чувствительных к дефициту MMR
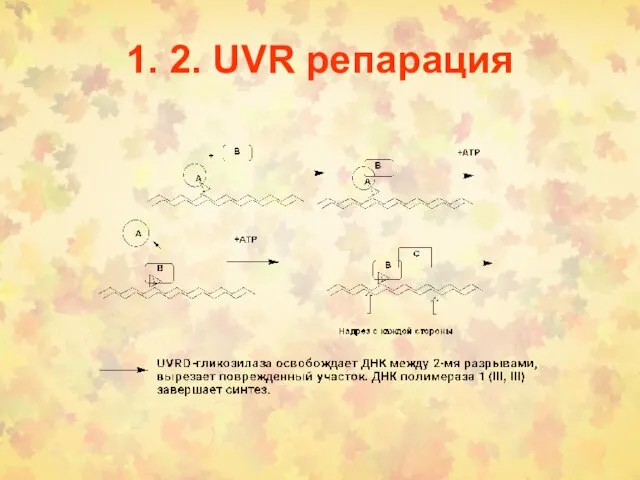
- 15. 1. 2. UVR репарация
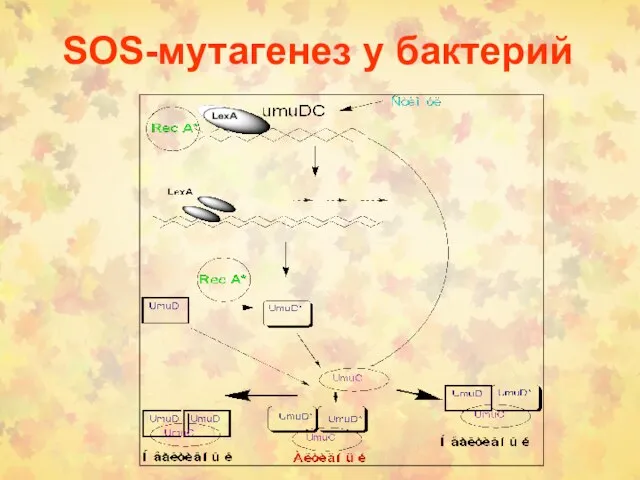
- 16. SOS-мутагенез у бактерий
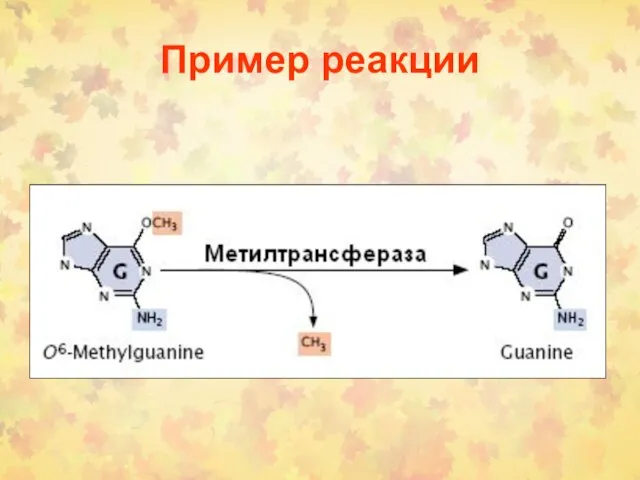
- 17. 2. Прямая репарация Репарируются О6-метилгуанин и О4-метилгуанин ферментом МТаза (MGMT). У Е. coli 2 фермента (гены
- 18. Пример реакции
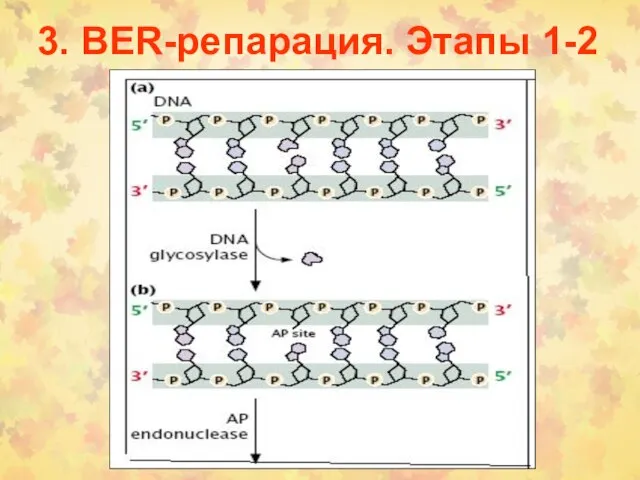
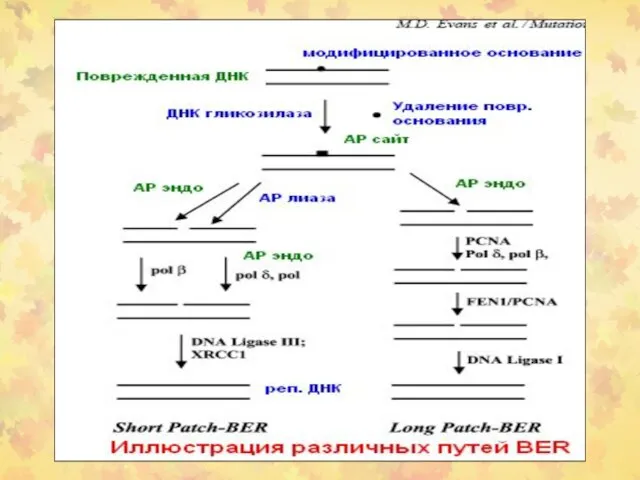
- 19. 3. BER-репарация. Этапы 1-2
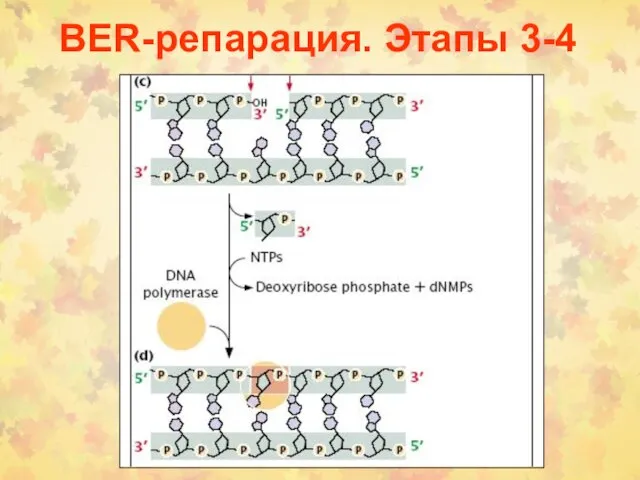
- 20. BER-репарация. Этапы 3-4
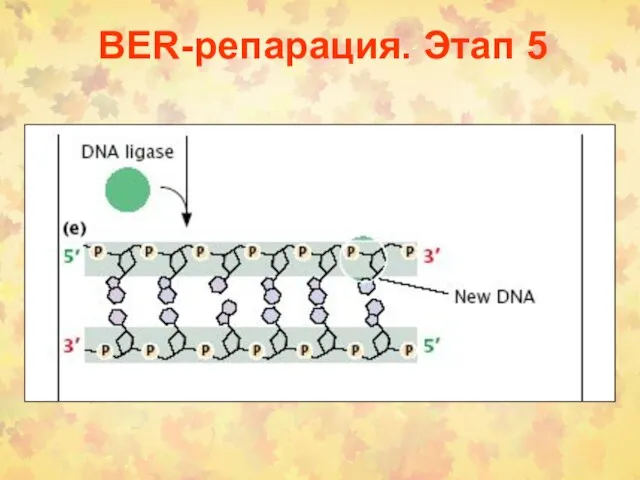
- 21. BER-репарация. Этап 5
- 23. 4. NER-репарация 1. TCR – transcription coupled repair (преимущественная репарация траснкрибируемых цепей гена) 2. GGR –
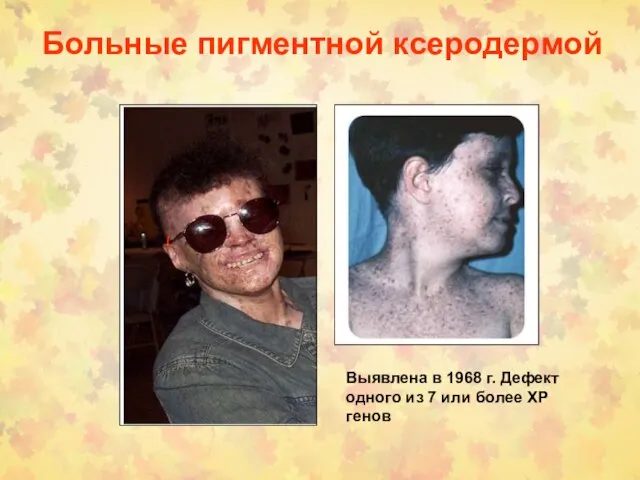
- 24. Больные пигментной ксеродермой ( Выявлена в 1968 г. Дефект одного из 7 или более XP генов
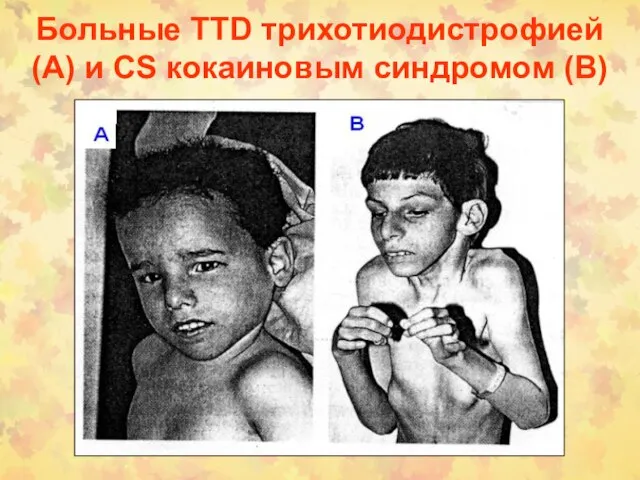
- 25. Больные TTD трихотиодистрофией (А) и CS кокаиновым синдромом (В)
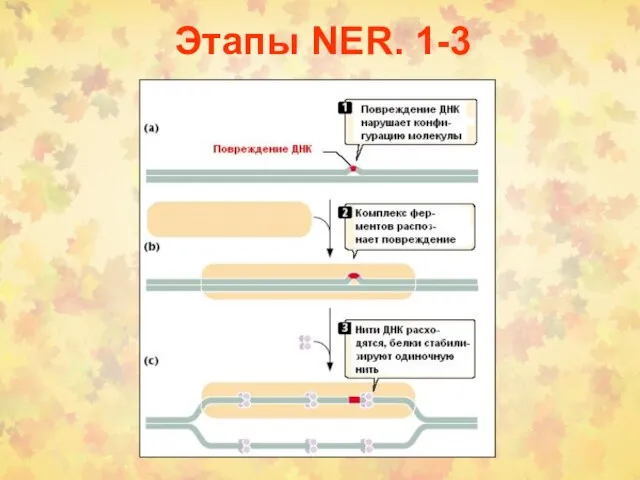
- 26. Этапы NER. 1-3
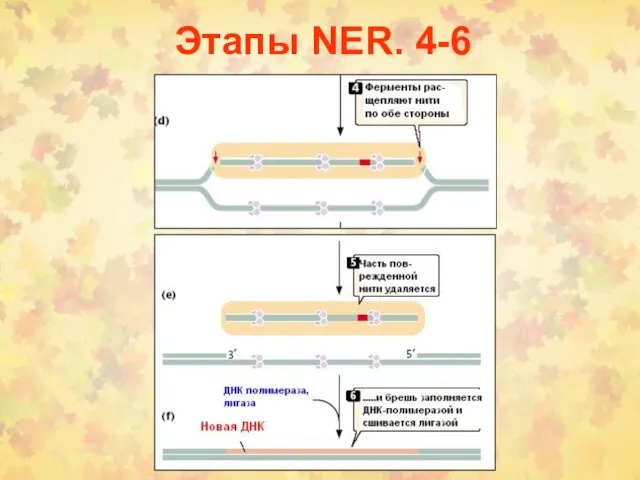
- 27. Этапы NER. 4-6
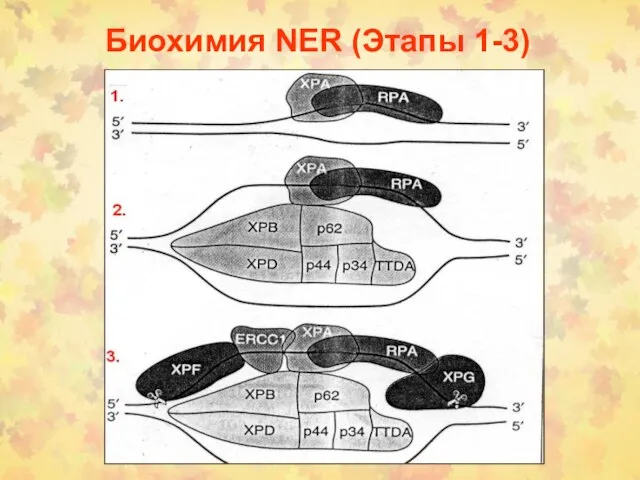
- 28. Биохимия NER (Этапы 1-3)
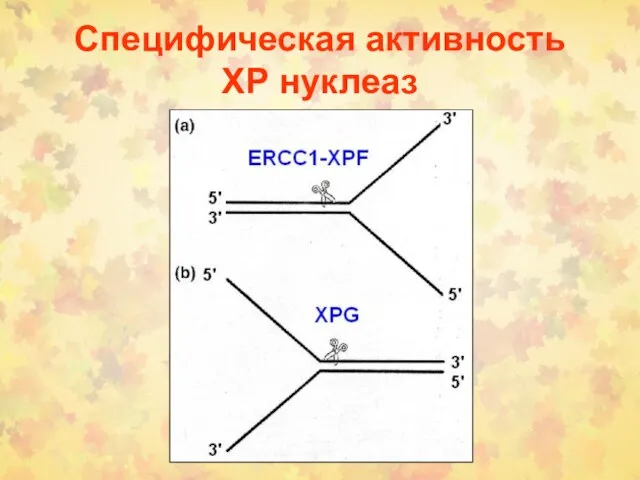
- 29. Специфическая активность ХР нуклеаз
- 30. Биохимия NER (Этапы 4-5)
- 31. Повреждение ХР при болезнях ХР – мутации в генах XP A-D,F,G TTD – серо-дефицитные хрупкие волосы,
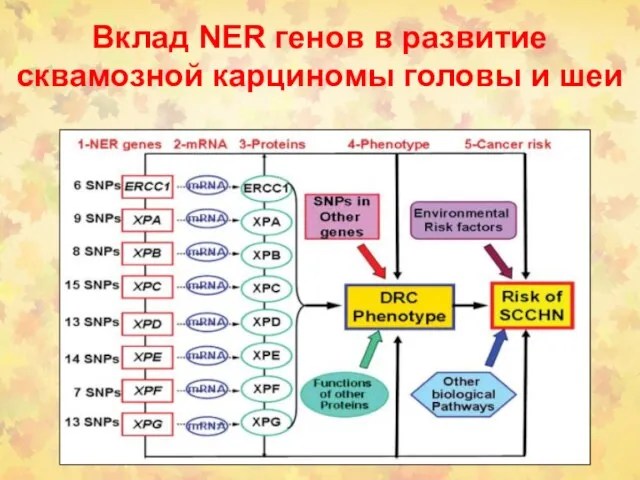
- 32. Вклад NER генов в развитие сквамозной карциномы головы и шеи
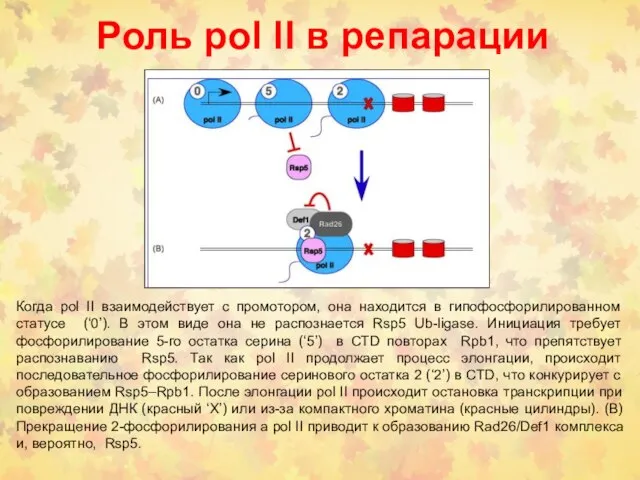
- 33. Роль pol lI в репарации Когда pol II взаимодействует с промотором, она находится в гипофосфорилированном статусе
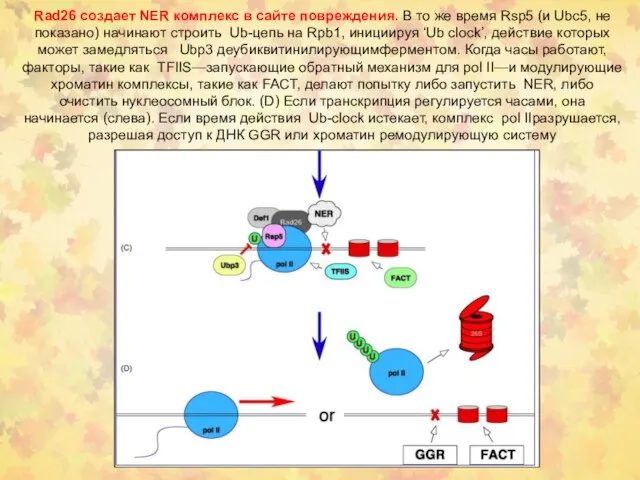
- 34. Rad26 создает NER комплекс в сайте повреждения. В то же время Rsp5 (и Ubc5, не показано)
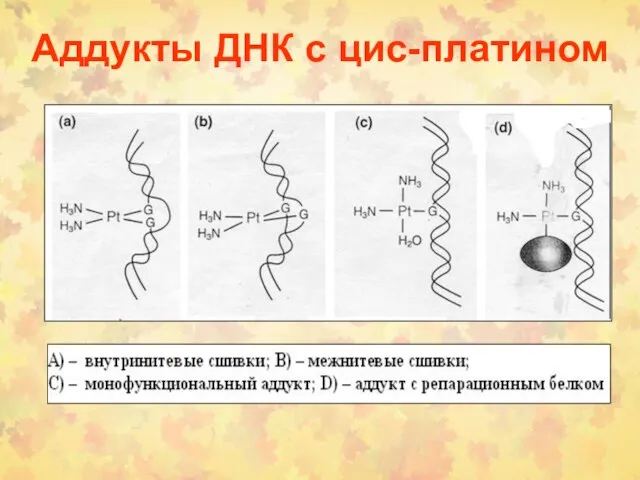
- 35. Аддукты ДНК с цис-платином
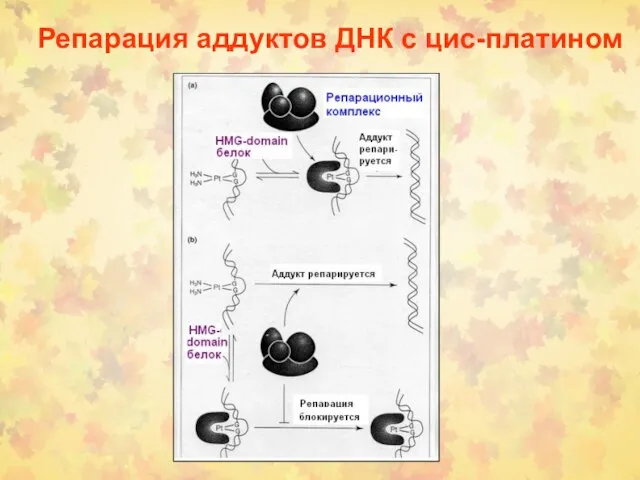
- 36. Репарация аддуктов ДНК с цис-платином
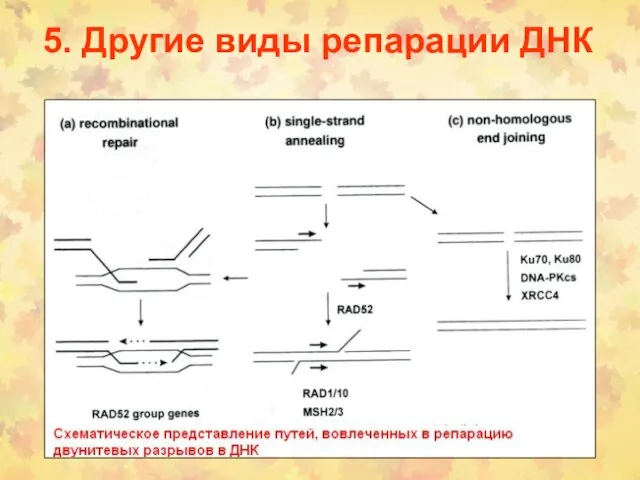
- 37. 5. Другие виды репарации ДНК
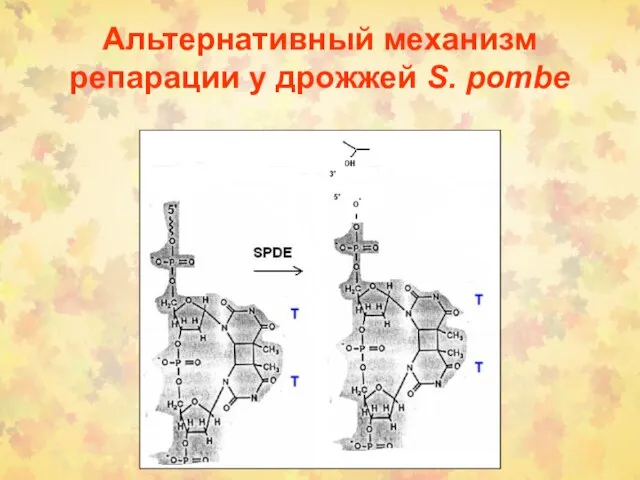
- 38. Альтернативный механизм репарации у дрожжей S. pombe
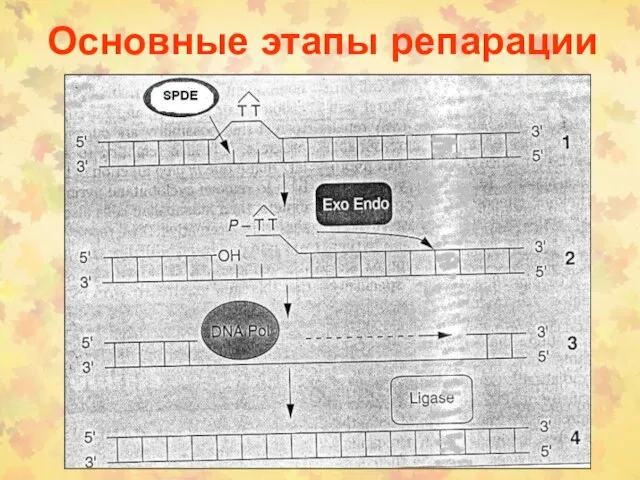
- 39. Основные этапы репарации
- 40. Белки ATM ATM (="ataxia telangiectasia mutated") получила название от болезни, пациенты, среди прочего, имеют высокий риск
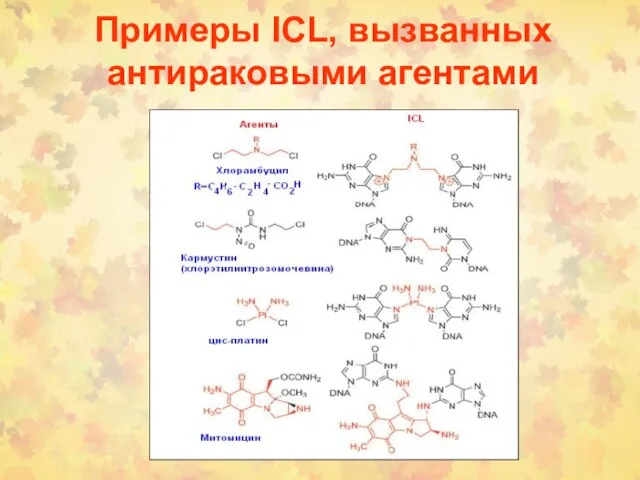
- 41. Примеры ICL, вызванных антираковыми агентами
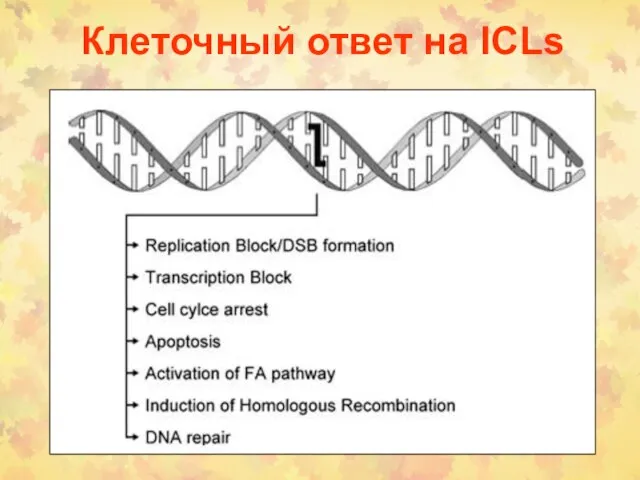
- 42. Клеточный ответ на ICLs
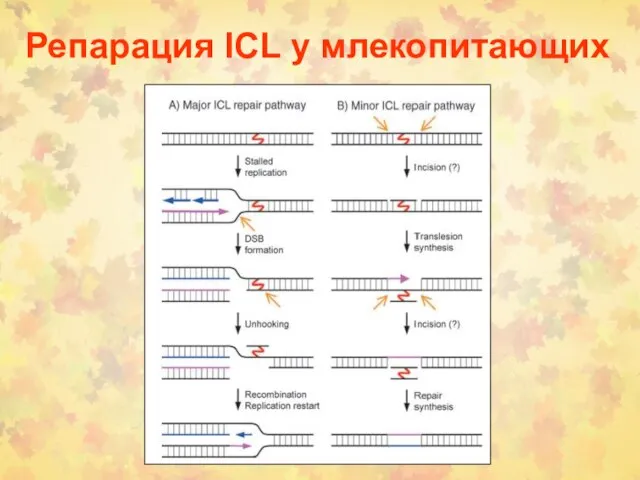
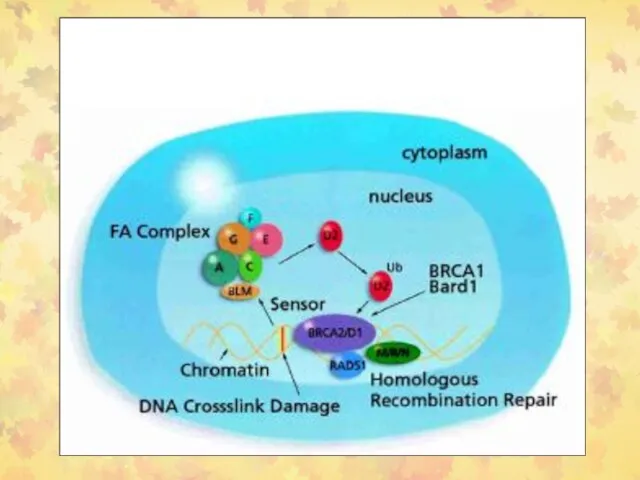
- 43. Репарация ICL у млекопитающих
- 44. Fanconi Anemia (FA)путь репарации У пациентов с FA повреждено, по крайней мере, 13 генов: FANCA, B,
- 45. Сравнение FA генов у человека, Drosophila, Dictyostelium and C. elegans
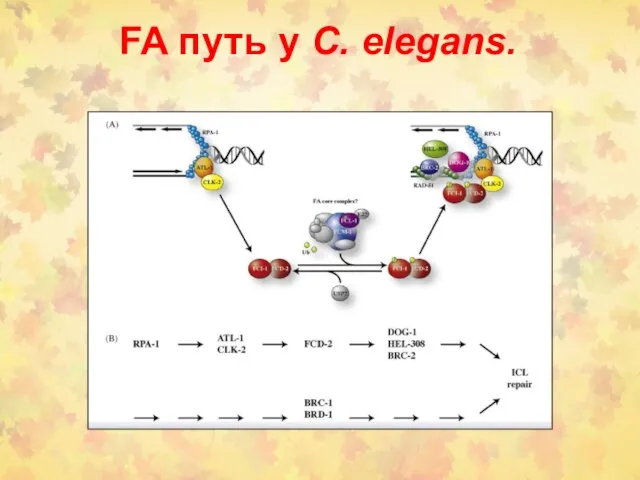
- 46. FA путь у C. elegans.
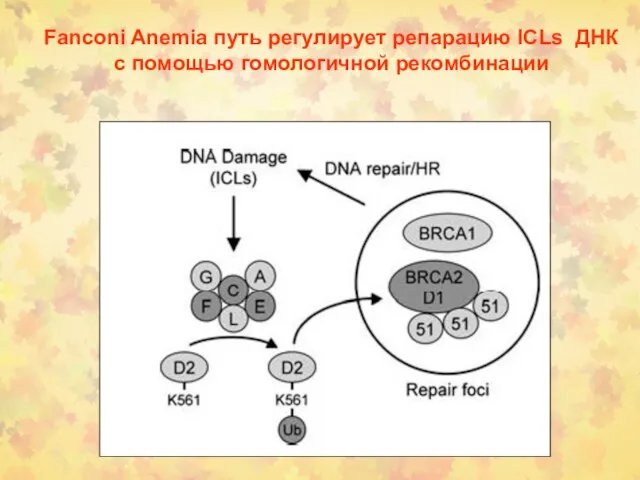
- 47. Fanconi Anemia путь регулирует репарацию ICLs ДНК с помощью гомологичной рекомбинации
- 49. SUMO (small ubiquitin-related modifier) конъюгация Несколько SUMO E3 лигаз выявлено: SP-RING (secretory protein with a RING
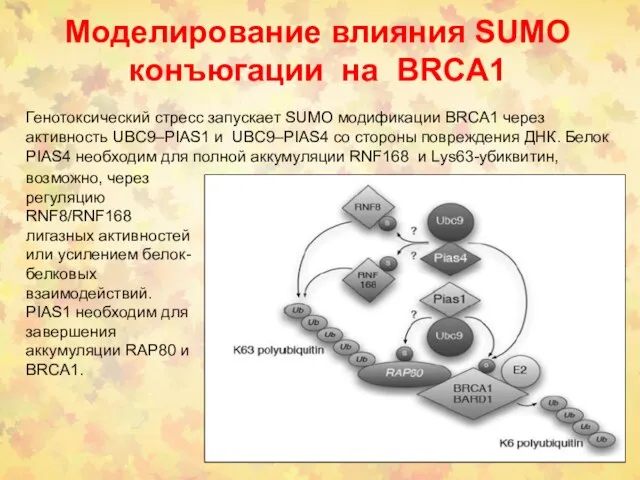
- 50. Моделирование влияния SUMO конъюгации на BRCA1 Генотоксический стресс запускает SUMO модификации BRCA1 через активность UBC9–PIAS1 и
- 51. IY. Эпигенетические модификации ДНК Модификации хроматина, Метилирование ДНК, Геномный импринтинг.
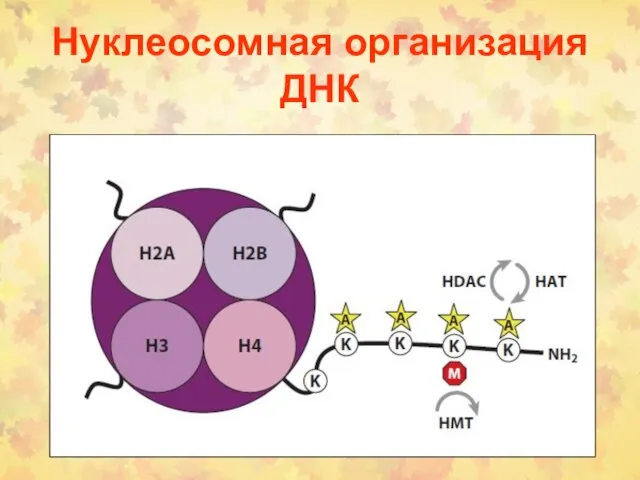
- 52. Нуклеосомная организация ДНК
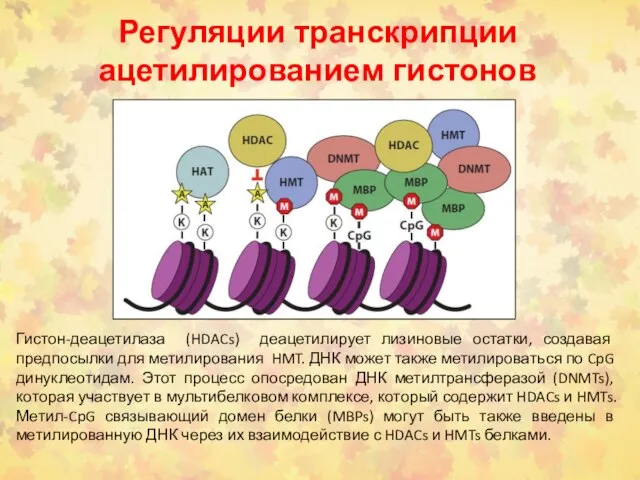
- 53. Регуляции транскрипции ацетилированием гистонов Гистон-деацетилаза (HDACs) деацетилирует лизиновые остатки, создавая предпосылки для метилирования HMT. ДНК может
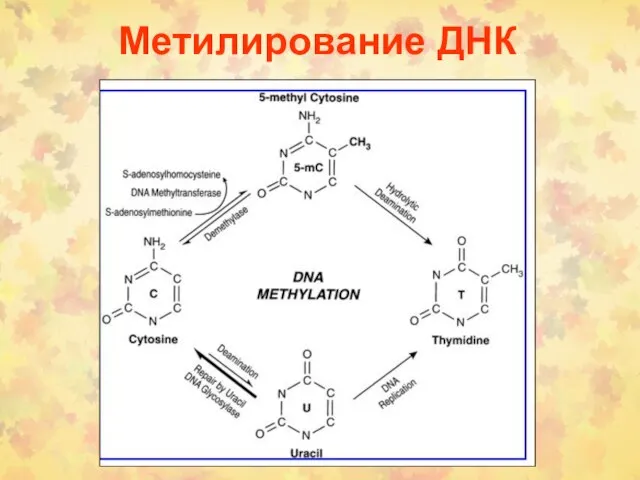
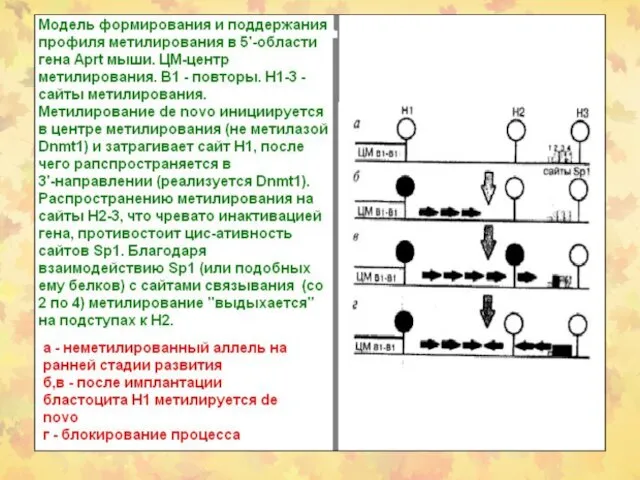
- 54. Метилирование ДНК
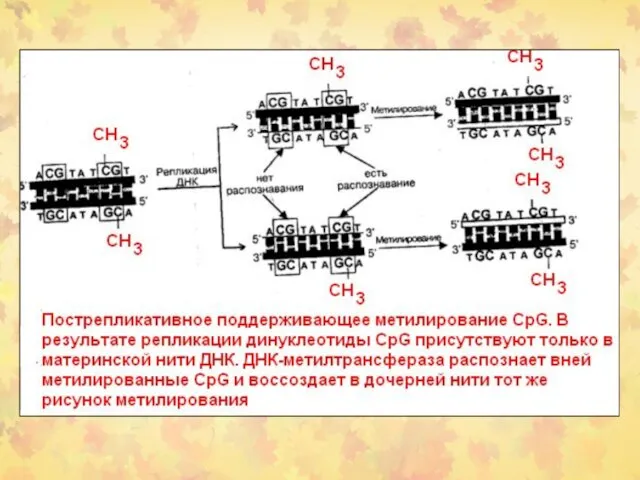
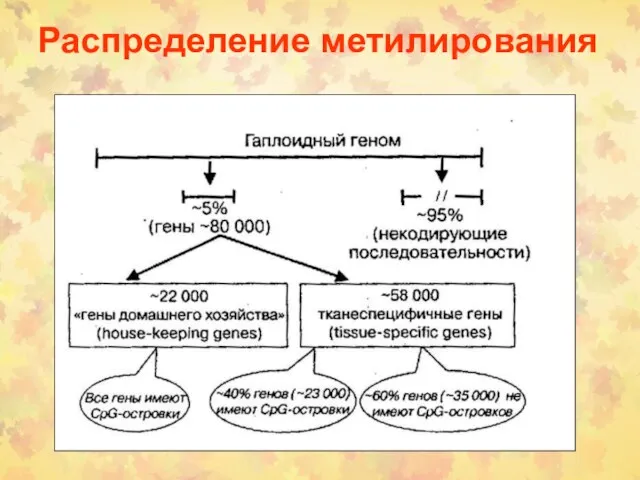
- 56. Распределение метилирования
- 57. Статус метилирования и функциональные особенности промоторов, содержащих CpG-островки
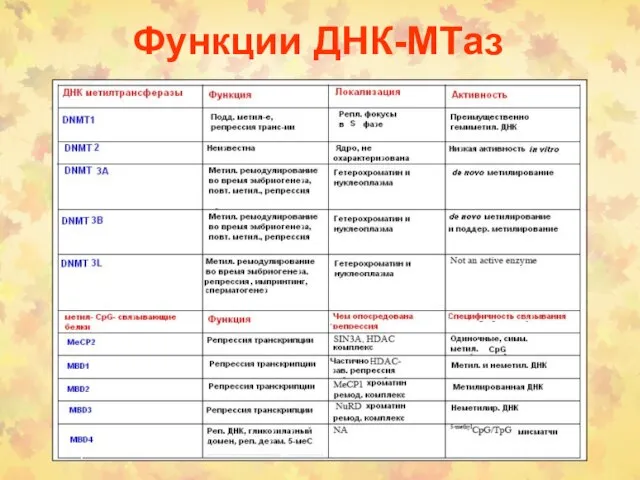
- 59. Функции ДНК-МТаз
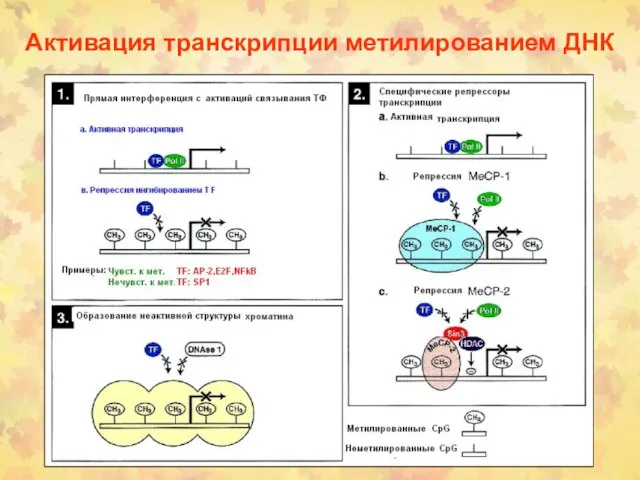
- 60. Активация транскрипции метилированием ДНК
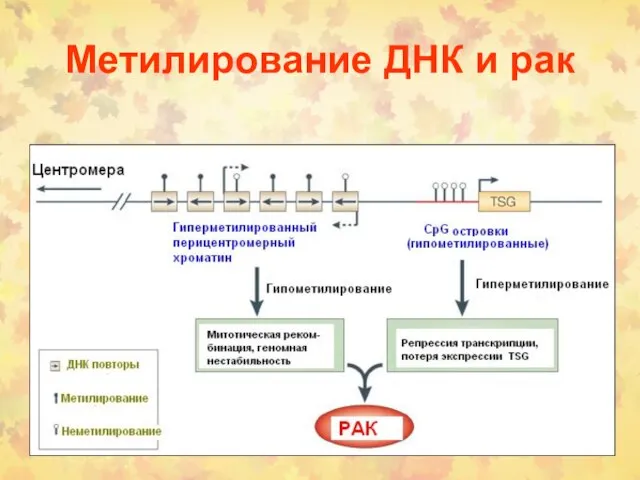
- 62. Метилирование ДНК и рак
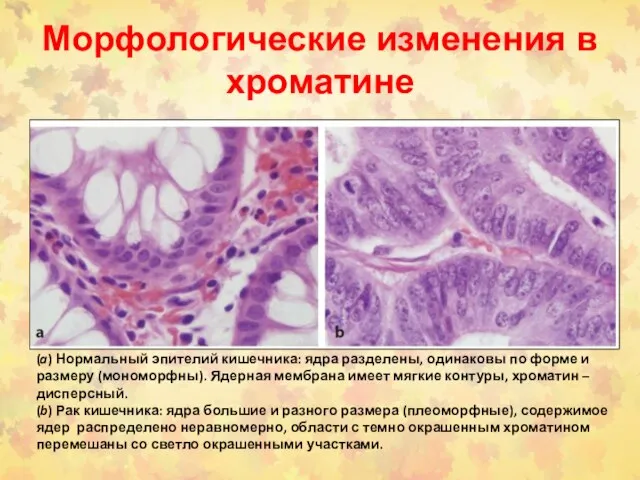
- 63. Морфологические изменения в хроматине (a) Нормальный эпителий кишечника: ядра разделены, одинаковы по форме и размеру (мономорфны).
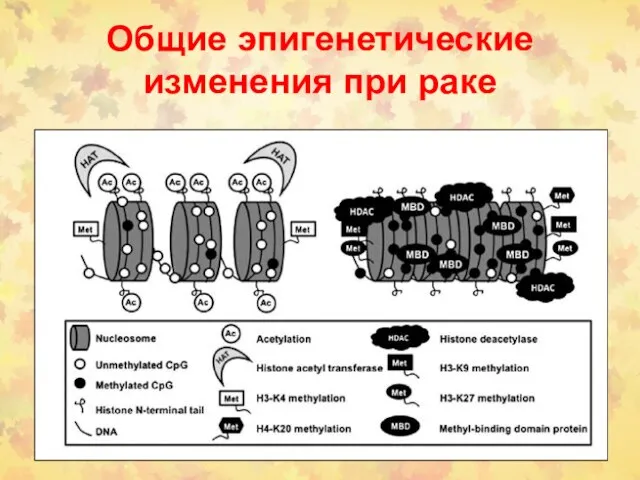
- 64. Общие эпигенетические изменения при раке
- 65. Метилирование ДНК и рак
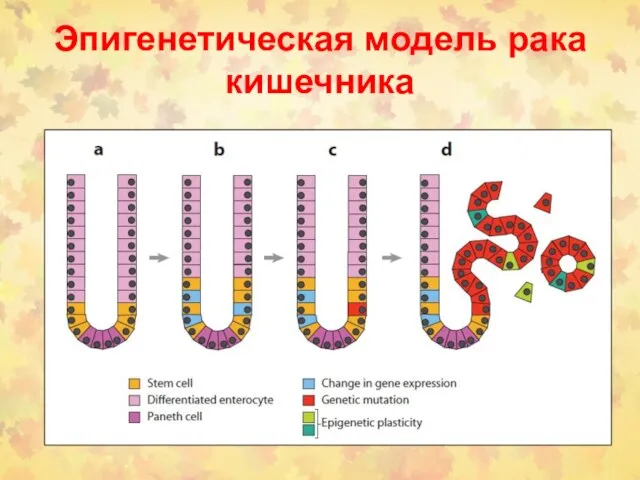
- 66. Эпигенетическая модель рака кишечника
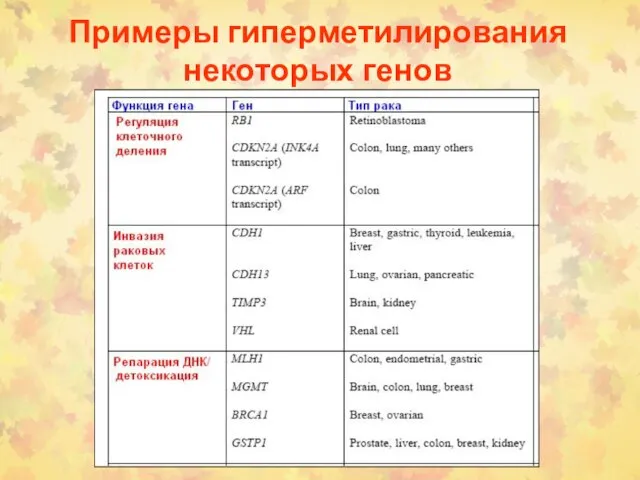
- 67. Примеры гиперметилирования некоторых генов
- 68. Примеры гиперметилирования некоторых генов
- 70. Скачать презентацию






















 Разработка алгоритма (программы), содержащей оператор цикла
Разработка алгоритма (программы), содержащей оператор цикла Дальнейшая интеграция в рамках таможенного союза России, Беларуси и Казахстана и ее влияние на бизнес
Дальнейшая интеграция в рамках таможенного союза России, Беларуси и Казахстана и ее влияние на бизнес Мода и здоровье в современном мире
Мода и здоровье в современном мире План деятельности по оздоровлению и пропаганде здорового образа жизни
План деятельности по оздоровлению и пропаганде здорового образа жизни Средневековье. Готика.
Средневековье. Готика. Ошибки и потенциал медийной рекламы
Ошибки и потенциал медийной рекламы Традиции и интерпретации. Вышивка по дереву
Традиции и интерпретации. Вышивка по дереву Тема «Законы XII таблиц»
Тема «Законы XII таблиц»  КРО: организационно-педагогические аспекты
КРО: организационно-педагогические аспекты Презентация на тему Гигиена сердечно – сосудистой системы
Презентация на тему Гигиена сердечно – сосудистой системы  Библиотека МКОУ СОШ с.Онот
Библиотека МКОУ СОШ с.Онот Перпендикулярность прямых и плоскостей (10 класс)
Перпендикулярность прямых и плоскостей (10 класс) Воланд
Воланд Капитализация насилия
Капитализация насилия Разнообразие птиц
Разнообразие птиц Информационные знаки
Информационные знаки Лингвориторическая парадигма: теоретические и прикладные аспекты
Лингвориторическая парадигма: теоретические и прикладные аспекты Презентация на тему Политическая раздробленность на Руси (XII - начало XIII вв.)
Презентация на тему Политическая раздробленность на Руси (XII - начало XIII вв.) Дневник шоппера
Дневник шоппера Kate’s Top 10 Tips to learn a foreign
Kate’s Top 10 Tips to learn a foreign Устав изготовителей скобяных изделий, 1612 г. Париж
Устав изготовителей скобяных изделий, 1612 г. Париж Работа Редколлегии. Выпуск Стен-газеты
Работа Редколлегии. Выпуск Стен-газеты Ознакомительная презентация по возможным направлениям сотрудничества Юдин Вадим Викторович 8-905-724-22-90 vadimu80@mail
Ознакомительная презентация по возможным направлениям сотрудничества Юдин Вадим Викторович 8-905-724-22-90 vadimu80@mail А.А. Фет (1820-1892)
А.А. Фет (1820-1892) Урок русского языка по теме «Синтаксис и пунктуация». «Город, опоясанный мостами»
Урок русского языка по теме «Синтаксис и пунктуация». «Город, опоясанный мостами» Таймвайз. Регенерирующая сыворотка + С
Таймвайз. Регенерирующая сыворотка + С Русские и немцы глазами друг друга
Русские и немцы глазами друг друга Глагол БЫТЬ
Глагол БЫТЬ