Слайд 2разные сети
белок-белковые взаимодействия
регуляторные сети (фактор-ген)
метаболические

Слайд 3свойства сетей
N = количество вершин
распределение степеней вершин
P(k) = вероятность того, что

у случайно взятой вершины будет k ребер
средняя длина пути между вершинами L
Слайд 4случайная сеть
пуассоновское распределение
P(k) = exp(-λ) λk / k!
Теорема Эрдеша-Реньи: фазовый переход

– возникновение гигантской компоненты
средняя длина пути ~ log N
Слайд 5scale-free network
P(k) ~ k–γ
γ>3 – ничего особенного
2<γ<3 – hubs, иерархия

γ=2 большой hub, соединенный с большой долей вершин
При γ<3 удаление случайной вершины не разрушает сеть, удаление hub’а – разрушает
средняя длина пути (при 2<γ<3) ~ log log N
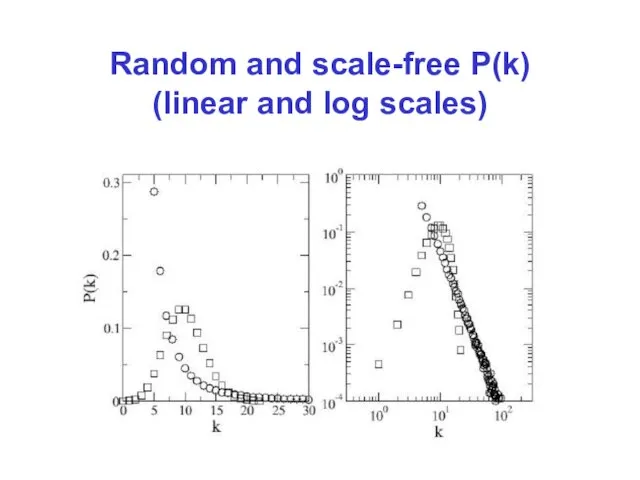
Слайд 6Random and scale-free P(k)
(linear and log scales)
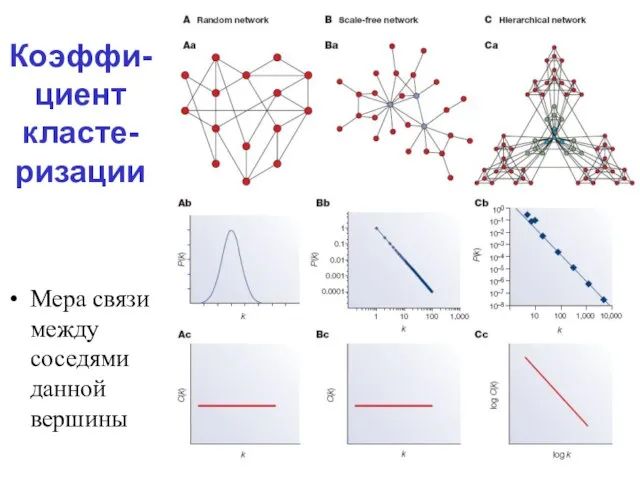
Слайд 7Коэффи-циент класте-ризации
Мера связи между соседями данной вершины
Слайд 8примеры
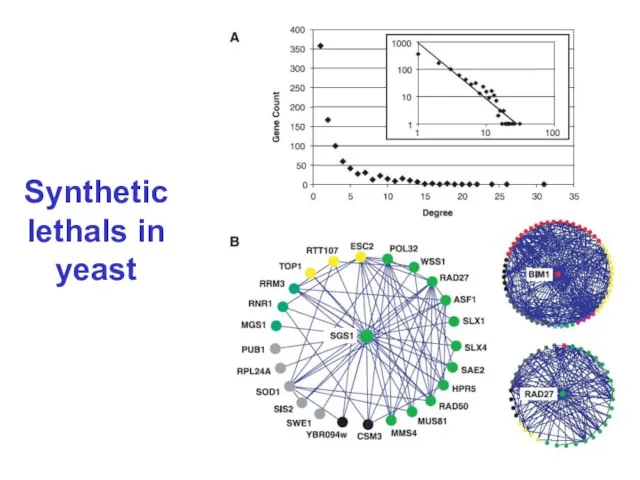
белок-белковые взаимодействия
синтетические летали
регуляция транскрипции
метаболические сети

Слайд 9Yeast protein interaction network
Data from the high-throughput two-hybrid experiment (T. Ito,
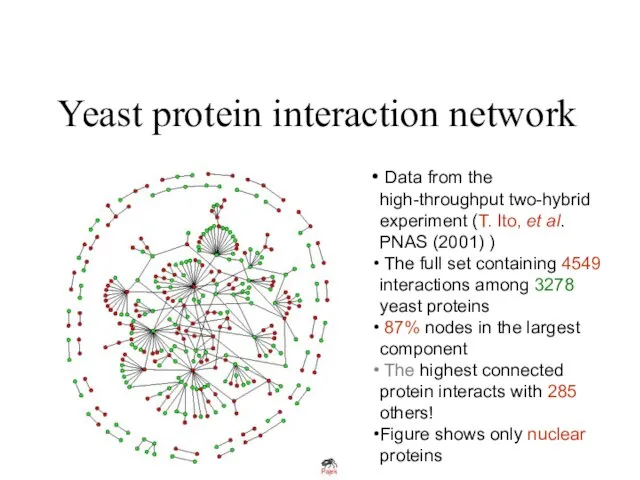
et al. PNAS (2001) )
The full set containing 4549 interactions among 3278 yeast proteins
87% nodes in the largest component
The highest connected protein interacts with 285 others!
Figure shows only nuclear proteins
Слайд 11Гигантская компонента в графе белок-белковых взаимодействий в дрожжах
Красный – летальная мутация
Оранжевый –
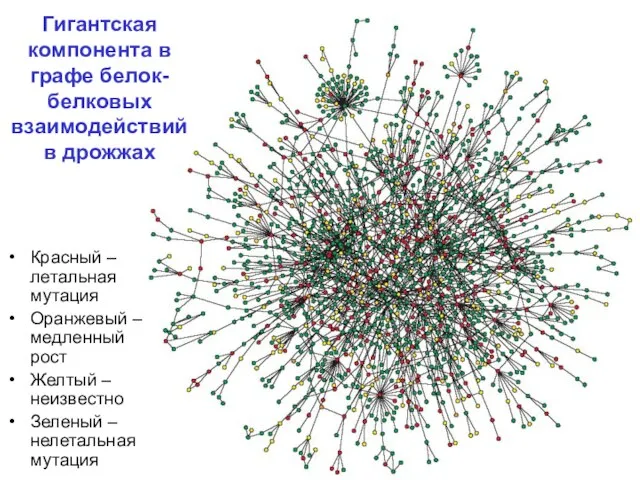
медленный рост
Желтый – неизвестно
Зеленый – нелетальная мутация
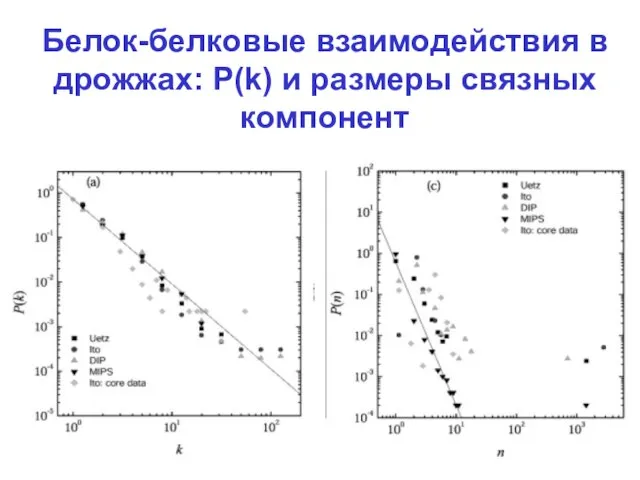
Слайд 12Белок-белковые взаимодействия в дрожжах: P(k) и размеры связных компонент
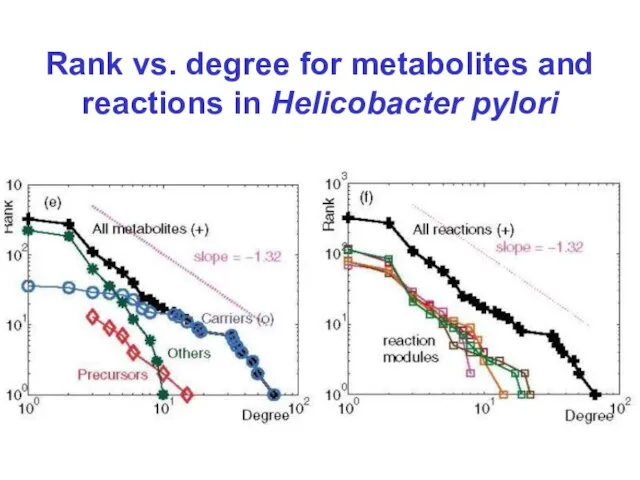
Слайд 14Rank vs. degree for metabolites and reactions in Helicobacter pylori
Слайд 15Transcription regulatory
network in baker’s yeast
Downloaded from the
YPD database: 1276
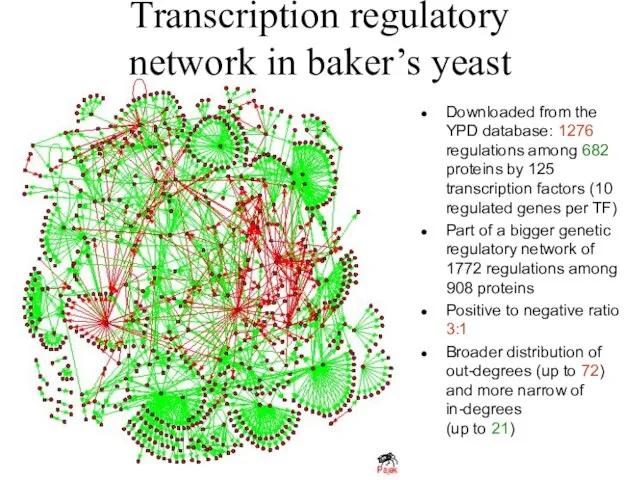
regulations among 682 proteins by 125 transcription factors (10 regulated genes per TF)
Part of a bigger genetic regulatory network of 1772 regulations among 908 proteins
Positive to negative ratio 3:1
Broader distribution of out-degrees (up to 72) and more narrow of in-degrees
(up to 21)
Слайд 16регуляция транскрипции (дрожжи, ChIP-chip)
A: in-degree (относительно регулируемых генов): гистограмма (в полулогарифмических координатах)
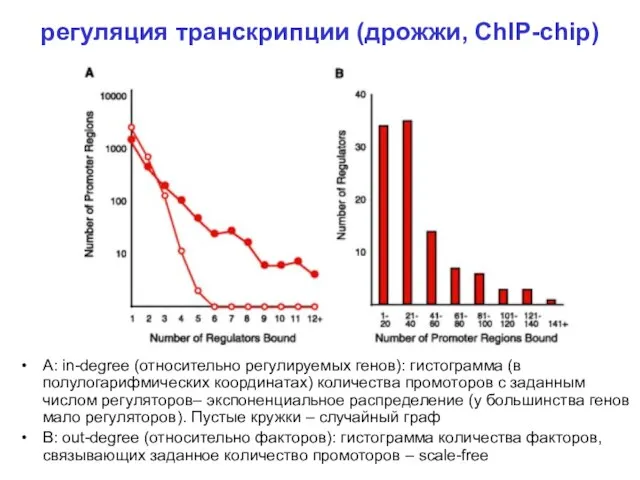
количества промоторов с заданным числом регуляторов– экспоненциальное распределение (у большинства генов мало регуляторов). Пустые кружки – случайный граф
В: out-degree (относительно факторов): гистограмма количества факторов, связывающих заданное количество промоторов – scale-free
Слайд 17Transcription regulatory
network in Homo Sapiens
Data courtesy of Ariadne Genomics obtained
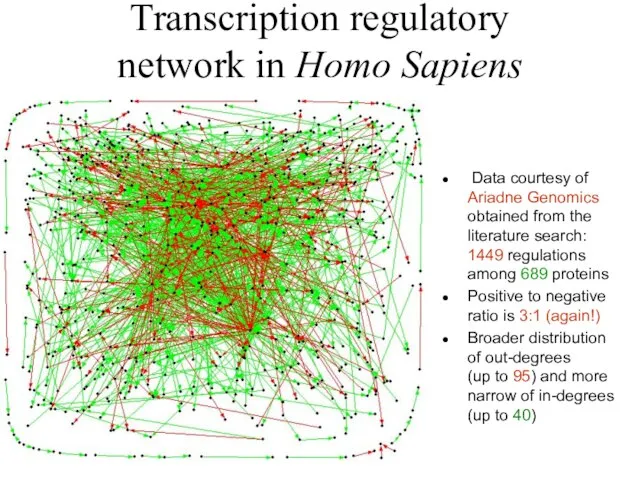
from the literature search: 1449 regulations among 689 proteins
Positive to negative ratio is 3:1 (again!)
Broader distribution of out-degrees
(up to 95) and more narrow of in-degrees (up to 40)
Слайд 18Transcription regulatory
network in E. coli
Data (courtesy of Uri Alon) was
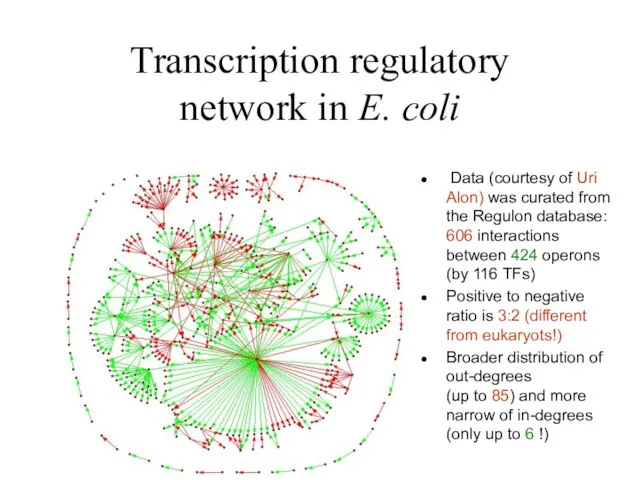
curated from the Regulon database: 606 interactions between 424 operons (by 116 TFs)
Positive to negative ratio is 3:2 (different from eukaryots!)
Broader distribution of out-degrees
(up to 85) and more narrow of in-degrees (only up to 6 !)
Слайд 19зависимость физиологических и геномных свойств от топологии
дрожжи:
~10% genes with <5 links are

essential
>60% genes with >15 links are essential
гены с большим числом связей
с большей вероятностью имеют ортологов в многоклеточных эукариотах
ближе к ортологам из C. elegans
Слайд 20party hubs и date hubs
Бимодальное распределение корреляций уровня экспрессии
Красный: hubs
Голубой: non-hubs
Черный: случайный
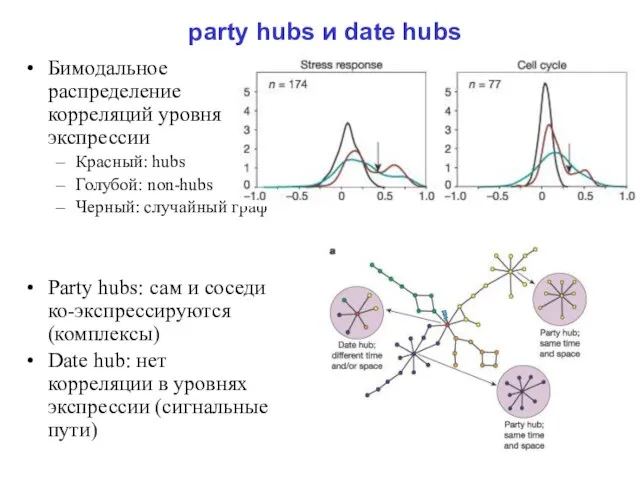
граф
Party hubs: сам и соседи ко-экспрессируются (комплексы)
Date hub: нет корреляции в уровнях экспрессии (сигнальные пути)
Слайд 21Устойчивость к атаке
(распадение гигантской компоненты)
основа сети
– party hubs
Красный: атака на
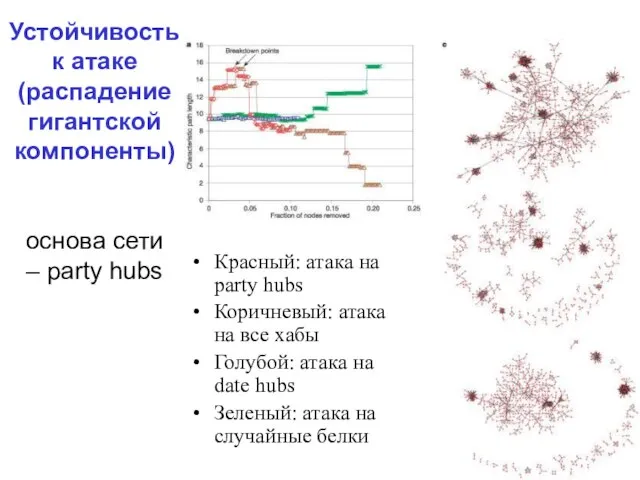
party hubs
Коричневый: атака на все хабы
Голубой: атака на date hubs
Зеленый: атака на случайные белки
Слайд 22мотивы
клики
много в графах белок-белковых взаимодействий (масс-спек. анализ комплексов – по определению)
подграфы фиксированной

структуры, встречающиеся существенно чаще, чем в случайном графе (с теми же свойствами)
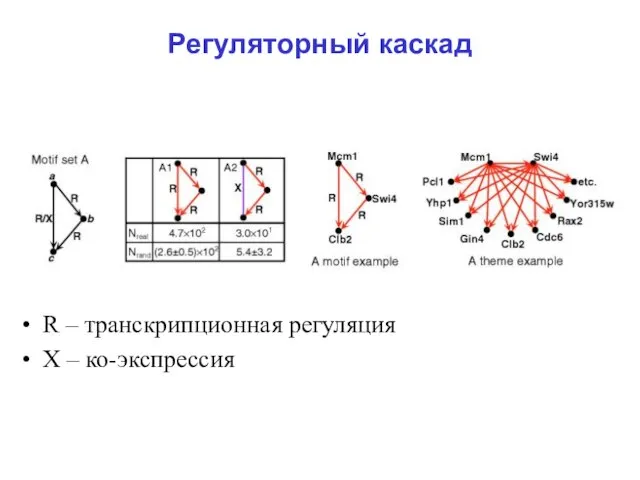
Слайд 23Регуляторный каскад
R – транскрипционная регуляция
Х – ко-экспрессия
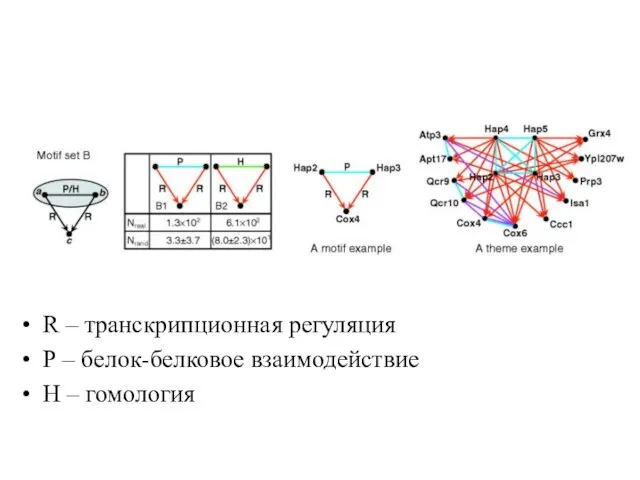
Слайд 24R – транскрипционная регуляция
Р – белок-белковое взаимодействие
Н – гомология
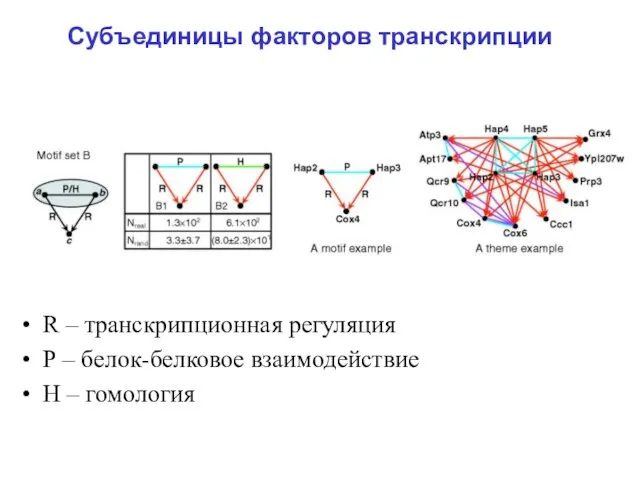
Слайд 25Субъединицы факторов транскрипции
R – транскрипционная регуляция
Р – белок-белковое взаимодействие
Н – гомология
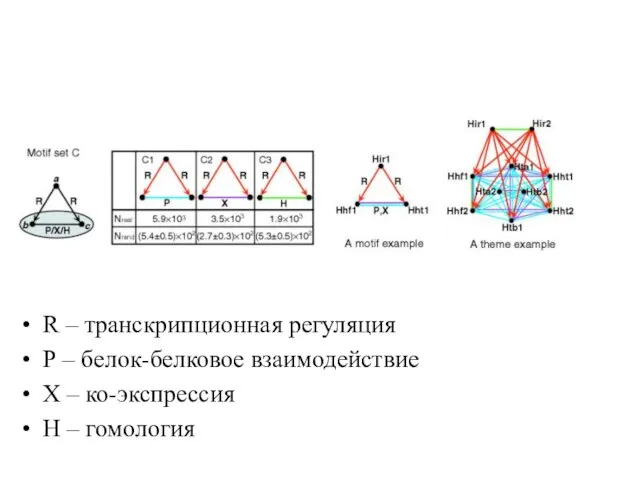
Слайд 26R – транскрипционная регуляция
Р – белок-белковое взаимодействие
Х – ко-экспрессия
Н – гомология
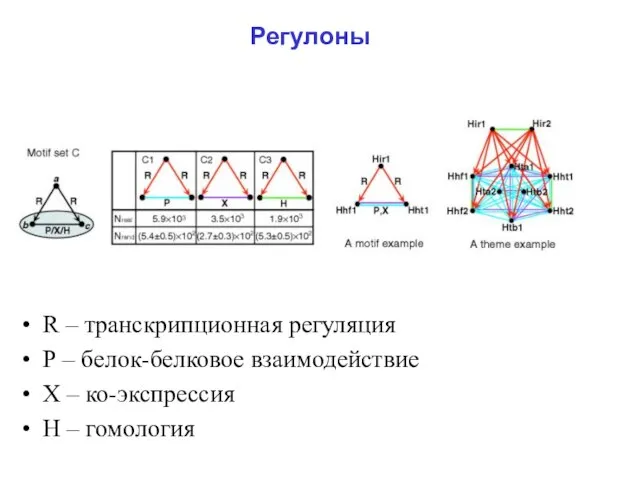
Слайд 27Регулоны
R – транскрипционная регуляция
Р – белок-белковое взаимодействие
Х – ко-экспрессия
Н – гомология
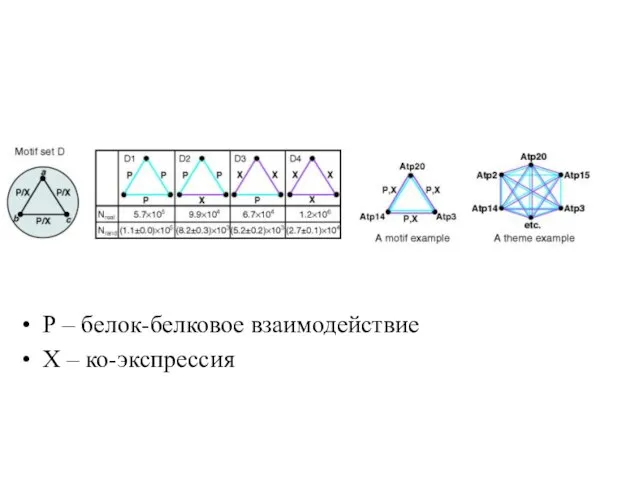
Слайд 28Р – белок-белковое взаимодействие
Х – ко-экспрессия
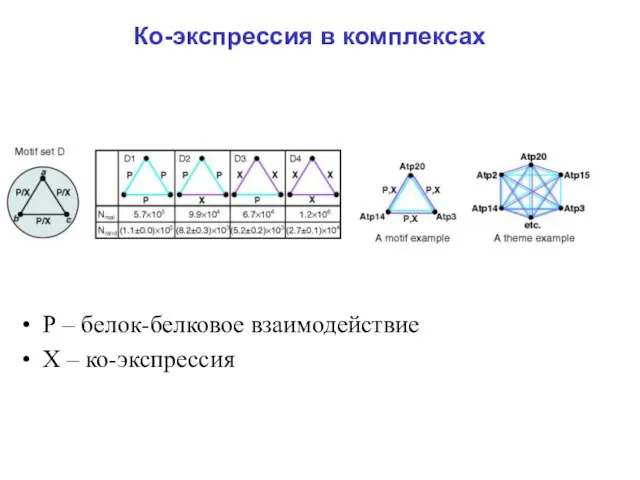
Слайд 29Ко-экспрессия в комплексах
Р – белок-белковое взаимодействие
Х – ко-экспрессия
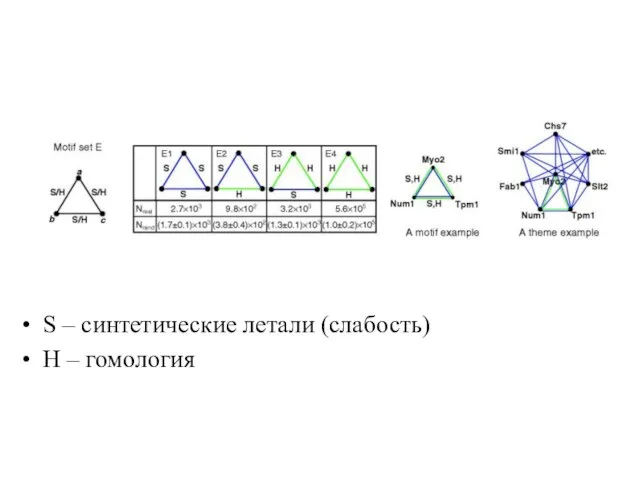
Слайд 30S – синтетические летали (слабость)
Н – гомология
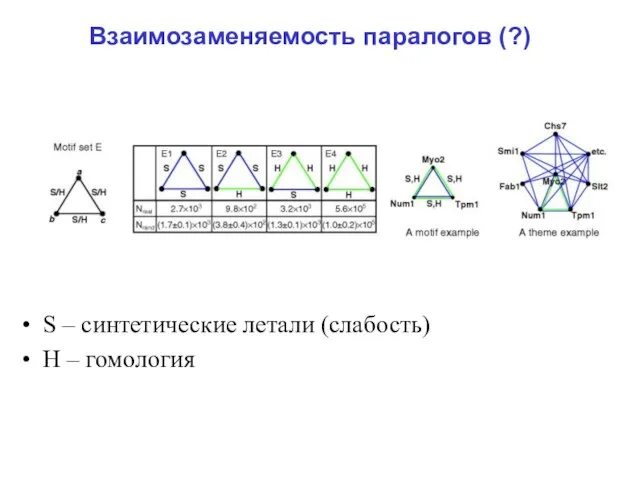
Слайд 31Взаимозаменяемость паралогов (?)
S – синтетические летали (слабость)
Н – гомология
Слайд 32Компенсаторные комплексы (?)
S – синтетические летали (слабость)
Н – гомология
Р – белок-белковое взаимодействие
Х
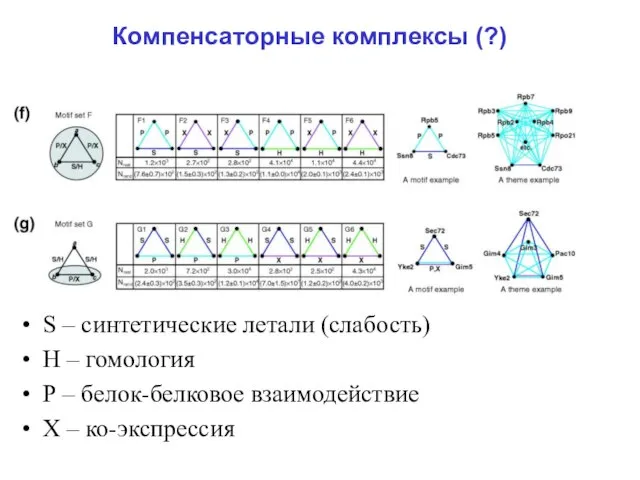
– ко-экспрессия
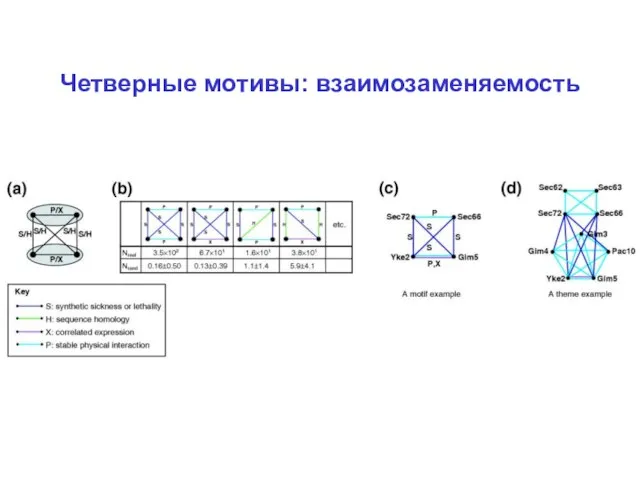
Слайд 33Четверные мотивы: взаимозаменяемость
Слайд 34Почти все “bi-fan” мотивы связаны друг с другом
Регуляция транскрипции в E.coli
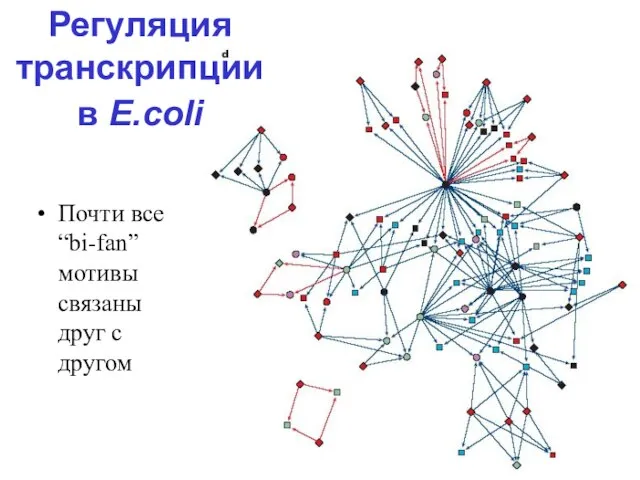
Слайд 35эволюция
rich get richer
дупликации
случайные рождения/исчезновение ребер


































 Всё о профессии ХИРУРГА
Всё о профессии ХИРУРГА Стабилизационный фонд РФ
Стабилизационный фонд РФ Русская трапеза
Русская трапеза Star Hour
Star Hour Бизнес-план антикафе Час за минуту
Бизнес-план антикафе Час за минуту Разработка алгоритмов
Разработка алгоритмов  Урок истории в 5 классе по теме: «Поход Александра Македонского на восток»
Урок истории в 5 классе по теме: «Поход Александра Македонского на восток» Жанры изобразительного искусства
Жанры изобразительного искусства Подготовка к ЕГЭ (задание В7).» Презентация по математике для урока по теме: «Нахождение значения выражения
Подготовка к ЕГЭ (задание В7).» Презентация по математике для урока по теме: «Нахождение значения выражения Терморегуляторы. Комплект для управления снеготаянием
Терморегуляторы. Комплект для управления снеготаянием Федеральная миграционнная служба
Федеральная миграционнная служба Цветковые растения
Цветковые растения  Технология формирования диагностических навыков у обучающихся
Технология формирования диагностических навыков у обучающихся ОСЕВАЯ И ЦЕНТРАЛЬНАЯ Симметрии
ОСЕВАЯ И ЦЕНТРАЛЬНАЯ Симметрии Подведение итогов
Подведение итогов Золотые правила питания
Золотые правила питания Нижний Новгород 15 февраля 2012 г.
Нижний Новгород 15 февраля 2012 г. Лист — боковой орган побега
Лист — боковой орган побега Слагаемые успеха
Слагаемые успеха Поделки из вторсырья
Поделки из вторсырья Исследование демографической ситуации села Бала Верхоянского района Республики Саха (Якутия) по половозрастному составу
Исследование демографической ситуации села Бала Верхоянского района Республики Саха (Якутия) по половозрастному составу У истоков русской журналистики и журналистского образования в Санкт-Петербургском государственном университете
У истоков русской журналистики и журналистского образования в Санкт-Петербургском государственном университете Литературный турнир по произведениям русских писателей 20 века
Литературный турнир по произведениям русских писателей 20 века ПРЕЗЕНТАЦИЯ на QS-форуме КООМЕТ СИСТЕМЫ МЕНЕДЖМЕНТА КАЧЕСТВА ВНИИФТРИ
ПРЕЗЕНТАЦИЯ на QS-форуме КООМЕТ СИСТЕМЫ МЕНЕДЖМЕНТА КАЧЕСТВА ВНИИФТРИ Кадровый состав. В школе работает 24 педагога До 5 летОт 5 до10летОт10-15летБолее15лет 40119 Имеют категории высшуюпервуювторуюНе имеют ка
Кадровый состав. В школе работает 24 педагога До 5 летОт 5 до10летОт10-15летБолее15лет 40119 Имеют категории высшуюпервуювторуюНе имеют ка Путешествие на поезде «Здоровье» - презентация для начальной школы
Путешествие на поезде «Здоровье» - презентация для начальной школы Презентация на тему Хлеб - драгоценность
Презентация на тему Хлеб - драгоценность Особенности скелета человека связанные с прямохождением и трудовой деятельностью
Особенности скелета человека связанные с прямохождением и трудовой деятельностью