Содержание
- 2. BLAST – алгоритм для нахождения участков локального сходства между последовательностями. Алгоритм сравнивает входную последовательность с последовательностями
- 3. Почему локальное выравнивание? Глобальное выравнивание следует применять только в случае заранее известной гомологии последовательностей по всей
- 4. Protein BLAST: поиск гомологов данного белка в банке аминокислотных последовательностей Алгоритмы blastp psi-blast phi-blast Можно использовать:
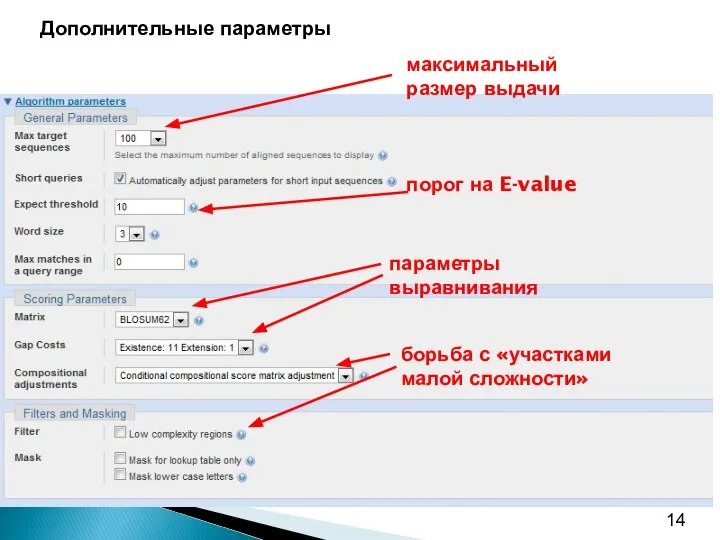
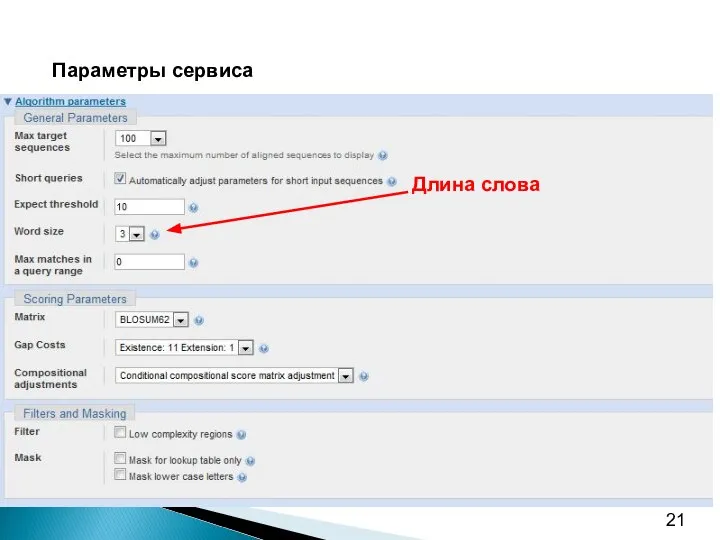
- 5. Что подаётся на вход программе BLAST? Последовательность запроса Банк последовательностей Параметры: параметры выравнивания: матрица аминокислотных замен,
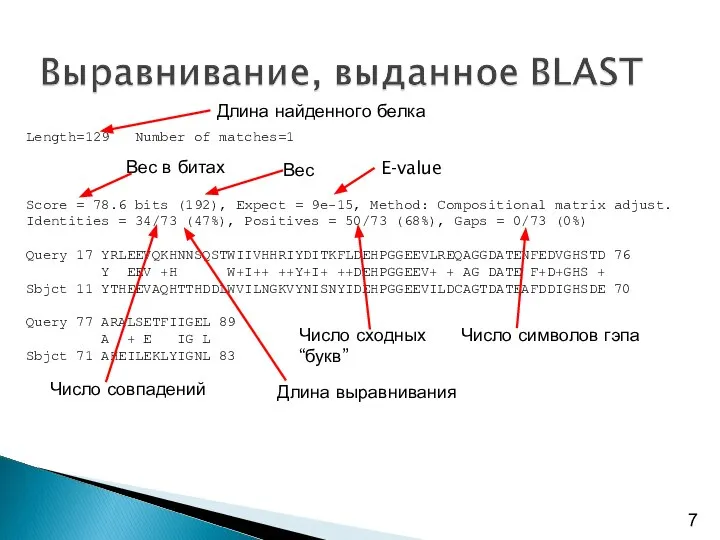
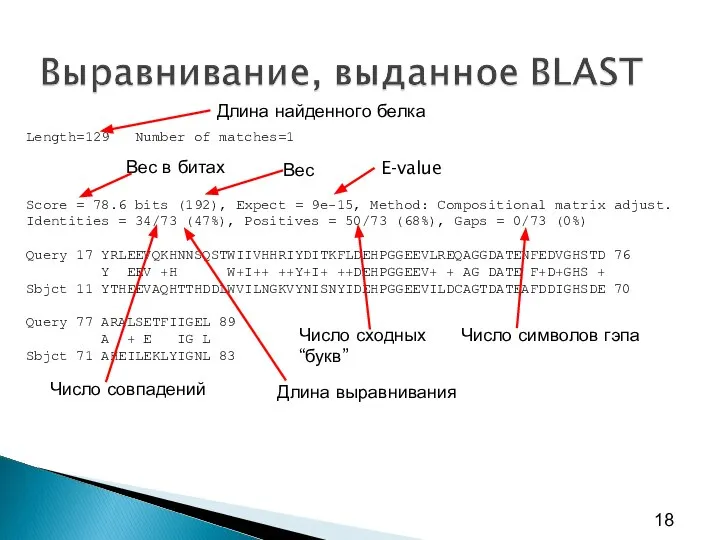
- 6. Что выдает BLAST? Выдача самой программы состоит из четырёх частей: – заголовок с описанием программы, банка,
- 8. E-value – ожидаемое количество случайных находок с таким же и лучшим весом (в той же базе
- 9. Как посчитать E-value Прямой способ — вычислительный эксперимент: перемешать банк (или запрос) очень много раз, каждый
- 10. Как посчитать E-value Имеется замечательная теорема (С.Карлина): E-value=Kmn·e-λS S – Score (вес) m – длина исходной
- 11. Вес в битах Вес в битах B зависит от обычного веса S и параметров вычисления веса.
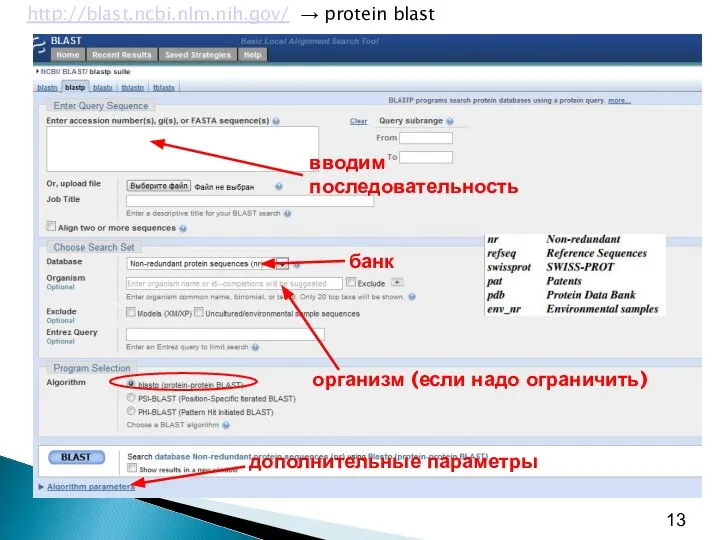
- 12. Здесь описан интерфейс, установленный на «родине» BLAST: National Center for Biotechnology Information (NCBI) в США, http://blast.ncbi.nlm.nih.gov/
- 13. http://blast.ncbi.nlm.nih.gov/ → protein blast
- 15. Участок малой сложности Ищем: белок P02929 если отключить “Compositional adjustment” и фильтр, то одной из находок
- 16. Определяется как участок с смещенным составом (biased composition) • Гомополимерные участки • Короткие повторы • Перепредставленность
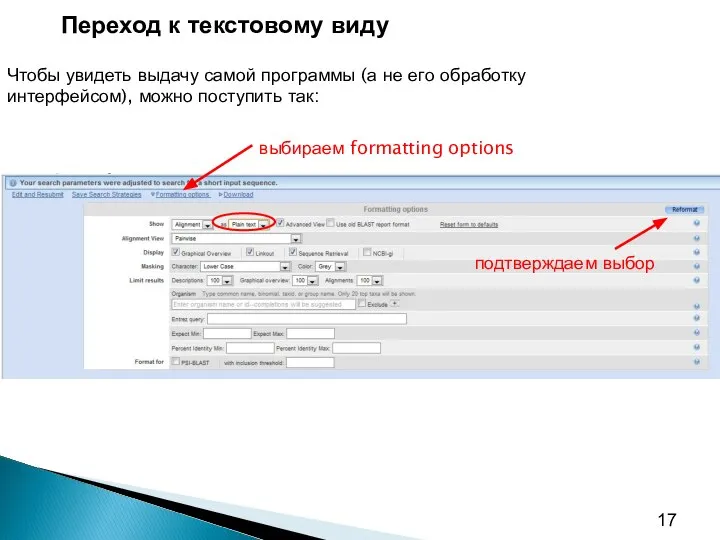
- 17. выбираем formatting options подтверждаем выбор Переход к текстовому виду Чтобы увидеть выдачу самой программы (а не
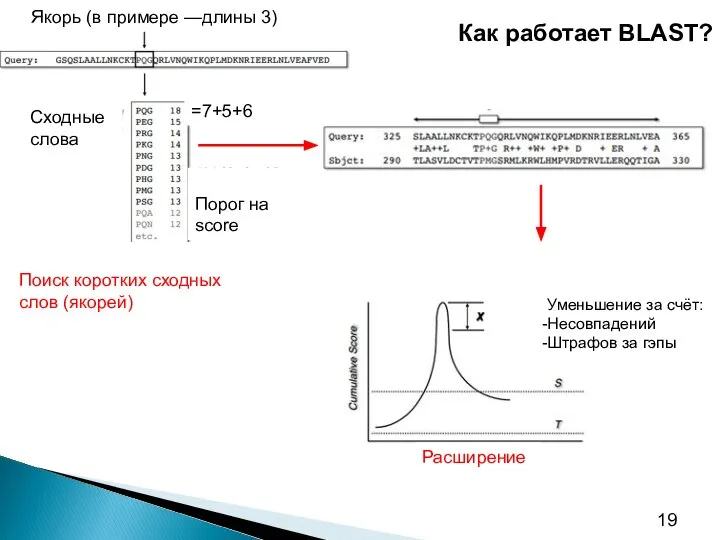
- 19. Как работает BLAST? Поиск коротких сходных слов (якорей) Якорь (в примере —длины 3) Сходные слова Порог
- 20. BLAST — эвристический алгоритм Алгоритмы биоинформатики можно разделить на точные и эвристические. Точные алгоритмы решают какую-либо
- 23. Скачать презентацию













 Словари. Методические указания
Словари. Методические указания Работа в Photoshop. Из фото в гравюру. Задание 2
Работа в Photoshop. Из фото в гравюру. Задание 2 Шаблон паспорта проектной идеи
Шаблон паспорта проектной идеи Постановка целей поста
Постановка целей поста Partnership program
Partnership program Алгоритмические языки и программирование
Алгоритмические языки и программирование Измерение информации
Измерение информации Взаимоотношение имени переменной и её адреса в ОП
Взаимоотношение имени переменной и её адреса в ОП Иерархия классов
Иерархия классов Poddotazy
Poddotazy Компьютерные вирусы и антивирусы
Компьютерные вирусы и антивирусы Макет здания
Макет здания Построение сети FTTB
Построение сети FTTB Финальный проект на Mit App Inventor
Финальный проект на Mit App Inventor Современные информационные технологии в процессе подготовки специалистов
Современные информационные технологии в процессе подготовки специалистов Макет СНК ПДБ
Макет СНК ПДБ Создание красивых картинок в фотошопе
Создание красивых картинок в фотошопе Типы алгоритмов
Типы алгоритмов Системы управления базами данных
Системы управления базами данных Исследование прохождения амплитудно-модулированного колебания через колебательный контур. Лабораторная работа 3
Исследование прохождения амплитудно-модулированного колебания через колебательный контур. Лабораторная работа 3 Алгоритмы
Алгоритмы Инструкция по получению электроной почты студента
Инструкция по получению электроной почты студента Clubhouse – социальная сеть на голосовом общении
Clubhouse – социальная сеть на голосовом общении История развития информационных технологий. Электронная технология
История развития информационных технологий. Электронная технология Использование информационно-коммуникационных технологий в процессе преподавания химии
Использование информационно-коммуникационных технологий в процессе преподавания химии HART-протокол. Архітектура HART
HART-протокол. Архітектура HART Операционные системы и среды. Лекция 6
Операционные системы и среды. Лекция 6 Современный киберспорт
Современный киберспорт