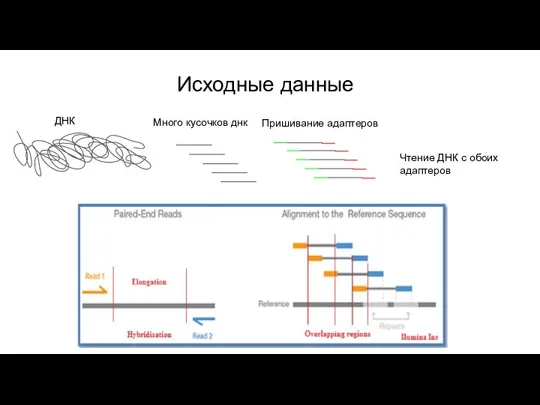
mem -R "@RG\tID:4\tSM:$p\tPL:illumina\tLB:lib1\tPU:unit1" ./Homo_sapiens_assembly38.fasta "$p"_1.fastq "$p"_2.fastq -t 25 > "$p".sam
echo "####################Deleting fastq files###################"
#rm "$p"_1.fastq
#rm "$p"_2.fastq
echo "##################### Converting files to Bam and sort ###################"
/samtools view -Su "$p".sam | samtools sort -@25 - -o "$p".sorted.bam
echo "##############Deleting sam files###############"
rm "$p".sam
echo "#################mark duplicates################"
java -Dpicard.useLegacyParser=false -Xmx16G -jar ../picard.jar MarkDuplicates\
-I "$p".sorted.bam\
-O "$p".MD.bam\
-METRICS_FILE metrics.txt\
-CREATE_INDEX true
echo "#############Deleting sorted.bam################"
rm "$p".sorted.bam
done < samp_list
resist_1
resist_2
resist_3
resist_4
resist_5
resist_6
sensitive_1
sensitive_2
sensitive_3
sensitive_4
sensitive_5
sensitive_6
samp_list







 Аттестационная работа. Проектная деятельность учащихся как средство развития мотивации учения
Аттестационная работа. Проектная деятельность учащихся как средство развития мотивации учения Педагогическое проектирование. Практическое занятие 1
Педагогическое проектирование. Практическое занятие 1 Соціальна робота в родині, де виховується дитина з особливими проблемами
Соціальна робота в родині, де виховується дитина з особливими проблемами Имитационные технологии на уроках иностранного языка
Имитационные технологии на уроках иностранного языка Хәл һәм аның төрләре. Урын хәле
Хәл һәм аның төрләре. Урын хәле Израиль
Израиль Убери лишний слог. Интерактивные задания для мультимедийной доски
Убери лишний слог. Интерактивные задания для мультимедийной доски Олины именины. Автоматизация [ль] в предложениях
Олины именины. Автоматизация [ль] в предложениях Моите очаквани подаръци от Дядо Коледа
Моите очаквани подаръци от Дядо Коледа Оборудование центров элементарного экспериментирования в дошкольных учреждениях
Оборудование центров элементарного экспериментирования в дошкольных учреждениях Сколько слогов
Сколько слогов Развитие навыка чтения
Развитие навыка чтения Мой родной Кореновск
Мой родной Кореновск Игровое дидактическое пособие для детей старшего возраста Каруселька знаний
Игровое дидактическое пособие для детей старшего возраста Каруселька знаний Игрушки из носков
Игрушки из носков Типы деструктивного поведения
Типы деструктивного поведения Изготовление поделок из бумаги. 1 класс
Изготовление поделок из бумаги. 1 класс Праздник 8 марта Мамин День. Развивающие игры
Праздник 8 марта Мамин День. Развивающие игры Портфолио
Портфолио Презентация на тему Организация процесса самообразования в педагогической деятельности учителя
Презентация на тему Организация процесса самообразования в педагогической деятельности учителя  Составляем рассказы дома. Методические рекомендации по развитию речи у детей
Составляем рассказы дома. Методические рекомендации по развитию речи у детей Интеллектуальная игра Эрудиты
Интеллектуальная игра Эрудиты Угадай слова по первым слогам
Угадай слова по первым слогам Месяц февраль. Занятие в детском саду
Месяц февраль. Занятие в детском саду Нетрадиционные формы работы с родителями. Презентация на педсовет
Нетрадиционные формы работы с родителями. Презентация на педсовет Объемное конструирование
Объемное конструирование Деятельность воспитателя детского сада по реализации основной общеобразовательной программы по всем направлениям развития детей
Деятельность воспитателя детского сада по реализации основной общеобразовательной программы по всем направлениям развития детей Учебно-исследовательская и проектная деятельность учащихся на уроках и во внеурочной деятельности при реализации ФГОС
Учебно-исследовательская и проектная деятельность учащихся на уроках и во внеурочной деятельности при реализации ФГОС