Содержание
- 2. ЦЕЛЬ И ЗАДАЧИ Цель: построить модель машинного обучения на основе алгоритма “случайных лесов” (random forest), которая
- 3. АКТУАЛЬНОСТЬ Сердечно-сосудистые заболевания являются основной причиной смерти и инвалидизации во всем мире. Поиск новых способов лечения
- 4. МАТЕРИАЛЫ И МЕТОДЫ Машинное обучение (machine learning, ML) – совокупность методов искусственного интеллекта, позволяющих строить алгоритмы
- 5. Скрипты для обработки данных и построения модели были написаны на языке программирования Python. Для 1D представления
- 6. Для отбора ингибиторов ренина использовалась база данных ChEMBL. Всего было найдено 5154 лиганда. Для дальнейшей работы
- 7. Фингерпринты – представление молекул в виде битовой строки, где каждый бит соответствует наличию (1) либо отсутствию
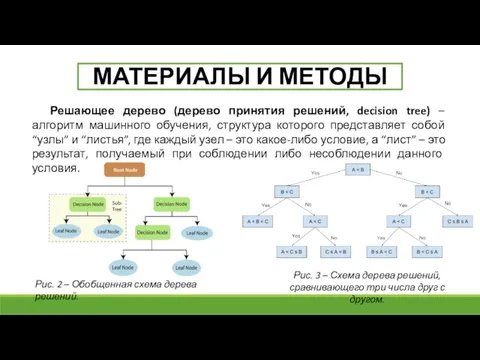
- 8. Решающее дерево (дерево принятия решений, decision tree) – алгоритм машинного обучения, структура которого представляет собой “узлы”
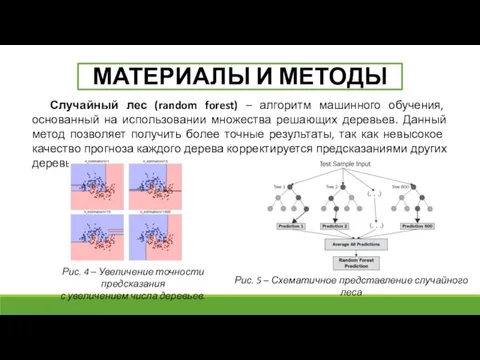
- 9. Случайный лес (random forest) – алгоритм машинного обучения, основанный на использовании множества решающих деревьев. Данный метод
- 10. МАТЕРИАЛЫ И МЕТОДЫ В качестве меры активности молекул использовалась IC50. Для удобства анализа данной меры активности
- 11. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ С помощью метода RandomForestClassifier была построена QSAR модель “случайных лесов”. Обучение модели
- 12. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ Метрики качества модели составили: Точность предсказания на тренировочных данных: 0,9472; Точность предсказания
- 13. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ Конфузионная матрица (confusion matrix) – таблица, представляющая результаты предсказания в сравнении с
- 14. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ По метрикам качества модели можно сделать вывод, что данная модель находит практически
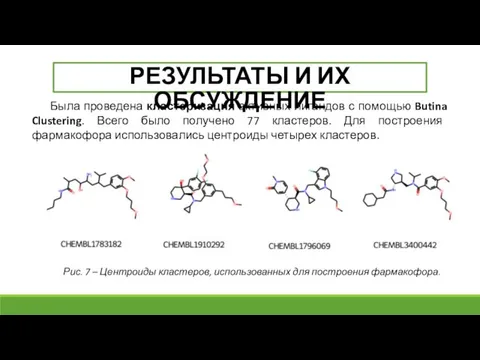
- 15. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ Была проведена кластеризация активных лигандов с помощью Butina Clustering. Всего было получено
- 16. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ Построение фармакофора проводилось с помощью алгоритма MAPex. Полученный фармакофор имеет 4 фармакофорных
- 17. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ По данному фармакофору был проведен поиск в базе данных ZINCPharmer, были сохранены
- 18. РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ 1811 соединений, которые удовлетворяли критериям Липински были проверены по фильтру PAINS (Pan-assay
- 19. Для оставшихся 1245 соединений были получены их фингерпринты. С использованием QSAR модели, которая была построена ранее
- 21. Скачать презентацию















 Патофизиология системы выделения
Патофизиология системы выделения Neurosurgical and Vascular Surgical anatomy
Neurosurgical and Vascular Surgical anatomy Физическое развитие. Характеристика его основных показателей
Физическое развитие. Характеристика его основных показателей факультет педиатрический курос 5 дисциплина психиатрия, медицинская психология тема Шизофрения лекция
факультет педиатрический курос 5 дисциплина психиатрия, медицинская психология тема Шизофрения лекция Тітіркендіргіш заттар. Рефлексті әсерлі қақырық түсіретін дәрілері. ФОҚ-мен улану
Тітіркендіргіш заттар. Рефлексті әсерлі қақырық түсіретін дәрілері. ФОҚ-мен улану Микоплазмоз. Хламидиоз. Уреоплазмоз
Микоплазмоз. Хламидиоз. Уреоплазмоз 16 октября - Международный день здорового питания
16 октября - Международный день здорового питания Лучевые методы исследования гидроторакса, пневмоторакса, пиоторакса
Лучевые методы исследования гидроторакса, пневмоторакса, пиоторакса Твое здоровье в твоих руках
Твое здоровье в твоих руках Новые возможности в лечении вируса гепатита С. Викейра пак
Новые возможности в лечении вируса гепатита С. Викейра пак Кровотечения и оказание первой помощи
Кровотечения и оказание первой помощи История развития психопатологии в России
История развития психопатологии в России Синдром карпального канала
Синдром карпального канала Иерсиниоз, вызванный бактериями вида Yersinia Enterocolitica
Иерсиниоз, вызванный бактериями вида Yersinia Enterocolitica Правовое регулирование психиатрической помощи
Правовое регулирование психиатрической помощи Личностные особенности людей с отклонениями в состоянии здоровья (алкогольная аддикция) в системе комплексной реабилитации
Личностные особенности людей с отклонениями в состоянии здоровья (алкогольная аддикция) в системе комплексной реабилитации Первая помощь при травмах груди, живота, в области таза, при повреждении позвоночника
Первая помощь при травмах груди, живота, в области таза, при повреждении позвоночника СМВО et liberandum (спасение)
СМВО et liberandum (спасение) Гангрена Фурнье
Гангрена Фурнье Osteomyelit. Symptoms
Osteomyelit. Symptoms эндокринология-диф ток зоб
эндокринология-диф ток зоб Первая помощь при повреждениях опорно-двигательной системы
Первая помощь при повреждениях опорно-двигательной системы Влияние стресса на желудочнокишечный тракт у детей
Влияние стресса на желудочнокишечный тракт у детей Медициналық мекемелер 2 ге бөлінеді. Амбулаториялық. Статционарлық
Медициналық мекемелер 2 ге бөлінеді. Амбулаториялық. Статционарлық Полиомиелит
Полиомиелит Значение и гомеостатические константы крови
Значение и гомеостатические константы крови Бауыр тігісі мен өт қапшығындағы және өт түтігіндегі операциялар техникасы
Бауыр тігісі мен өт қапшығындағы және өт түтігіндегі операциялар техникасы Заболеваемость антропонозными бактериальными инфекциями в РФ. Вопросы по ОКИ
Заболеваемость антропонозными бактериальными инфекциями в РФ. Вопросы по ОКИ